Выданный мне контиг NW_001843865 состоит из 505340 пар нуклеотидов (число генов, как и CDS 40) и принадлежит Хламидомонаде Рейнгардта.
Chlamydomonas reinhardtii — это подвижная одноклеточная зелёная водоросль, представитель рода хламидомонада (Chlamydomonas). Эти водоросли широко распространены в почве и пресной воде.
Диаметр клетки около 10 микрометров, плавает при помощи двух одинаковых (равных) жгутиков, расположенных на суженном переднем конце. Возле основания жгутиков имеются две небольшие сократительные вакуоли. Основной компонент клеточной стенки — гликопротеины, богатые гидроксипролином. В клеточной стенке также присутствует растворимая фракция моносахаридов и олигосахаридов. Вопреки данным ранних работ, целлюлоза в ней отсутствует. Хлоропласт крупный, чашеобразный, содержит крупный пиреноид и светочувствительный глазок (стигму). Обычные (немутантные) штаммы Chlamydomonas могут расти на простой культуральной среде, содержащей неорганические соли, на свету используя фотосинтез для обеспечения клетки энергией. Также могут расти в полной темноте, используя в качестве источника углерода ацетат.
С. reinhardtii, так же как и другие представители рода Chlamydomonas, имеет сложный жизненный цикл. Гаплоидные вегетативные клетки размножаются митозом. В условиях недостатка питательных веществ (например, азота) они многократно делятся митозом, образуя половые клетки — гаметы. Затем разнородные гаметы попарно сливаются, образуя диплоидные зиготы. Зигота окружена плотной клеточной стенкой, что позволяет пережить неблагоприятное время. При наступлении благоприятных для жизни условий зитога делится мейозом на 4 гаплоидные вегетативные клетки.
[1]
Таксономия организма: Eukaryota; Viridiplantae; Chlorophyta; Chlorophyceae; Chlamydomonadales; Chlamydomonadaceae; Chlamydomonas [2]
Chlamydomonas reinhardtii[1]
В данном контиге не было генов с альтернативным сплайсингом, поэтому я взяла ген PRPL3. с координатами complement(164347..167937), длинной 3590. Для него известен один вариант mRNA и соответсвующий вариант белка. Это рибосомальный белок хлоропласта L3. У mRNA идентификатор XP_001700690.1 (длина - 259аа), а у белка идентификатор XP_002785455.1 (длина - 130аа)
Изображение гена PRPL3 в геномнои браузере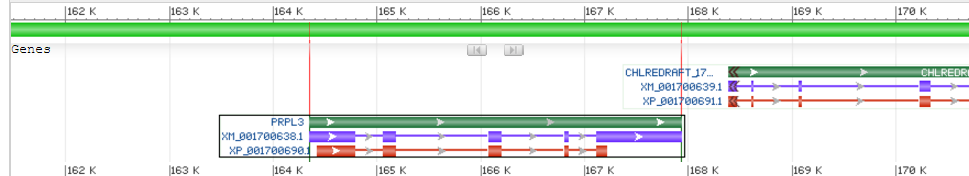
Изображение белка PRPL3
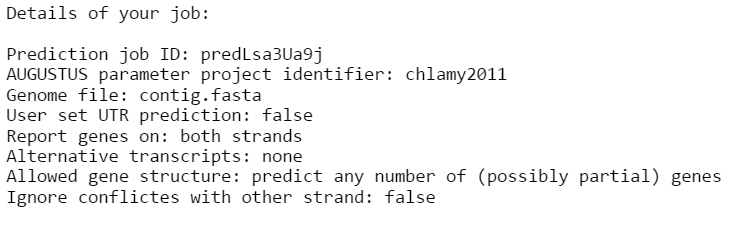
Далее для выданного мне контига были предсказаны гены и белок-кодирующие области с помощью сервера AUGUSTUS в режиме prediction.
Параметры запуска представлены ниже:
После завершения работы сервис выдает архив с результатами, представляющий собой набор файлов вида augustus.xxx (где xxx - aa, cdsexons, codingseq, gbrowse, gff, gtf). Подробнее в таблице ниже.
| augustus.aa | Файл с аминокислотными последовательностями, транслированными из предсказанных генов (формат fasta) |
| augustus.cdsexons | Файл с последовательностями экзонов предсказанных генов (формат fasta) |
| augustus.codingseq | Файл с CDS (формат fasta) |
| augustus.gbrowse | Файл с информацией о координатах, ориентации, структурных особенностях и т.д. pre-mRNA предсказанных генов для геномного браузера |
| augustus.gff | Файл со всей информацией: нуклеотидная, аминокислотная последовательности, координаты, ориентация (формат gff) |
| augustus.gtf | Файл с предсказанными генами в формате .gtf |
Далее, чтобы сравнить результаты предсказания AUGUSTUS с реальными, я получила файлы с координатами и ориентацией генов с помощью скриптов на языке python:
из файла augustus.gbrowse скриптом aug_to_txt.py - augustus.txt;
из файла contig.gff (который я предварительно перевела из файла формата gb, скачанного с NCBI,
командой featcopy пакета EMBOSS) скриптом contig_to_txt.py - contig.txt
Затем скриптом comp.py были сравнены txt-файлы, полученные ранее.
Результат сравнения предсказания AUGUSTUS с реальными генами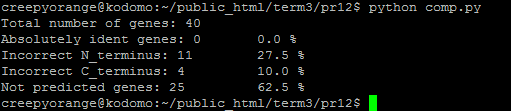
Судя по результатам, AUGUSTUS сработал крайне плохо: не угадан полностью ни один ген, 15 генов из 40 угаданы частично: ошибка на N- или С- конце. Все остальные (62,5%) есть в скэффолде, но предсказаны не были.
[1].
Хламидомонада Рейнгардта on Wikipedia
[2].
NCBI Taxonomy
[2].
AUGUSTUS
© Кучеренко Варвара 2015