Мне был выдан код доступа проекта по секвенированию бактерии Buchnera aphidicola - SRR4240360.
Сначала был скачан архив с заданными ридами, который затем был распакован в папку /nfs/srv/databases/ngs/kucherenko командой gunzip. Затем из файлов в папке с адаптерами был создан один файл, содержащий все адаптеры, с помощью команды:
cat *.fa > /nfs/srv/databases/ngs/kucherenko/adapters.fastaИз полученного файла были удалены все адаптеры командой:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240360.fastq SRR4240359_mod.fastq ILLUMINACLIP:adapters.fasta:2:7:7После этого были удалены нуклеотиды с низким качеством с концов ридов и риды длиной менее 30 нуклеотидов:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240360_mod.fastq final.out TRAILING:20 MINLEN:30В результате был получен файл final.out, с ридами после очистки. Далее для анализа использовалась команда:
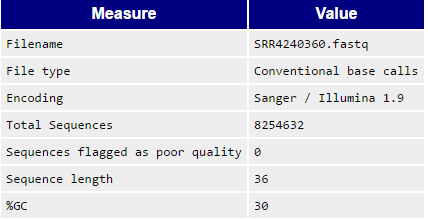
fastqc SRR4240360.fastq
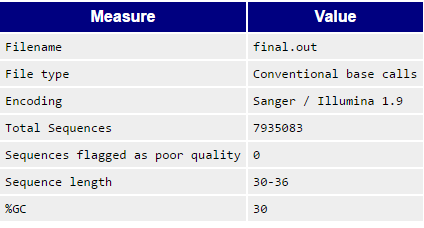
fastqc final.out
Сравнение чтений до (слева) и после(справа) чистки
| Размер файла (MБ) | 832 | 798 |
| Ссылка на страницу с отчетом | SRR4240360_fastqc.html | final.out_fastqc.html |
| Основные характеристики |  |  |
Таким образом после чистки было удалено 319549 ридов (3,87% от общего числа) или 34 МБ - качество отдельных пар нуклеотидов повысилось.
Сначала были подготовлены k-меры длины 29, риды короткие и непарные (short):
velveth velveth 29 -fastq -short final.outТаким образом, в директории velveth находятся все результаты работы программы. Далее были собраны контиги на основе полученных k-меров:
velvetg velvethВ итоге было получено 2 файла: contigs.fa содержит последовательности контигов и stats.txt, содержащий статистику.
Основные подсчеты по контигам проведены с помощью excel (ссылка на файл)
| ID контига | Длина | Покрытие | Файл с последовательностью | 1 | 94956 | 43,68 | con1.fasta |
| 5 | 70305 | 49,35 | con5.fasta |
| 9 | 70300 | 42,00 | con9.fasta |
В нашем графе 1509 вершина, N50=67050, max 94956, total 707455, using 0/7935083 reads.
| ID контига | Длина | Покрытие |
| 632 | 59 | 1,58 |
| 1882 | 37 | 116,89 |
Случаи аномально большого покрытия, вероятно, объясняются сранительно малой длиной контигов.
С помощью алгоритма megablast я сравнила каждый из трёх самых длинных контигов с хромосомой Buchnera aphidicola (CP009253). Контиги с аномально большим и аномально маленьким покрытием не выраниваются в blast из-за маленького размера. Query - исследуемый контиг, Subject - геном бактерии длиной 628164 пн.
Сравнение самых длиных контигов с хромосомой Buchnera aphidicola
| ID контига | Координаты участка хромосомы (для одного мэтча) | Max score | Total score | Query cover | E value | Identities | Matches | Gaps |
| 1 | 467412 to 474667 | 4047 | 32384 | 60% | 0.0 | 77% | 20 | 208/7389(2%) |
| 5 | 35124 to 44693 | 8517 | 34072 | 71% | 0.0 | 83% | 13 | 130/9633(1%) |
| 9 | 389348 to 398726 | 4748 | 30562 | 79% | 0.0 | 75% | 14 | 390/10884(3%) |
© Кучеренко Варвара 2015