Капиллярный секвенатор по Сангеру выдает файлы с хроматограммой и автоматически прочтенной последовательностью в формате .ab1. В качестве исходных для задания было выдано два файла: Прямая цепь и Обратная цепь.
С помощью программы Chromas были обработаны исходные файлы: обратная цепочка для удобства была заменена на комплементарную (Edit > Reverse+Complement), удалены нечитаемые участки в начале и конце последовательностей, путем сравнения двух цепей и анализа хроматограмм исправлены ошибки.
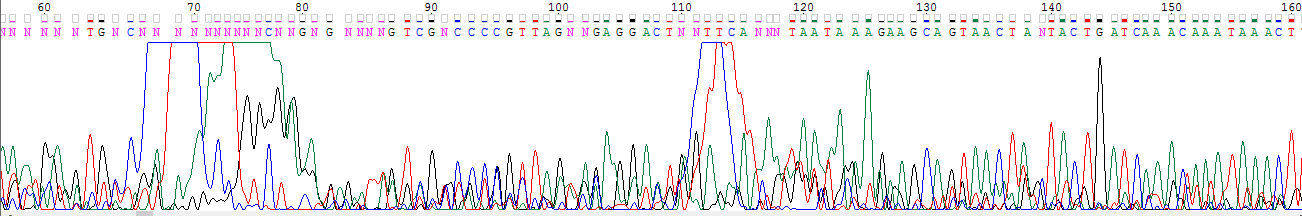
Границы нечитаемых участков |
||
Прямая цепь |
Обратная цепь |
|
Начальный участок (номера нуклеотидов) |
1-27 |
1-14 (для комплементарной цепи) |
Конечный участок (номера нуклеотидов) |
687-717 |
650-690 (для комплементарной цепи) |
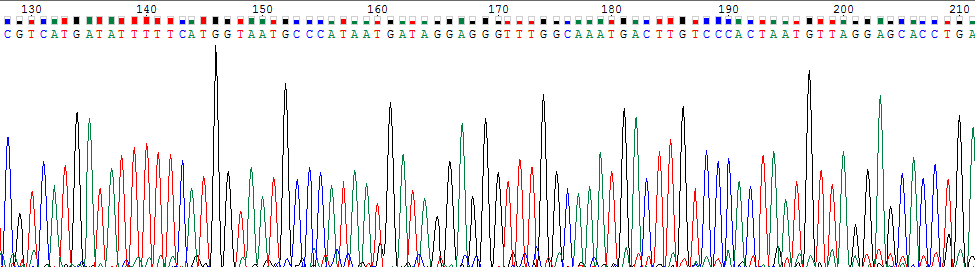
Обе исходные хроматограммы хорошего качества: короткие нечитаемые участки наблюдаются только в начале и в конце последовательностей, в остальной части можно легко различить пики (шум не перекрывает сигналы). Ближе к 3'-концу прямой и к 5'-концу обратной последовательности качество хроматограмм падает: появляются раздвоенные пики и усиливается шум.Примерное соотношение уровня сигналов к уровню шума для прямой цепи - 1/10, для обратной - 1/7.
Четкие пики сигналов из хроматограммы прямой цепи
После редактирования в программе Chromas были получены новые последовательности прямой и обратной цепей, которые были выровнены в программе Jalview (ссылка на проект). Проблемные нуклеотиды обозначены строчными буквами; предполагаемые полиморфизмы обозначены по номенклатуре IUPAC.
Изображение полученного выравнивания с раскраской по нуклеотидам. Проблемные нуклеотиды выделены строчными буквами. Явный полиморфизм найден один и обозначен строчной буквой m.

Обоснование решений для проблемных нуклеотидов |
||
1 |
|
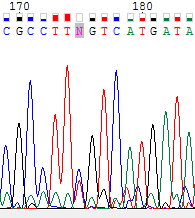
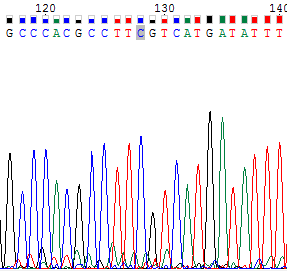
Нуклеотид, обозначенный N находится в обратной цепи (верхнее изображение). Он не был определен по причине того, что на его позиции обнаруживаются сразу два почти одинаковых довольно слабых сигнала от тимина и цитозина (настолько слабых, что сравнимы с шумом). Однако в прямой цепи в соответствующей позиции различим довольно четкий сигнал от цитозина (нижнее изображение), поэтому было решено заменить N на C. |
|
||
2 |
|
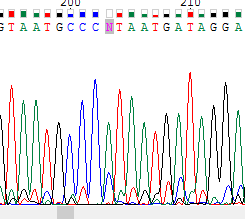
Нуклеотид, обозначенный N находится в также обратной цепи (верхнее изображение). Он не был определен по причине того, что на его позиции есть два сигнала от аденина и цитозина . Сигнал от цитозина слабее, но он сравним во высоте с пиком аденина. Однако в прямой цепи в соответствующей позиции четко виден сигнал от цитозина при небольшом шуме, поэтому было решено заменить N на А. |
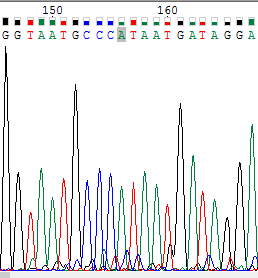
|
||
3 |
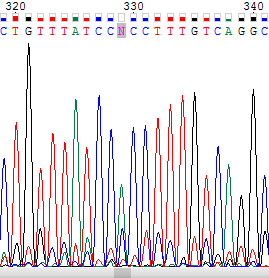
|
Нуклеотид, обозначенный N находится в обеих цепях. (верхнее изображение – прямая цепь, нижнее - обратная). Он не был определен по причине того, что на его позиции есть два сигнала от аденина и цитозина. Пик цитозина по высоте схож с шумовыми сигналами, но в обеиз цепях сила сигналов от аденина и цитозина не сильно различаются. Поэтому было решено считать данный нуклеотид полиморфизмом заменить N на M. |
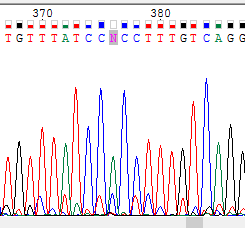
|
||
4 |
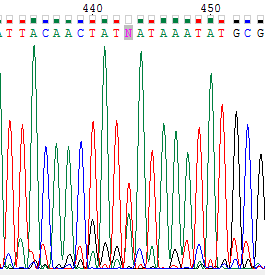
|
Нуклеотид, обозначенный N находится в также прямой цепи (верхнее изображение). Он не был определен по причине того, что на его позиции есть два схожих по силе сигнала от аденина и тимина . В обратной цепи в соответствующей позиции четко виден сигнал от тимина, поэтому было решено заменить N на T. |
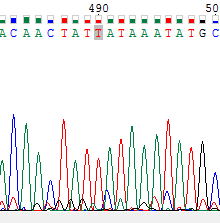
|
||
Изображение фрагмента хроматограммы из файла.
Большой уровень шума говорит о сильной загрязненности образца,
возможно с несколькими разными ДНК. Также явно видны пятна краски (высокие размытые пики) слева и в центре изображения.
© Кучеренко Варвара 2015