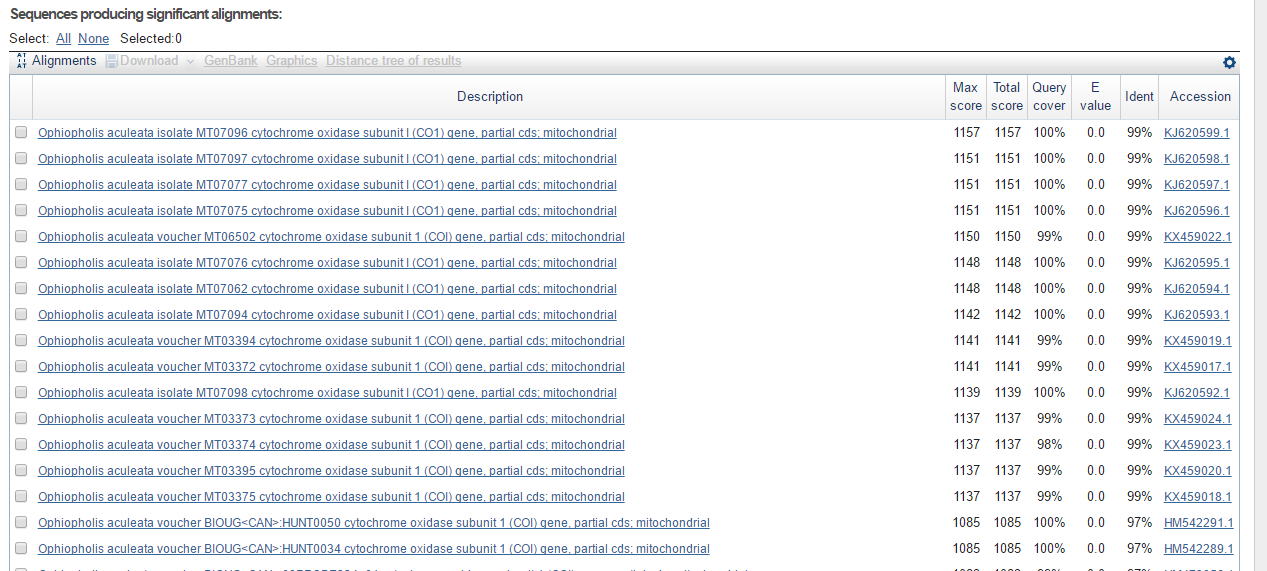
Результат работы алгоритма blastn
Для выполнения задания была взята последовательность, полученная при выполнении практикума 6. Затем с помощью нуклеотидного BLASTа с алгоритмом blastn (Somewhat similar sequences) по базе данных Nucleotide collection (nr/nt) при дефолтных параметрах.

Результат работы алгоритма blastn
Из этого можно сделать вывод, что скорее всего, данная последовательность является митохондриальный ген, кодирующий субъединицу 1 цитохром-С оксидазы дыхательного комплекса IV. Таксономию изучаемой последовательности можно восстановить по нескольким найденным самым схожим. С помощью программы Jalview было построено выравнивание с первыми 10 находками.

В данном случае будут рассматриваться три алгорится: blastn, megablast и discontiguous megablast. Чтобы сравнить их, нужно расширить область поиска. Результаты запроса по роду Ophiactidae (taxid:41169): 18 последовательности для megablast, 28 для discontiguous megablast, 28 для blastn.
Параметры ограничения таксонов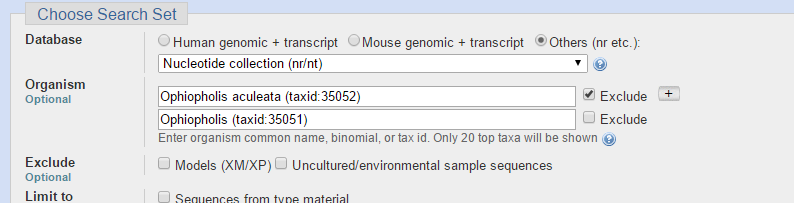
| Параметры запуска BLAST | |||||||
|---|---|---|---|---|---|---|---|
| Algorithm | Database | Max Target Sequences | Expect Threshold | Word Size | Max matches | Match/Mismatch Scores | Gap Costs |
| megablast | Nucleotide collection (nr/nt) | 1000 | 0.001 | 28 | 0 | 1, -2 | Linear |
| discontiguous megablast | Nucleotide collection (nr/nt) | 1000 | 0.001 | 11 | 0 | 2, -3 | Existence:5, Extention: 2 |
| blastn | Nucleotide collection (nr/nt) | 1000 | 0.001 | 11 | 0 | 2, -3 | Existence:5, Extention: 2 |
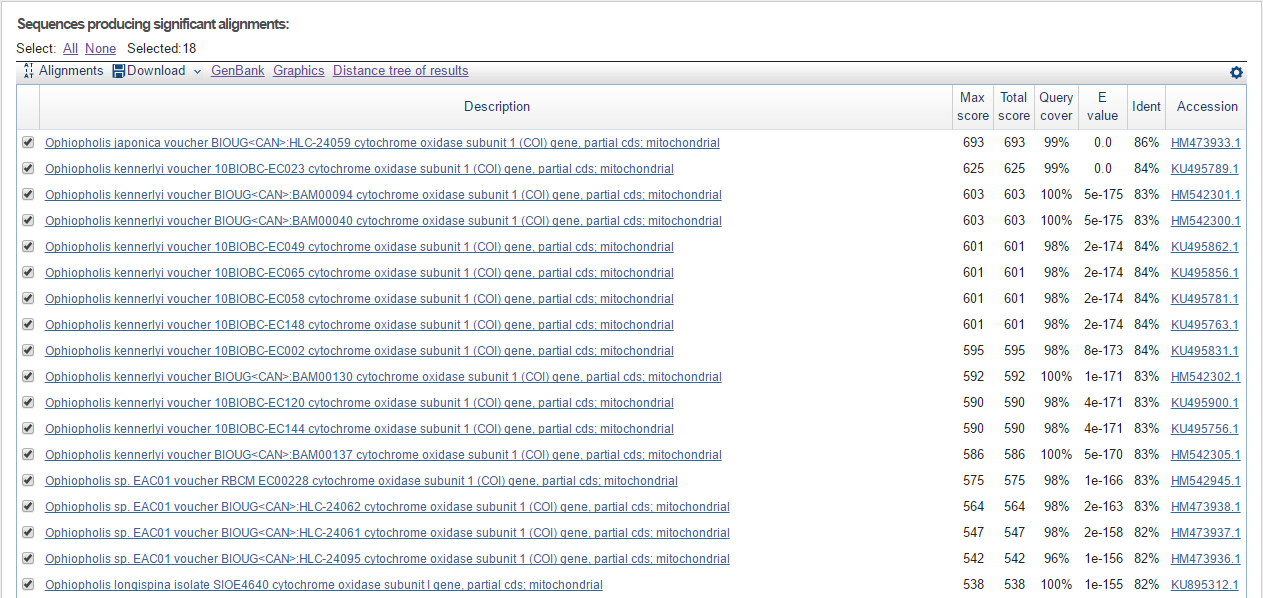
Результаты работы алгоритма megablast
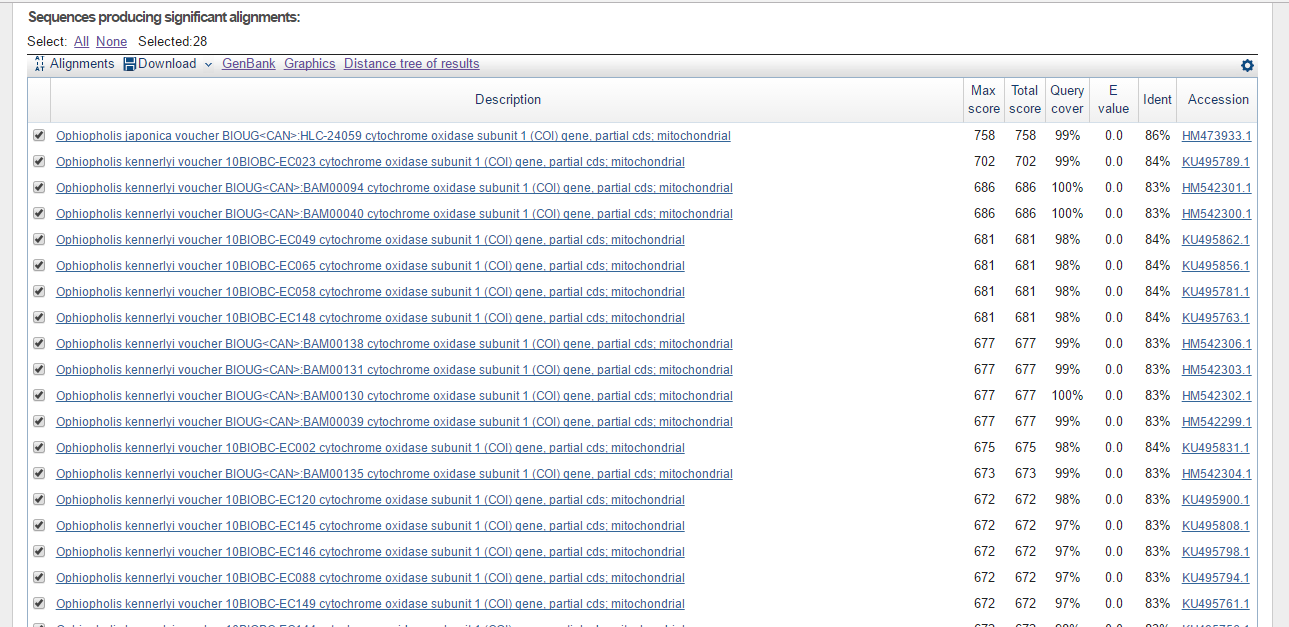
Результаты работы алгоритма discontiguous megablast
Результаты работы алгоритма discontiguous blastn
| Сравнение алгоритмов | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Алгоритм | Число находок | Score лучшей находки | Score худшей находки | E-value лучшей находки | E-value худшей находки | Ident лучшей находки | Ident худшей находки | Query cover лучшей находки | Query cover худшей находки |
| megablast | 18 | 693 | 538 | 0.0 | 1e-155 | 86% | 82% | 99% | 100% |
| discontiguous megablast | 28 | 758 | 429 | 0.0 | 9e-132 | 86% | 81% | 99% | 74% |
| blastn | 28 | 758 | 459 | 0.0 | 9e-132 | 86% | 81% | 99% | 74% |
Больше всего находок выдали алгоритмы discontiguous megablast и blastn (нашли одинаковые последовательности).
При расширении зоны поиска до семейства ситуация аналогичная: найдены 237 одинаковых пунктов
у обоих алгоритмов, при этом разница с числом находок megablast (34 штуки)
сильно увеличивается. При дальнейшем укрупнении запрашиваемого таксона,
появляется небольшая разница между blastn и discontiguous megablast (blastn находит немного больше).
Пример находки, найденной ими и не найденной megablast: Ophiopholis sp. EAC01 voucher BIOUG<CAN>:HLC-24064 cytochrome oxidase subunit 1 (COI) gene, partial cds; mitochondrial
Все находки megablast пересекаются со списком discontiguous megablast и blastn, но их значительно меньше.
Из полученных данных можно сделать вывод, что blastn и discontiguous megablast
работают одинаково, с единственным отличием, что blastn выдает немного больше последовательностей.
Blastn (или discontiguous megablast) и megablast находят одни и те же находки,
но отличные по параметрам max score, total score и query cover.
Megablast работает гораздо строже, отсеивая большее количество находок и, следовательно,
выдавая последовательности, лишь наиболее близкие к исходной.
Он подходит для поиска близкородственных последовательностей, работает достаточно быстро.
Необходимо проверить наличие гомологов определенных белков в геноме организма X5 (Amoboaphelidium protococarum) c помощью локального BLAST.
Задание выполнялось с помощью версии BLAST+, установленной на kodomo.
Для начала я создала локальную базу данных (makeblastdb -in X5.fasta -dbtype nucl).Параметр "-dbtype"
указывает на тип последовательности (в случае нуклеотидной последовательности нужно указать "-dbtype nucl".
Затем для каждого из выбранных белков запустила по ней алгоритм tblastn,
находящий гомологи белка в формальной трансляции нуклеотидного банка
(tblastn -query xxx.fasta -db X5.fasta > xxx.out)[2]
Белок HSP7C - белок теплового шока. Выступает репрессором активации транскрипции. Ингибирует транскрипционную активность CITED1 Smad-опосредованной транскрипции. Консервативный шаперон HSP70. Компонент комплекса PRP19-CDC5L, формирующий концевую часть сплайсосомы, необходим для активации сплайсинга pre-mRNA. Контактирует со всеми ее компонентами, поэтому может выполнять структурную функцию. Связывается с бактериальными LPS, выступает посредником в LPS-индуцированной воспалительной реакции, включая секрецию моноцитами фактора некроза опухолей TNF.[2]
Результат работы программы tblastn в файле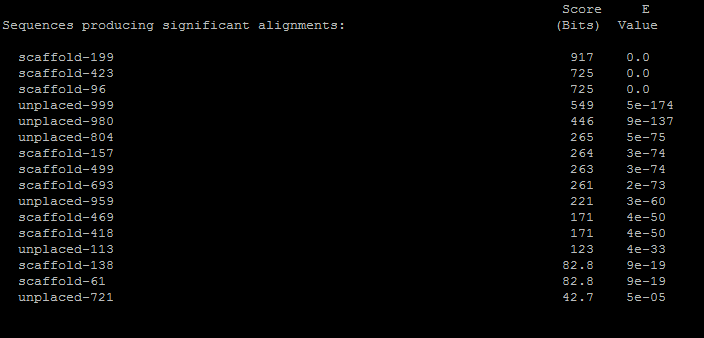
Лучший результат:
> scaffold-199
Length=1112851
Score = 917 bits (2369), Expect = 0.0, Identities = 474/607 (78%), Positives = 538/607 (89%), Gaps = 0/607 (0%), Frame = -2
Лучшая находка имеет хороший E-value и остальные параметры, таким образом, что эту находку можно назвать гомологом, и скорее всего она несет схожие функции.
TERT_HUMAN - теломераза, восстанавливающая длину хромосомы при репликации. Имеется у большинства, но не у всех эукариот. Активна в прогениторных и раковых клетках, в нормальных же соматических не активна или проявляет очень низкую активность. Играет важную роль в процессах старения и предотвращении апоптоза.[3]
Результат работы программы tblastn в файле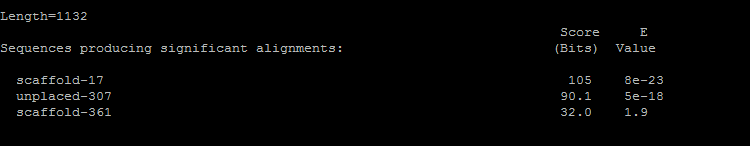
Лучший результат:
> scaffold-17
Length=2125590
Score = 105 bits (263), Expect = 8e-23, Method: Compositional matrix adjust.
Identities = 151/568 (27%), Positives = 248/568 (44%), Gaps = 43/568 (8%)
Frame = +1
Находку можно охарактеризовать как условно положительную: совпадения в последовательностях есть, хоть и параметры сходства довольно низкие (не можем говорить о похожих функциях), и они так разбросаны, что о гомологии доменов говорить не приходится.
CISY_HUMAN - митохондриальная цитратсинтаза. Участвует в цикле трикарбоновых кислот. Проявляет каталитическую активность в реакции Acetyl-CoA + H2O + oxaloacetate = citrate + CoA. [4]
Результат работы программы tblastn в файле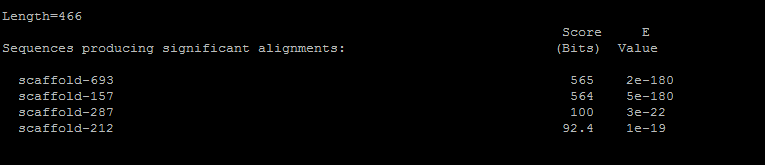
Лучший результат:
> scaffold-693
Length=1268102
Score = 565 bits (1457), Expect = 2e-180, Method: Compositional matrix adjust.
Identities = 262/377 (69%), Positives = 315/377 (84%), Gaps = 3/377 (1%)
Frame = +1
Параметры сходства снова достаточно хорошие, так что с уверенностью можно утверждать, что данный белок является гомологом.
С помощью команды infoseq пакета EMBOSS была получена информация о длинах контигов (infoseq X5.fasta -only -name -length.) Я выбрала последовательность scaffold-693 (длина 1268102 нуклеотидов), т.к. она достаточно большая для наличия в ней кодирующего белок гена, и поместила ее в отдельный файл (командой seqret X5.fasta:scaffold-693 -out scaf693.fasta).
Далее был запущен blastn с параметрами по умолчанию и ограничению по таксону Amoeboaphelidium (taxid:1243176).
Результат работы blastn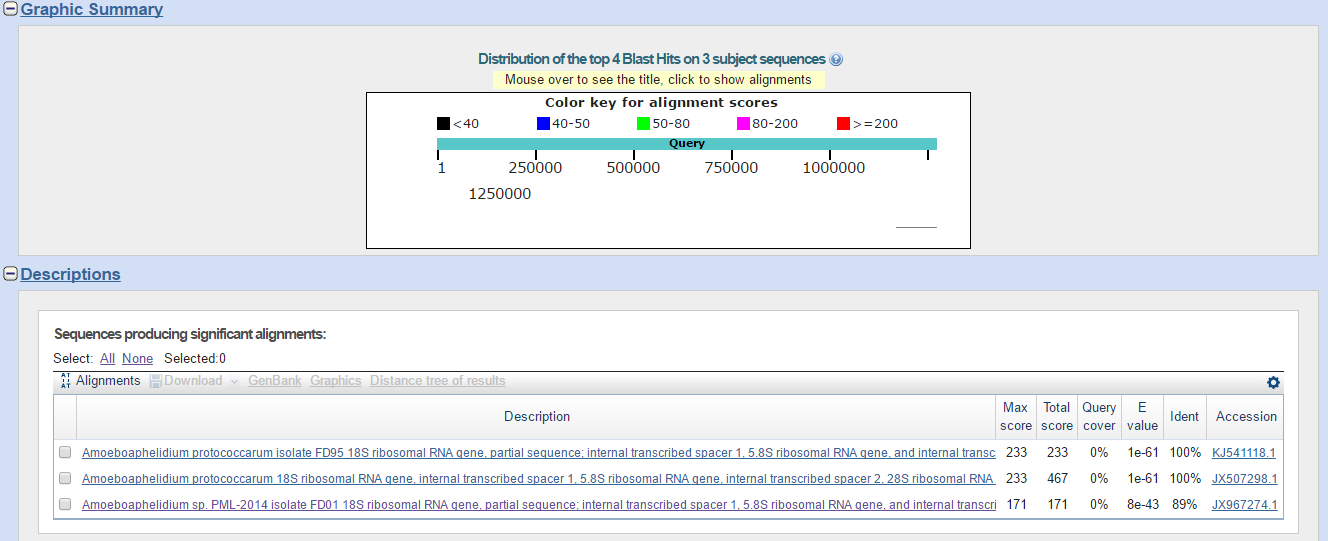
Как видно из результатов, у нас есть 3 последовательности генов со 100% Identity и хорошим E-value. Такой низкий
Query cover обусловлен малыми размерами гена по сравнению с длиной общей последовательности.
Следовательно, с уверенностью можно утверждать, что данный контиг содержит найденные гены.
Лучший в списке находок -
частичная последовательность рибосомального гена 18S изолята FD95;
полная последовательность внутреннего транскрибируемого сплайсера 1 5.8S рибосомального РНК гена и внутренноего транскрибируемого сплайсера 2;
частичная последовательность 28S рибосомального РНК гена.
[1].
Heat shock cognate 71 kDa protein (UniProt)
[2].
Инструкция к BLAST на kodomo
[3].
Telomerase reverse transcriptase (UniProt)
[4].
Citrate synthase, mitochondrial (UniProt)
© Кучеренко Варвара 2015