
Для построения филогенетического дерева был выбран белок из семейстся энолаз (мнемоника ENO). Соответсвующие последовательности белков у выбранных бактерий (см. прошлый практикум) были загружены с Uniprot и выравнены в Jalview (ссылка на проект и выравнивание в fasta).
Выравнивание белковых последовательностей бактерий

Затем методом Neighbor Joining Using % Identity в Jalview было построено дерево и открыто в программе MEGA:
| Изображение дерева с учетом длин ветвей | Изображение дерева без учета длин ветвей |
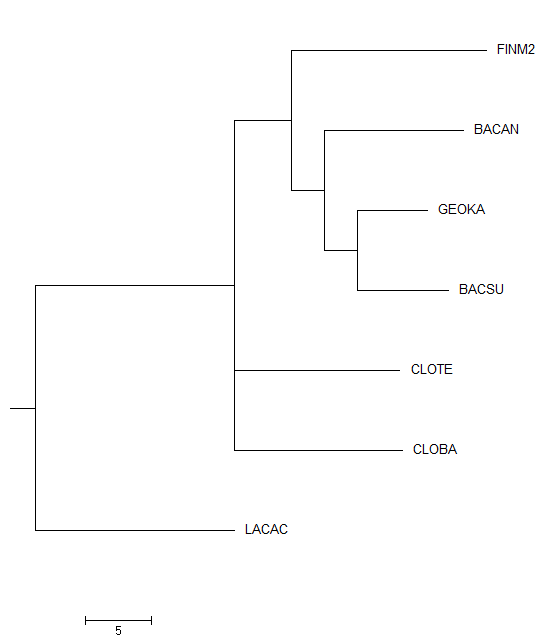 | 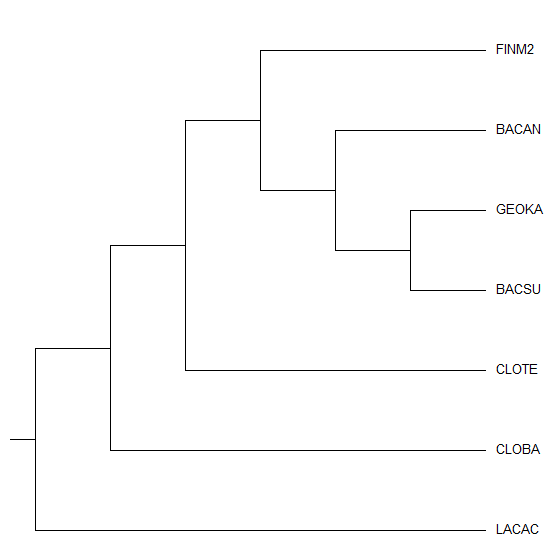 |
Как можно видеть, в случае с учетом длин дерево получается небинарным. Даже с учетом всех его решений ни одна из топологий не совпадает с таксономическим древом бактерий. Укоренение произошло в ветвь {LACAC} vs. {FINM2, BACAN, GEOKA, BASCU, CLOTE, CLOBA}. Если же длины ветвей не учтывать, то дерево получается бинарным (топология которого даже с учетом всех решений снова не совпадает с топологией таксономического дерева).
Из полученного филогенетического дерева бактерий с помощью программы retree пакета PHYLIP (команда М) было получено новое дерево с укоренением в среднюю точку:
| Изображение дерева с учетом длин ветвей | Изображение дерева без учета длин ветвей |
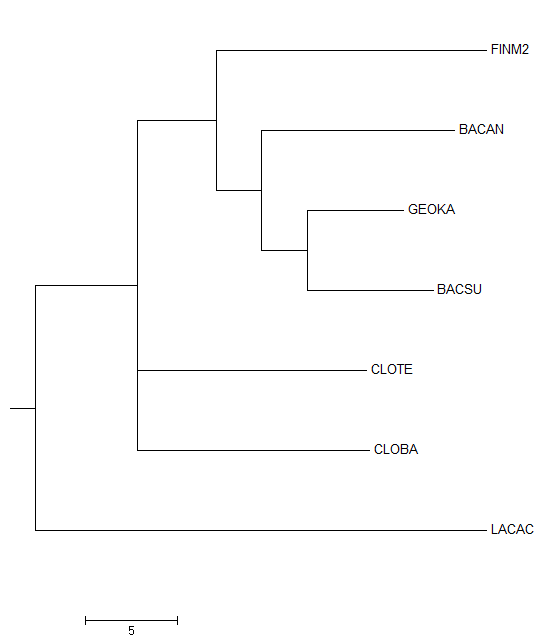 | 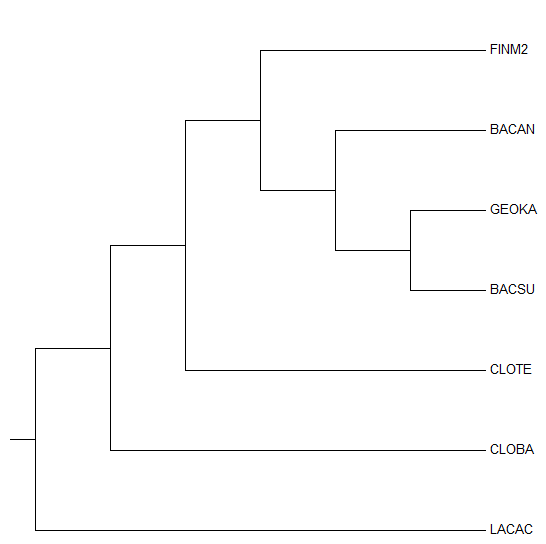 |
Укоренение в среднюю точку не изменило положение корня. Топология полностью совпадает с топологией деревьев, построенных методом NJ.
В укоренении методом максимальной экономии (Maximum Parsimony, MP) не используются длины ветвей, поэтому укоренение в среднюю точку невозможно. В качестве внешней группы был выбран белок из семейства энолаз e.coli (мнемоника ENO_ECOLI). Последовательность белка была добавлена к невыровненным остальным последовательностям и далее был использован алгоритм Muscle. С помощью программы MEGA по выравниванию, построенному в Jalview (ссылка на выравнивание в fasta) методом MP было построено дерево. Командой "Show Subtree Separately" было получено это же дерево без e.coli.
Изображение дерева, полученного методом Maximum Parsimony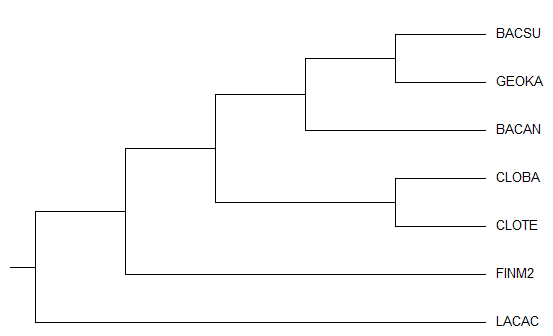
Если сравнивать с орининальным таксономическим деревом, то сходства снова будет немного: из общих нетривиальных ветвей только {BACAN, BASCU, GEOKA, LACAC, FINM2} vs {CLOTE, CLOBA} и {BACAN, BASCU, GEOKA} vs {LACAC, FINM2, CLOTE, CLOBA}. По-видимому, судить о филогении организмов по древу данного белка нельзя.
Статистический бутстрэп (бутстреп, бутстрэппинг, англ. bootstrap, bootstrapping) — практический компьютерный метод исследования распределения статистик вероятностных распределений, основанный на многократной генерации выборок методом Монте-Карло на базе имеющейся выборки. Суть метода состоит в том, чтобы по имеющейся выборке построить эмпирическое распределение. Используя это распределение как теоретическое распределение вероятностей можно с помощью датчика псевдослучайных чисел сгенерировать практически неограниченное количество псевдовыборок произвольного размера, например, того же как у исходной. На множестве псевдовыборок можно оценить не только анализируемые статистические характеристики, но и изучить их вероятностные распределения.[1]
В программе MEGA был проведен бутстреп-анализ дерева. Создается определенное количество "бутстреп-реплик", в каждой из которых половина случайно выбранных нуклеотидных столбцов заменяется на копии случайно выбранных из другой половины столбцов. Для каждой реплики строится дерево. Затем их объедитняют в одно итоговое методом расширенного большинства («Extended majority-rule tree»). Приоритетны в построении ветви, встречающиеся в большинстве деревьев, далее добавляются наиболее "поддержанные", не противоречащие уже имеющимся ветвям. Числа обозначают количество деревьев, построенных по бутстреп-репликам, с данной веткой. Количество реплик: 100.
| Оригинальное дерево, построенное методом NJ | Консенсусное бутстреп-дерево |
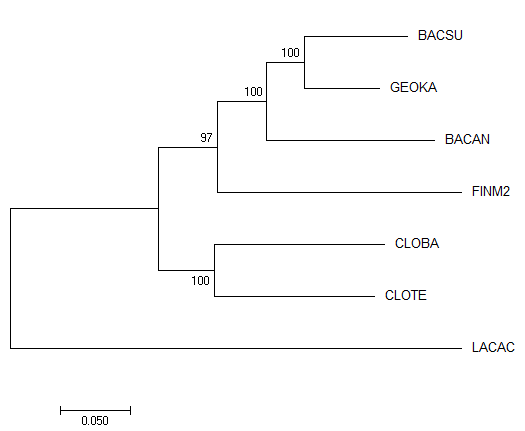 | 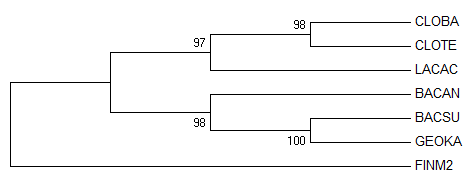 |
| Ветви: {BACAN, BASCU, GEOKA, LACAC, FINM2} vs {CLOTE, CLOBA} {BASCU, GEOKA} vs {BACAN, LACAC, FINM2, CLOTE, CLOBA} {BASCU, GEOKA, BACAN, FINM2} vs {LACAC, CLOTE, CLOBA} {BASCU, GEOKA, BACAN} vs {LACAC, FINM2, CLOTE, CLOBA} | Ветви: {BACAN, BASCU, GEOKA, LACAC, FINM2} vs {CLOTE, CLOBA} {BACAN, BASCU, GEOKA, FINM2} vs {CLOTE, CLOBA, LACAC} {BASCU, GEOKA} vs {BACAN, LACAC, FINM2, CLOTE, CLOBA} {BASCU, GEOKA, BACAN} vs {LACAC, FINM2, CLOTE, CLOBA} |
Топологии NJ и консенсусного деревьев не отличаются. Однако они совпадают с эталонным деревом только по 2 ветвям из 4, при этом все ветви достаточно хорошо "поддержаны" статистически.
Источники:
[1] Статистический бутстреп
© Кучеренко Варвара 2015