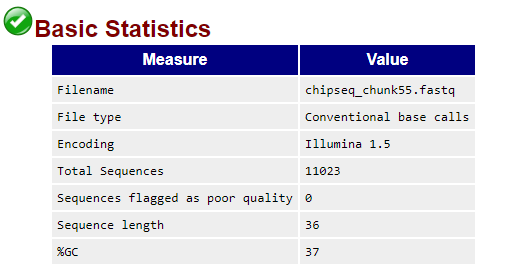
Для работы был выдан файл chipseq_chunk55.fastq с ридами Illumina, полученными
в результате ChIP-seq эксперимента.
С помощью программы FastQC был проведен контроль качества прочтений.
Команда: fastqc chipseq_chunk55.fastq.
 |
 |
| Информация о ридах в файле до чистки | Качество рида по основаниям |
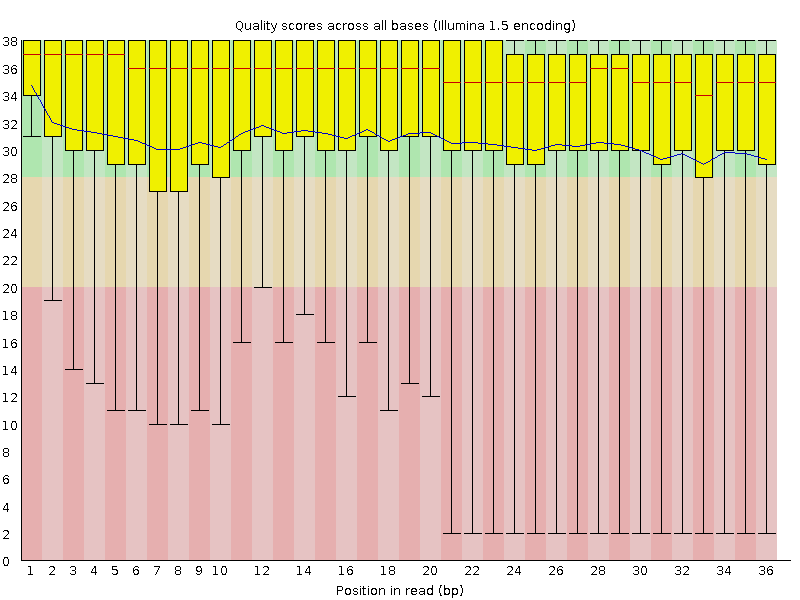
На диаграмме видно, что данные нуждаются в "чистке" (концы "усов" практически все в области низкого качества).
Поэтому для этих целей было решено воспользоваться программой Trimmomatic.
Команда: java -jar /usr/share/java/trimmomatic.jar SE -phred33 chipseq_chunk55.fastq chunk55_new.fastq
TRAILING:50 MINLEN:36.
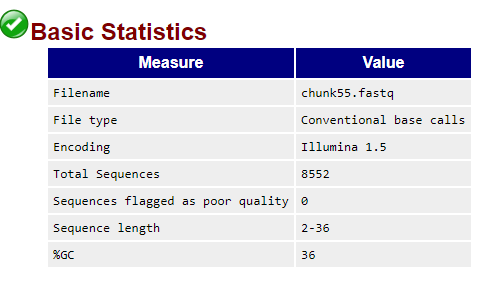
Результат очистки — файл chunk55_new.fastqc, содержал 8552 последовательностей (в
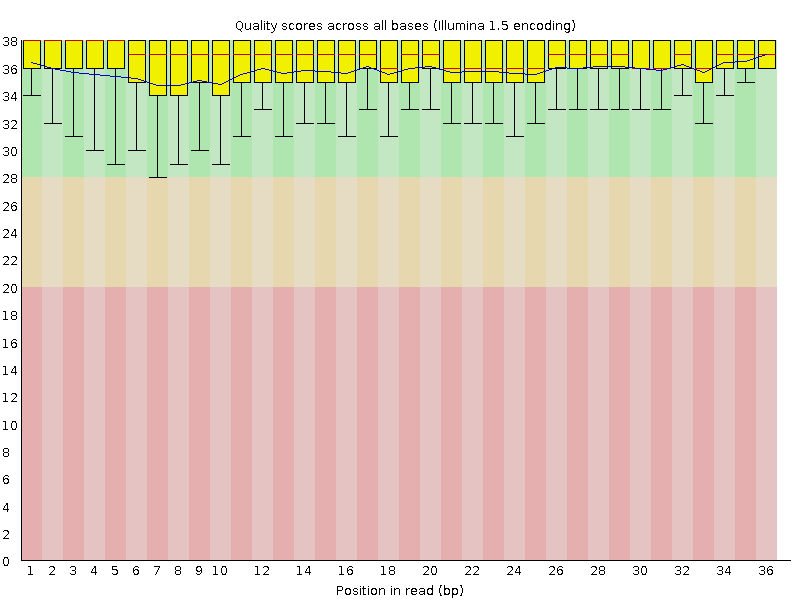
ходе работы Trimmomatic отсек порядка 22.4%). На диаграмме видно, что все "усы" теперь заканчиваются
в "благоприятной" области.
 |
 |
| Информация о ридах в файле после чистки | Качество рида по основаниям |
Далее последовательности были откартированы на проиндескированный геном человека.
Команда: bwa mem /srv/databases/ngs/hg19/GRCh37.p13.genome.fa chunk55_new.fastq > chunk55.sam .
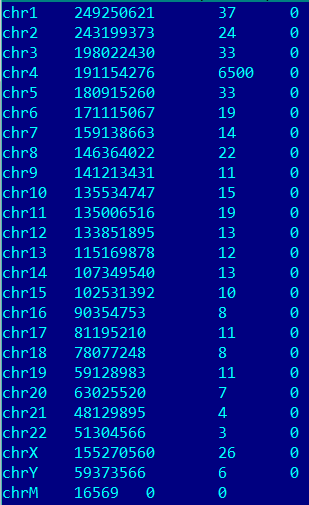
Картированные последовательности
Больше всего ридов откартировалось на chr4. Таким образом, мы можем устверждать, что данные чтения именно с этой хромосомы.
Далее для поиска пиков я воспользовалась программой MACS.
Команда: bwa mem macs2 callpeak -t chunk55.sorted.bam .
Результат: файлы chunk55_peaks.xls, chunk55_summits.bed, chunk55_peaks.narrowPeak.
| chr | start | end | length | abs_summit | pileup | -LOG10(pvalue) | fold_enrichment | -LOG10(qvalue) | name |
| chr4 | 124401051 | 124401278 | 228 | 124401163 | 22.00 | 15.70197 | 7.32484 | 9.08797 | chunk55_peak_1 |
| chr4 | 124426828 | 124427099 | 272 | 124426971 | 36.00 | 29.83622 | 10.75581 | 20.37157 | chunk55_peak_2 |
| chr4 | 124446470 | 124446765 | 296 | 124446629 | 21.00 | 16.18124 | 7.85714 | 9.53217 | chunk55_peak_3 |
| chr4 | 124476588 | 124476871 | 284 | 124476774 | 29.00 | 27.93735 | 12.09677 | 19.49099 | chunk55_peak_4 |
| chr4 | 124521277 | 124521493 | 217 | 124521332 | 30.00 | 23.66802 | 9.39394 | 16.45623 | chunk55_peak_5 |
| chr4 | 124570929 | 124571230 | 302 | 124571121 | 23.00 | 18.63315 | 8.69565 | 11.89196 | chunk55_peak_6 |
| chr4 | 124898897 | 124899263 | 367 | 124899069 | 31.00 | 26.37031 | 10.52632 | 18.87879 | chunk55_peak_7 |
Визуализация пиков проводилась в геномном браузере UCSC Genome Browser. Использовался файл chunk55_peaks.narrowPeak, в начало которого были предварительно напечатаны строчки: track type=narrowPeak visibility=3 db=hg19 name="my_peaks" description="chunk55_peaks" ; browser position chr4:124401000-124900000
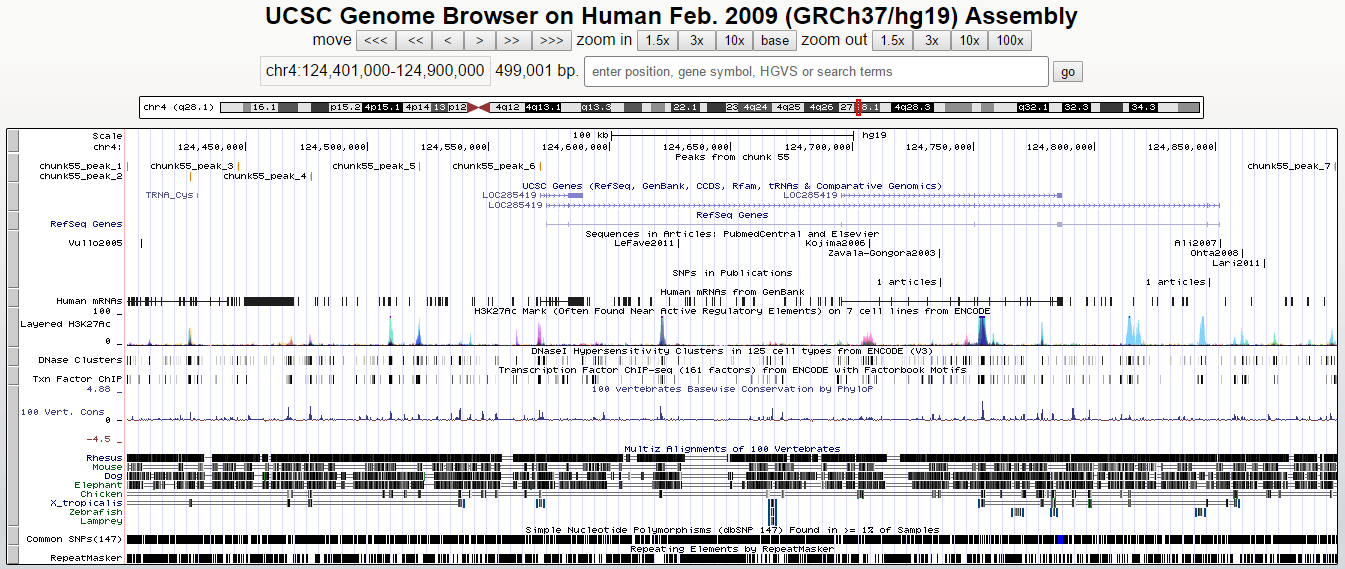
Пики в геномном браузере
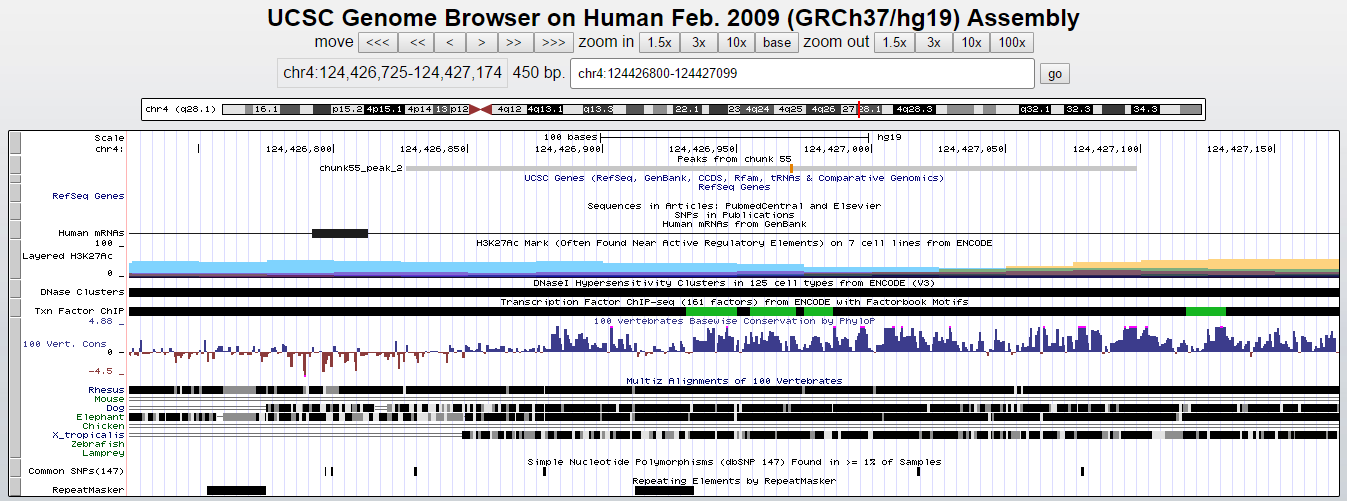
Наиболее достоверный пик (peak2)
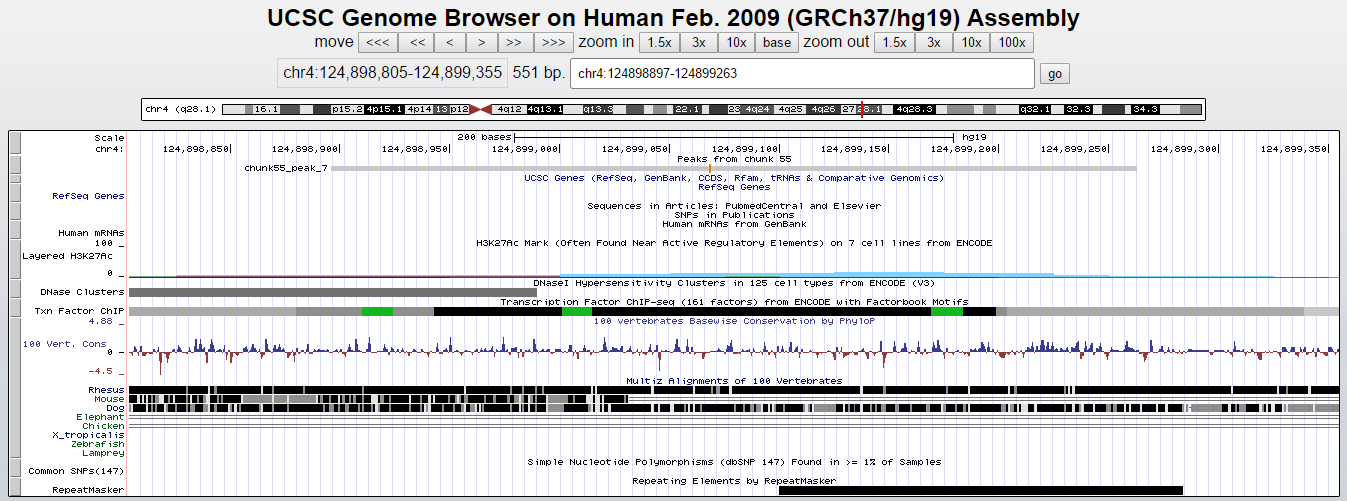
Наименее достоверный пик (peak7)
Расстояние от начала до вершины пика 2 = 143, пика 7 = 172. Как мы можем видеть, ни один из этих пиков не пересекается с областями каких-либо генов.
© Кучеренко Варвара 2015