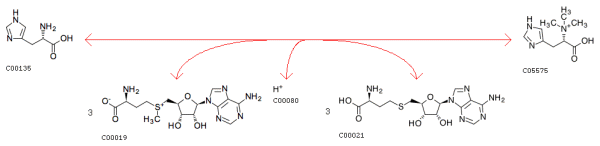
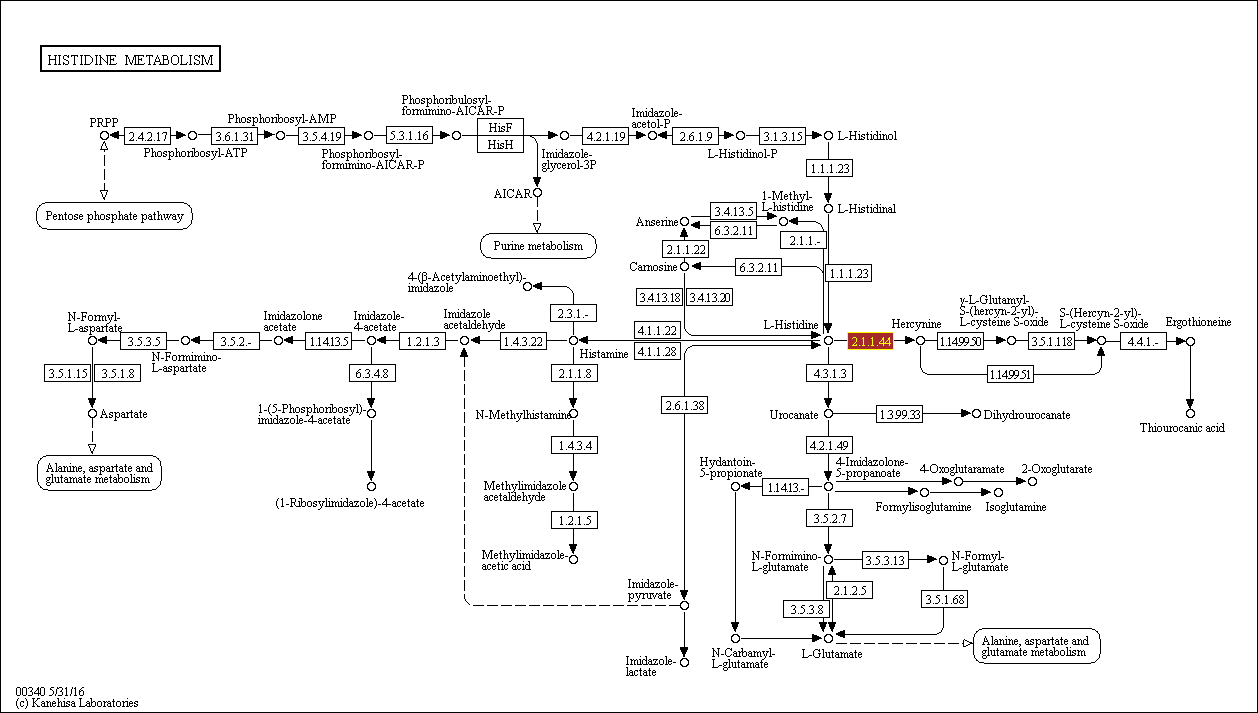
В базе данных KEGG был выбран
метаболический путь гистидина (Histidine metabolism),
в котором была взята реакция EC:2.1.1.44.
Реакция катализируется двумя рядами ортологических белков: K20246
(L-histidine Nalpha-methyltransferase / hercynylcysteine S-oxide synthase),
K18911(L-histidine Nalpha-methyltransferase).
K20246 содержит 108 белков.
K18911 содержит 173 белка.
Рис.1 Выбранная реакция: L-Histidine + 3 S-Adenosyl-L-methionine + H+ <=> Hercynine + 3 S-Adenosyl-L-homocysteine
Рис.2 Путь метаболизма гистидина (выбранная реакция выделена оранжевым цветом)
По спискам идентификаторов UniProt были получены аминокислотные последовательности белков ортологических рядов K20246 и K18911. С помощью скрипта на Python идентификаторы белков в файле были изменены до требуемого вида. Далее с помощью Muscle последовательности обоих рядов были выровнены как вместе, так и по отдельности (ссылка на выравнивание K20246 и K18911 в формате fasta). Из выравнивания видно, что ортологический ряд K18911 состоит из гомологов. Ряд K20246 состоит из последовательностей, сильно разнящихся по длине, причем четкого большинства белков определенной длины нет (при выравнивании именно ряда отдельно обнаруживается множество гэпов). Возможно, гомологию в данном ряду можно рассматривать только на уровне доменов(?). При общем выравнивании двух рядов вместе также гомология не прослеживается. Следовательно, строить филогенетическое дерево не имеет смысла.
© Кучеренко Варвара 2015