BLAST
При выполнении задания была использована последовательность белка L-rhamnose isomerase бактерии Bacteroides thetaiotaomicron (идентификатор Q8A1A2). Поиск проводился по базе Refseq_protein.
Задание 1. После запуска BLAST с заданными параметрами (алгоритм - blastp; база данных - Refseq_protein; максимальное число находок - 5000; Gap Existence - 10; Gap Extension - 2), число находок составило 1707 последовательностей. Все белки принадлежат организмам домена Bacteria, среди которых есть представители родов Salmonella, Escherichia, Bacteroides, Klebsiella и некоторые другие.
В таблице 1 представлены параметры для трех находок - "лучшей", "худшей" и находки из середины списка. В качестве "лучшей" взята последовательность из генома близкородственной бактерии Bacteroides finegoldii, так как первые 3 находки соответствует исходному белку; в качестве "худшей" взята находка из конца списка.
| Организм | Длина выравнивания | Bit Score | E-value | % идентичных остатков | % сходных остатков |
| Bacteroides finegoldii | 418 | 945 | 0.0 | 98% | 99% |
| Lactobacillus equi | 420 | 441 | 1e-144 | 48% | 65% |
| Mesorhizobium sp. LNJC384A00 | 274 | 38.6 | 9.9 | 28% | 42% |
Выравнивания, которые построил BLAST:
По условию, гомологами исходной последовательности можно считать те, у которых E-value < 1e-3 и Query cover не менее 70%. Для поиска гомологов я задал максимальное значение E-value 0.001 в formatting options; при этом результат содержал 1387 последовательности, из которых гомологами можно считать 1319 (те, у которых Query cover не ниже 70%). Параметры поиска можно посмотреть здесь.
Рисунок 1: графическое представление результатов поиска
Задание 2. Так как все найденные в задании 1 белки принадлежат организмам домена Bacteria, я решил сократить условия поиска и выбрал таксономическую группу род Escherichia (taxid 561). Сравнив полученный список со списком задания 1, я выбрал одну последовательность, попавшую в оба - l-rhamnose isomerase организма Escherichia coli HS. В том, что эт одна и та же последовательность, нас убеждает одинаковый Sequence ID: ref|WP_004025918.1 и одинакова длина - 419 ак. Выравнивания и Score у находок полностью совпадают, а E-value изменилось (стало меньше). Это можно объяснить тем, что второй банк меньше, и, следовательно, E-value, как раз зависящее от размеров банка, тоже уменьшилось.
Задание 3. Для последоваельности sugar isomerase организма Butyrivibrio fibrisolvens (Sequence ID: WP_027207399.1) я выполнил выравнивание [условия запроса] и получил карту локального сходства:
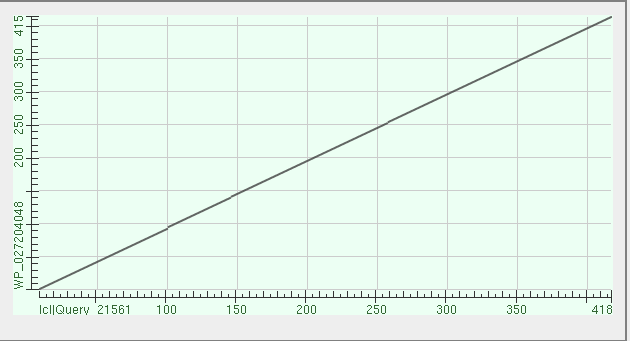
Рисунок 2: Карта локального сходства
Последовательности совпадают почти на всём своем протяжении, выравнивание содержит много консервативных и сходных позиций (сплошная линия на графике) и почти не содержит гэпов.
Задание 4. Для выполнения задания я взял выравнивание align_03.fasta из задания 8. Удалив из него все гэпы, я создал свою базу данных командой makeblastdb и затем осуществил в этой базе поиск последовательности своего белка командой blastp. Из двух находок была выбрана "лучшая" (такое деление весьма условно, тк величина E-value у обеих находок крайне велика):
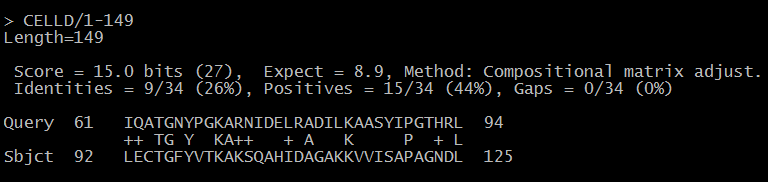
Рисунок 3: результаты поиска
В целом, это выравнивание гораздо "хуже" полученных ранее: величина E-value крайне велика даже при такой маленькой базе данных (8.9), bit score (15.0) и проценты совпавших (26%) и похожих (44%) аминокислотных остатков малы, выровнен небольшой участок. Всё это свидетельствует о случайности полученного выравнивания и полностью отрицает гомологию.
Назад к странице семестров