Семейства белковых доменов
При выполнении задания была использована последовательность белка L-rhamnose isomerase бактерии Bacteroides thetaiotaomicron (идентификатор Q8A1A2).

Рисунок 1: Домен pfam в последовательности белка
Ссылка на seed - выравнивание.
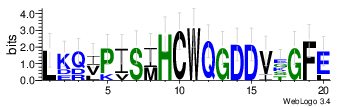
С помощью сайта http://threeplusone.com/weblogo/ был получен Logo выравнивания:
Также была получена консенсусная последовательность для этого участка:
>Consensus/1-19 Percentage Identity Consensus
LKQIPISIHCWQGDDV-GFE
.
Консенсусная последовательность всего белка.
Задание 4: использовался тот же блок, что и для построения Logo
- Сильный паттерн: L-[KDE]-Q-[IVL]-P-I-S-[IM]-H-C-W-Q-G-D(2)-V-[GETSK]-G-F-E
- Паттерн средней силы: L-[KDE]-[QDR]-[IVL]-[PK]-[IV]-S-[IM]-H-C-W-Q-G-D(2)-[VI]-[GETSK]-G-F-[EL]
- Слабый паттерн: L-x(5)-S-x-H-C-W-Q-G-D(2)-x(2)-G-F-x
Поиск последовательностей, содержащих мотив с данным паттерном, проводился на сайте
http://prosite.expasy.org/scanprosite/
по базе Swiss-Prot. Поиск по сильному паттерну дал только 3 находки, все - изомеразы L-рамнозы
(идентификаторы RHAA_BACHD,
RHAA_MANSM и
RHAA_OCEIH).
Поиск по "среднему" паттерну дал 33 находки, все - так же изомеразы L-рамнозы.
Список ID находок. Поиск по слабому паттерну выдал
67 находок,
все - опять же гомологи исходного белка.
Подобные результаты могут свидетельствовать либо о том, что
найденный мной паттерн сугубо специфичен для этого семейства, либо что выданного pfam'ом настолько
маленького (5 последовательностей) seed'а недостаточно для задания "нормального" паттерна.
Назад к странице семестров