Выравнивание геномов
Задание 1. Построение карты сходств двух родственных бактерий.
Для выполнения задания были выбраны следующие бактерии: Rickettsia prowazekii и
Rickettsia japonica.
Обе бактерии имеют одну хромосому, которые и были выравнены с помощью Blast2seq. На выход было получено выравнивание геномов (Рис.1) и карта локального сходства (Рис.2).

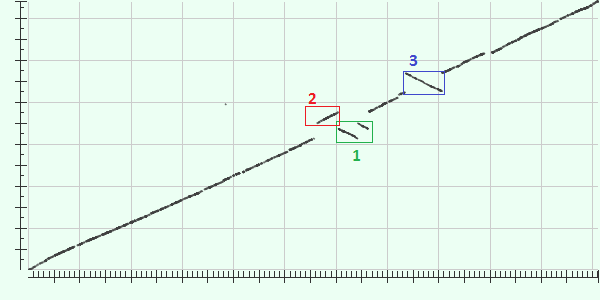
Значительные эволюционные изменения произошли на участках, выделенных на рисунке номерами 1, 2 и 3:
- На участке 1 произошли две последовательные инверсии.
- Между участками 1 и 2 произошла транслокация (участки поменялись местами).
- Участок 3 - еще одна инверсия.
Остальные участки гомологичны на довольно высоком уровне, что нам подтверждают данные рисунка 1: 92% идентичных позиций.
Задание 2. Описание сходства и различия геномов близкородственных бактерий.
Для сравнения я выбрал несколько штаммов вида Rickettsia prowazekii:
| Штамм | Идентификатор INSDC |
| Rickettsia prowazekii str. Breinl | CP004889.1 |
| Rickettsia prowazekii str. Dachau | CP003394.1 |
| Rickettsia prowazekii str. NMRC Madrid E | CP004888.1 |
| Rickettsia prowazekii str. Chernikova | CP003391.1 |
- Описание синтеничных участков - g-блоков - и их перестановок в геномах.
g-блоки состоят из последовательно идущих во всех геномах s-блоков, перемежающихся блоками других типов:
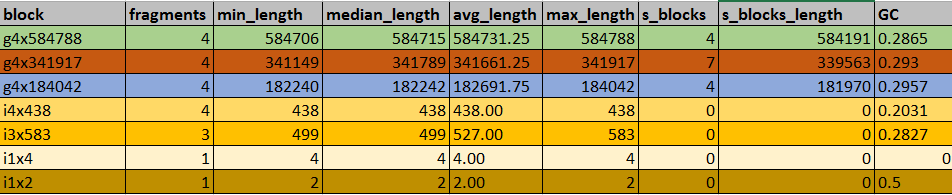
Рис.3. Список g-блоков
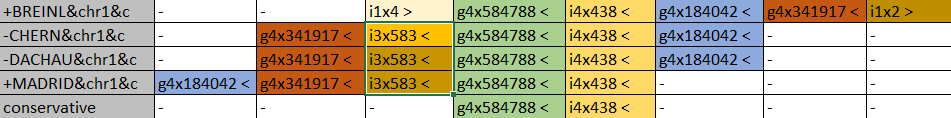
Рис.4. Выравнивание g-блоков Как видно из рисунка 3, всего наблюдается 7 блоков, выравненных на рисунке 4. Стоит заметить, что у штамма Rickettsia prowazekii str. Breinl отсутствуут блок i3x583, но вместо него присутствуют блоки i1x4 и i1x2, не представленные в других штаммах; позиции блоков g4x584788 и i4x438 являются консервативными для всех штаммов.
- Описание ядра геномов - s-блоков.
s-блоки - стабильные (коровые) блоки, по одному фрагменту из каждого генома.
Всего в четырех исследуемых геномах по 15 s-блоков в каждом геноме; их суммарная длина - 1105724, что составляет 99.59% от длины генома бактерии; процент консервативных позиций при объединении всех s-блоков равен 0.999; в среднем блок занимает 6,97% от длины генома бактерии. - Описание повторов на примерах r-блоков:
Блок Число фрагментов Длина Процент консервативных колонок Число генов r8x438 8 438 99% 0 r8x114 8 114 97% 4 - Описание крупных делеций на примерах h-блоков:
h-блоки - "полустабильные" блоки, содержащие по одному фрагменту из части геномов.
Блок Число фрагментов Длина Процент консервативных колонок Число генов h3x246 3 246 100% 3 h3x124 3 124 100% 3 - Описание уникальных последовательностей (u-блоки): Найденный u-блок - u1x1811 (Рис.5) - принадлежит только одному штамму, Rickettsia prowazekii str. NMRC Madrid E.
Последовательность блока была проанализирована алгоритмом blastn. Результат (Рис.6) показывает, что о параллельном переносе в этом случае вряд ли можно
говорить - все "хорошие" находки принадлежат бактериям того же рода, что и исследуемая.

Рис.5.U-block 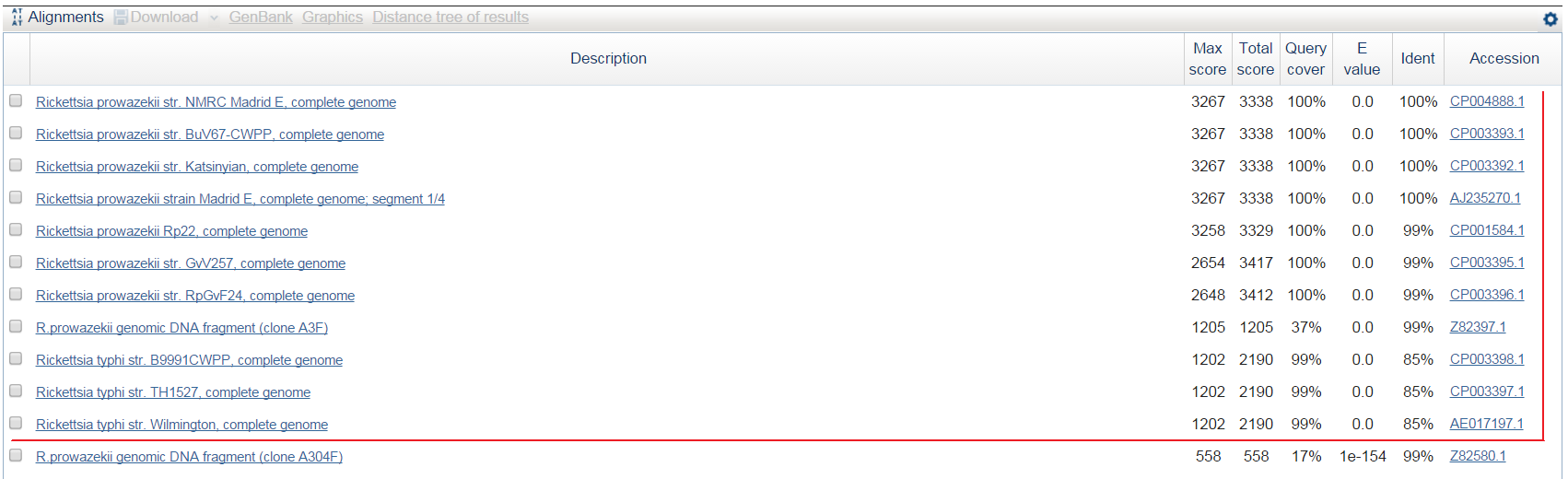
Рис.6.Выдача Blastn
- В блоке r8x343 qnpge выделяет только 2 гена, и оба - принадлежащие штамму NMRC Madrid E; первый кодирует тРНК-рибозилтрансферазу, содержащую гипермодифицированное основание Queuine, второй -
тРНК, переносящую лейцин (у человека кодируется митохондриальным геном MT-TL1).
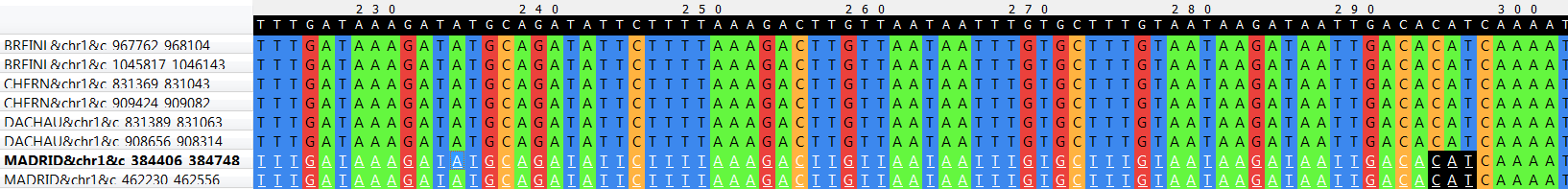
Pис.7.Расхождение в блоке r8x343 - В блоке r7x343 у всех четырех штаммов закодирован белок LicD (липополисахарид холинфосфотрансфераза, АС P14184), но у штаммов
NMRC Madrid E и Dachau в этом блоке дополнительно находится ген, кодирующий валин-тРНК лигазу (EC 6.1.1.9)
- фермент, катализирующий следующую реакцию:
ATP + L-valine + tRNAVal <-> AMP + diphosphate + L-valyl-tRNAVal
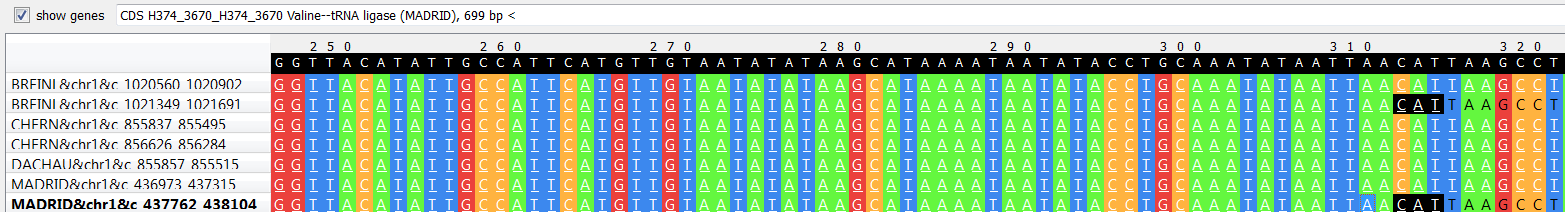
Pис.8.Расхождение в блоке r7x343
В целом, пакет программ npge показался мне не очень удобным в плане выдачи результатов, но он, безусловно, является важным для выполнения различных биоинформатических задач, а визуализатор qnpge значительно облегчает анализ.
Назад к странице семестров