Нуклеотидный blast
Задание 1. (Также процитировано в практикуме 7). С помощью нуклеотидного BLAST'а (BLASTN) была предпринята попытка установить, какому гену какого организма принадлежит последовательность, полученная в практикуме 6.
Результат выглядит так:
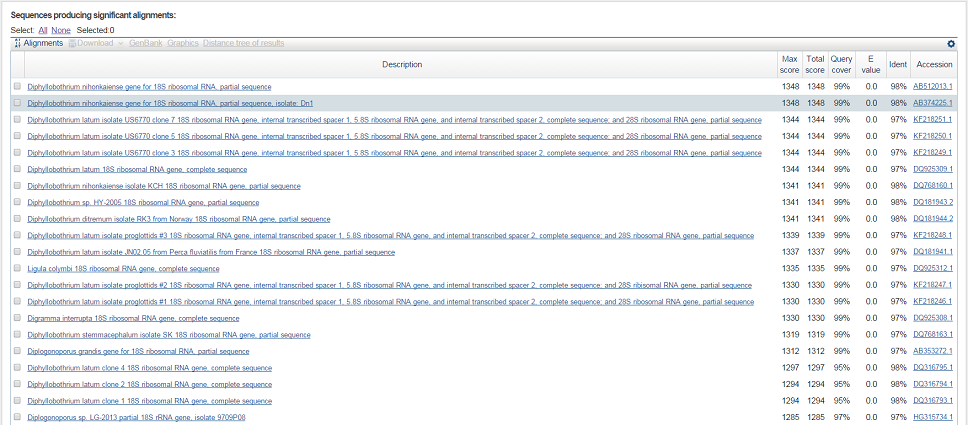
Как можно заметить, довольно большое количество полученных последовательностей имеют высокую степень сходства и исходной (E-value=0.0, уровень сходства 97-98%), но наиболее подходящими являются первые два результата (наивысший score). Следовательно, полученная в практикуме 6 последовательность, вероятнее всего, является геном 18S рибосомальной РНК лентеца Diphyllobothrium nihonkaiense (он же D. klebanovskii) - уровень сходства 98%, Query cover 99%, score 1348.
Ближайшая находка из другого вида того же рода - Diphyllobothrium latum 18S ribosomal RNA gene, complete sequence: уровень сходства 97%, Query cover 99%, score 1344.
Выравнивание лучших 10 находок с исходной последовательностью представлено здесь.

Задание 2.
В этом задании требовалось провести поиск тремя алгоритмами
blast: blastn, megablast, discontiguous megablast. Для лимитирования количества находок
в параметрах поиска всех алгоритмо были внесены дополнительные условия:
- Указан род: Diphyllobothrium (taxid:28844).
- Изменены штрафы за гэпы: Existence: 2, Extension: 1
- Изменена "стоимость" параметра score: Match: 1, Mismatch: -1
| Алгоритм | Число находок | E-value худшей находки | Сходство худшей находки | Примеры, найденные только этим алгоритмом |
| blastn | 65 | 8.3 | 74% | LL622094.1, LL592749.1 |
| megablast | 41 | 0.022 | 81% | - |
| discontiguous megablast | 64 | 7.7 | 80% | - |
Задание 3.1
Поиск по базе Refseq результатов не дал в принципе,
поэтому была использована база nr/nt; даже в ней поиск по виду, роду и даже семейству
результатов не давал, область поска была расширена до класса Cestoda (taxid:6199). Объяснить это можно тем, что ленточные черви - паразиты, утратившие пищеварительную систему и живущие за счет организма-хозяина,
и их метаболические пути отличаются от млекопитающих, следовательно, могут отличаться и белки, принимающие участие этих путях. Малые значения параметров Identity и Query cover у некоторых находок можно объяснить тем,
что чем белок важнее, тем он консервативнее, и наоборот.
| Белок | AC | fasta | Число находок | Лучшая находка | Ident | Query cover |
| HSP7C_HUMAN (Heat shock cognate 71 kDa protein) | P11142; Q9H3R6; | HSP7C_HUMAN | 263 | Hymenolepis nana genome assembly H_nana_Japan, scaffold HNAJ_scaffold0000713 |
81% | 94% |
| TERT_HUMAN (Telomerase reverse transcriptase) | O14746; O14783; Q2XS35; Q8N6C3; Q8NG38; Q8NG46; |
TERT_HUMAN | 1 | Spirometra erinaceieuropaei genome assembly S_erinaceieuropaei, scaffold SPER_scaffold0114830 |
29% | 8% |
| CISY_HUMAN (Citrate synthase, mitochondrial) | O75390; Q71UT9; Q7KZH0; Q96FZ8; Q9BWN8; |
CISY_HUMAN | 13 | Hymenolepis diminuta genome assembly H_diminuta_Denmark, scaffold HDID_contig0003283 |
52% | 92% |
| RPB1_HUMAN (DNA-directed RNA polymerase II subunit RPB1) | P24928; A6NN93; B9EH88; Q6NX41; |
RPB1_HUMAN | 67 | Hymenolepis diminuta genome assembly H_diminuta_Denmark, scaffold HDID_scaffold0000835 |
65% | 76% |
| PABP2_HUMAN (Polyadenylate-binding protein 2) | Q86U42; D3DS49; O43484; | PABP2_HUMAN | 88 | Hydatigera taeniaeformis genome assembly H_taeniaeformis_Canary_Islands, scaffold TTAC_scaffold0000027 |
70% | 31% |
Для иллюстрации приведены выдачи blast'а для двух из пяти белков:
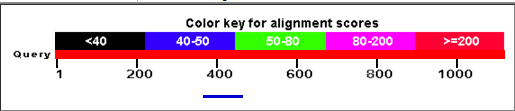 |
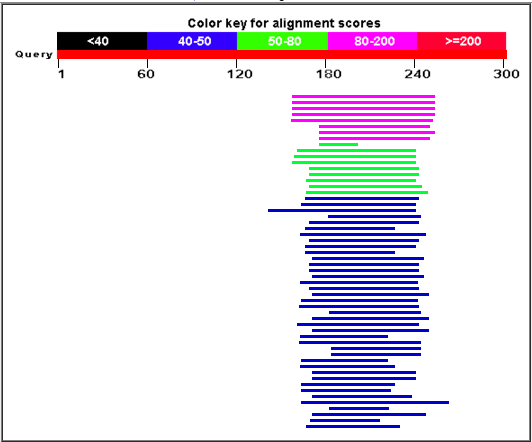 |
Часть II
Задание 4.
Для исследования было выбрано пять вирусов рода Tobamovirus:
- Tobacco mosaic virus
- Clitoria yellow mottle virus
- Tomato mottle mosaic virus
- Bell pepper mottle virus
- Wasabi mottle virus
Геномы всех вырусов были сохранены в одном fasta-файле. После этого
была создана база индексов blast для созданного fasta файла: makeblastdb -in tobamovirus.fasta -dbtype nucl. После этого был запущен tblastx, на вход которому был задан тот же самый fasta-файл:
tblastx -query tobamovirus.fasta -db tobamovirus.fasta -out tobamov.out -outfmt 7; на выход была получена таблица tobamov.out, из которой с помощью
скрипта были удалены неинформативные и слабо сходные находки. Для этого скрипт был запущен со следующими
параметрами:
python revise_blast_7.py --infile tobamov.out --identity 40 --max_exp 5 --min_bits 20 --outfile virustable
В результате был получен файл, преобразованный в excel-таблицу.
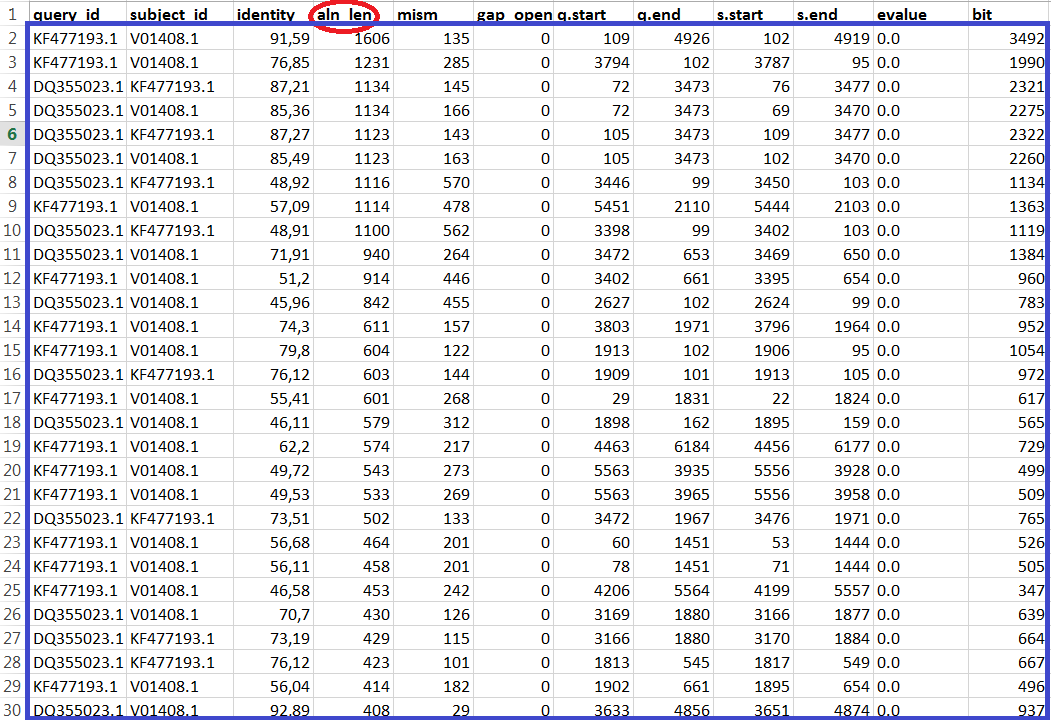
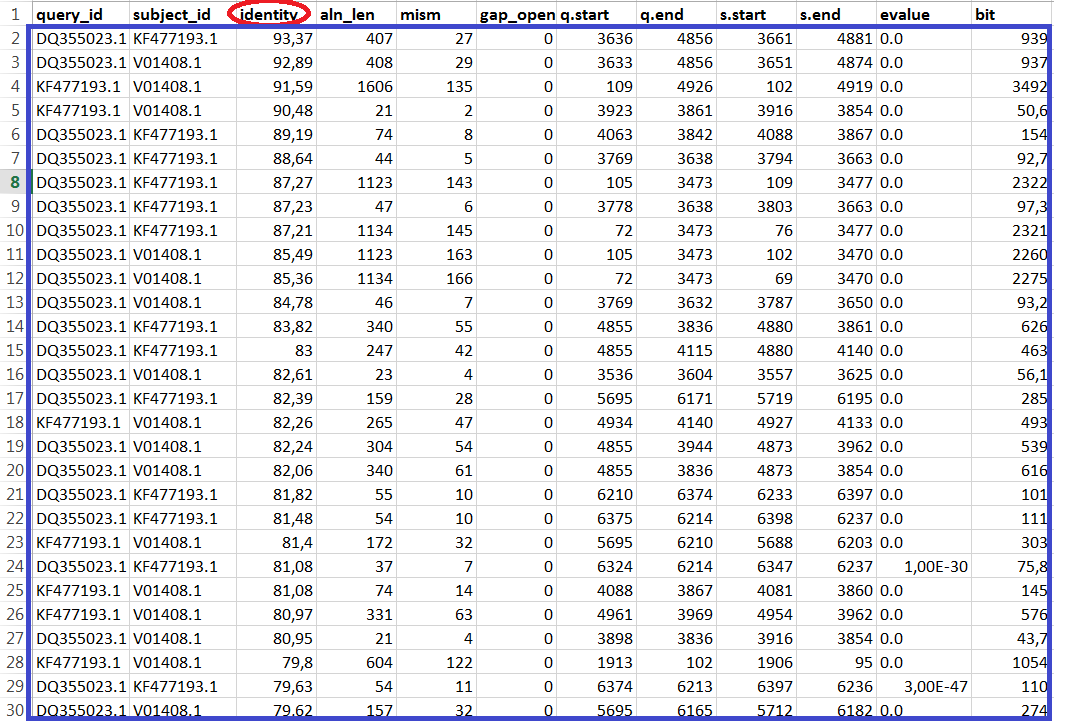
Таблица была отсортирована по значению identity, длине выравнивания и его счёту (рис. 4-6). Как оказалось, находки с наибольшим процентом идентичных нуклеотидов, наибольшей длиной выравнивания и максимальным счетом представляют собой сходные участки трёх вирусов : KF477193.1, V01408.1 и DQ355023.1 - соответственно Tobacco mosaic virus, Tomato mottle mosaic virus и Bell pepper mottle virus. Полученные данные позволяют признать, что геномы этих трёх вирусов наиболее схожи.
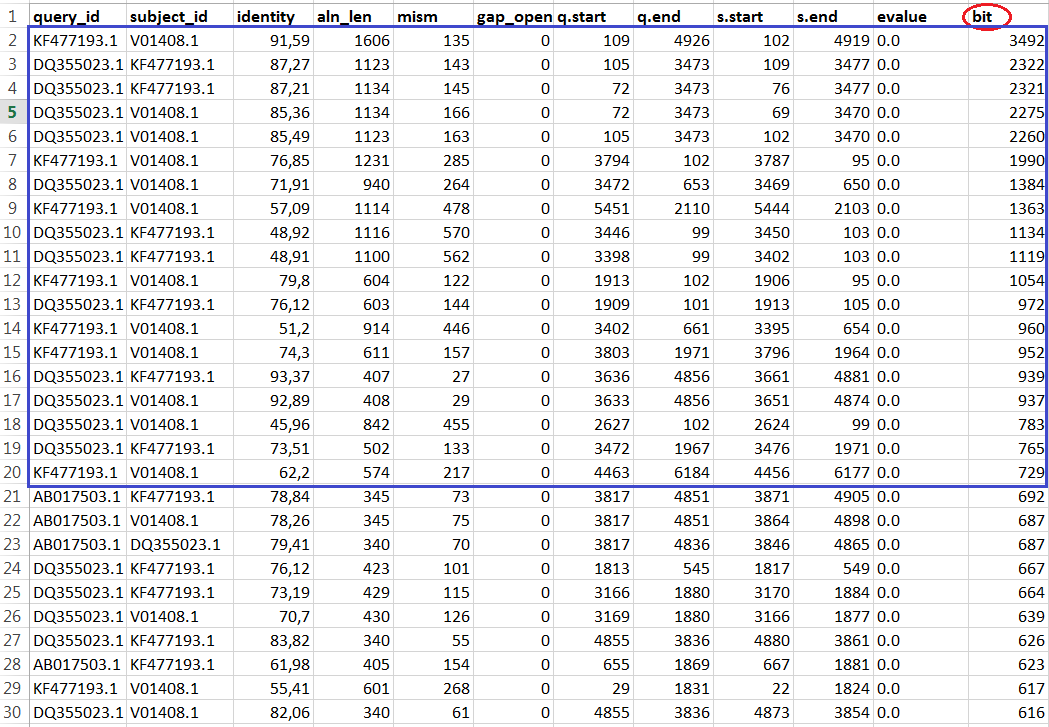 |
 |
 |
Назад к странице семестров