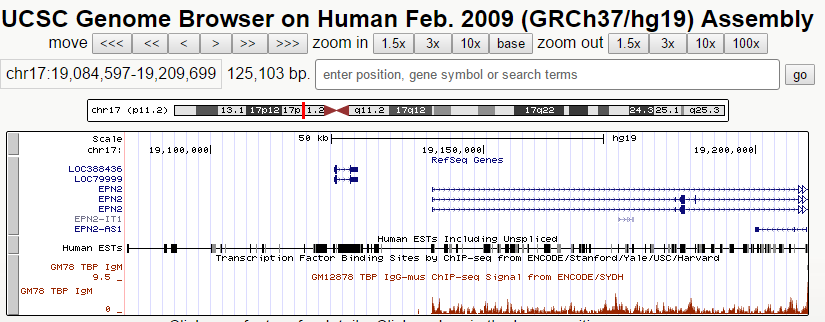
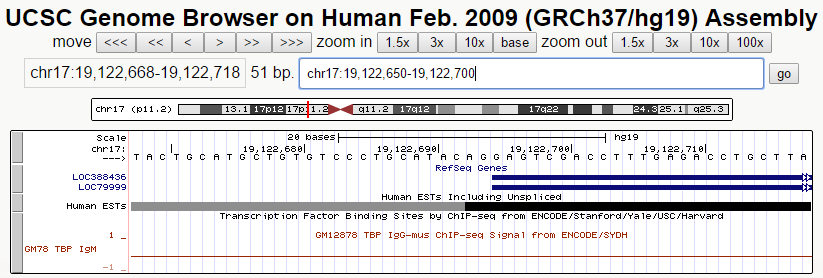
Поиск сигналов
Мне был выдан файл chipseq_chunk51.fastq, содержащий риды Illumina одного из участков хромосомы человека. Был проведен контроль качества ридов с помощью команды fastqc chipseq_chunk51.fastq. Результат выполнения команды - zip-архив - содержал файл fastqc_report.html, в котором содержался отчёт о проделанной программой работе. В том числе на этой странице находится и представленная на Рис.1 таблица с оценкой качества чтений:
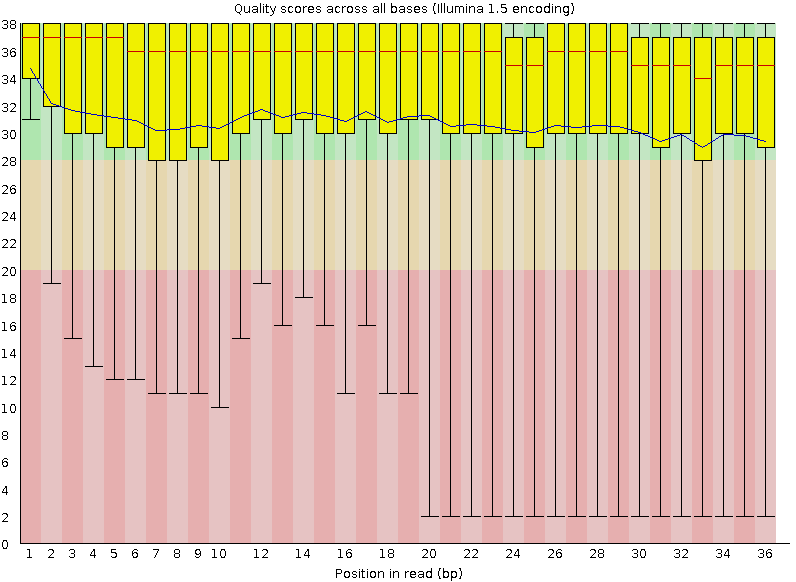
Всего ридов 12221. Как видно из рисунка, качество чтения очень высокое на всём протяжении последовательности, нет ридов со score < 28. Поэтому программа Trimmomatic для очистки чтений от участков плохого качества не применялась.
После прочтения были картированы на проиндексированный геном человека:
- bwa mem /srv/databases/ngs/hg19/GRCh37.p13.genome.fa chipseq_chunk51.fastq > chipseq_chunk51.sam
Проведем анализ полученного выравнивания:
- samtools view -b chipseq_chunk51.sam -o aln.bam - переведем его в бинарный формат;
- samtools sort aln.bam out.predix.bam - отсортируем по координате в референсе начала чтения;
- samtools index out.predix.bam - проиндексируем полученный файл;
- samtools idxstats out.predix.bam - узнаем, сколько ридов было откартировано. Результат - на рис.2:
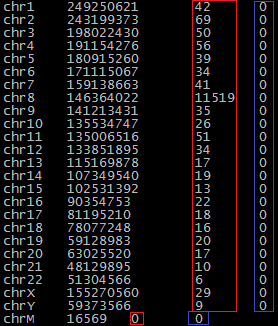
Как и ожидалось, на хромосому картировались все риды без исключения. При этом на митохондриальную хромосому (chrM) не картировалось ни одного рида, а подавляющее большинство (11519, или 94%) были картированы на восьмую хромосому. Логичным будет предположить, что именно с неё и были почитаны риды.
Как и ожидалось, запущенная со стандартными параметрами программа MACS не нашла достаточно пиков. Поэтому она была запущена со следующими параметрами:
- macs2 callpeak -n chunk51 -t chipseq_chunk51.sorted.bam --nomodel
Итогом работы программы являются три выходных файла:
Всего программа нашла 8 пиков, средняя длина пика - 341 нуклеотид. Для визуализации пиков в UCSC Genome Browser был выбран файл chunk51_peaks.narrowPeak. В него были добавлены строки
track type=narrowPeak visibility=3 db=hg19 name="my_peaks" description="Peaks from chunk 51" browser position chr8:12953962-13402563,
после чего он был загружен в форму custom tracks в UCSC Genome Browser. Скриншот- на рис.3:
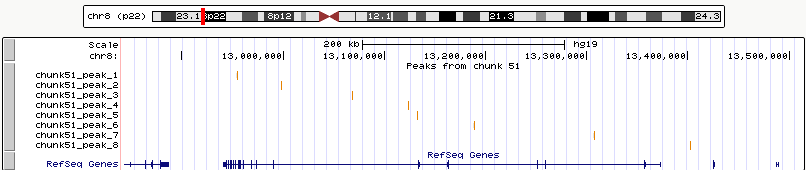
Рассмотрим подробнее пики 7 и 5.
| Пик | 5 | 7 |
| Длина пика | 441 | 292 |
| -LOG10(pvalue) | 51.07661 | 16.68596 |
| Вершина пика относительно начала | 181 от начала пика | 175 от начала пика |
| Положение в геноме | Оба пика пересекают экзоны гена DLC1, кодирующего белок GAP (GTPase-activating protein). Этот ген функционирует как онкосупрессор для нескольких видов рака, таких как рак легких или простаты. |
|
Чем больше величина -LOG10(pvalue), тем меньше числовое значение pvalue и, следовательно, тем достовернее пик. Таким образом, пик 5 является самым достоверным из всех восьми.
Задание 4. Инициация с помощью ТАТА-бокс связывающего белка
TBP, или ТАТА-бокс связывающий фактор - архейный и эукариотический белок, узнающий восьминуклеотидный сигнал в ДНК с консенсусом TATAWAAR. Он является одним из ключевых ДНК-узнающих белков при образовании на промоторе генов комплекса TFIID инициации транскрипции с помощью Pol II. Тем не менее, лишь часть промоторов имеет сигнал TATA-box, связываемый TBP. Для выполнении этого задания требовалось найти в геноме человека три гена, имеющих такой сигнал, и один ген, не имеющий сигнала. Поиск велся с помощью сервиса UCSC GenomeBrowser. Для белка TBP (TATA binding protein) был выбран ChiP-Seq-эксперимент, проведенный на клеточной линии GM12878. В эксперименте использовались антитела к TBP ab62126.
Первый ген, у которого нашелся пик - ген FOSB. Семейство генов Fos включает в себя 4 гена: FOS, FOSB, FOSL1 и FOSL2. Они кодируют белки - лейциновые молнии, которые учавствуют в димеризации белков семейства JUN, формируя комплекс - транскрипционный фактор AP-1. Размер кодирующей последовательности гена - 7185 нуклеотидов. В геноме расположен на "+"-цепи 19 хромосомы на позиции 45971253-45978437. Имеет 3 экзона.
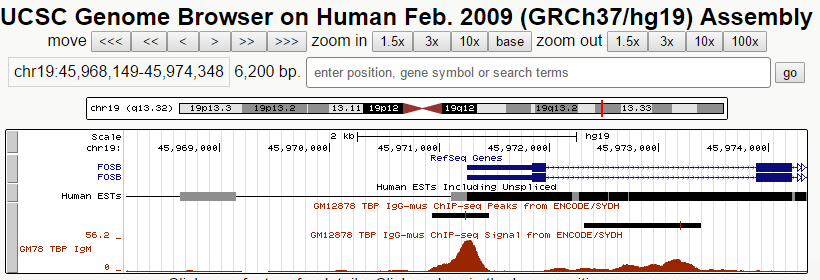
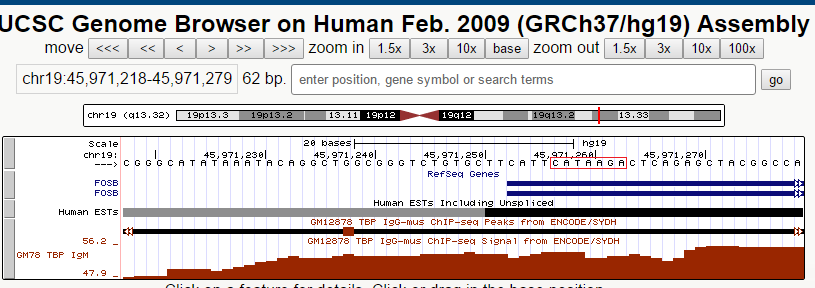
Второй ген - FAAP24. Продукт гена является частью ядра комплекса FA (Fanconi anemia), играющего важную роль в клеточном ответе на повреждение ДНК (Ciccia et al., 2007 [PubMed 17289582]). Размер кодирующей последовательности гена - 5279 нуклеотидов. В геноме расположен на "+"-цепи 19 хромосомы на позиции 33463123-33468401. Имеет 5 экзонов.
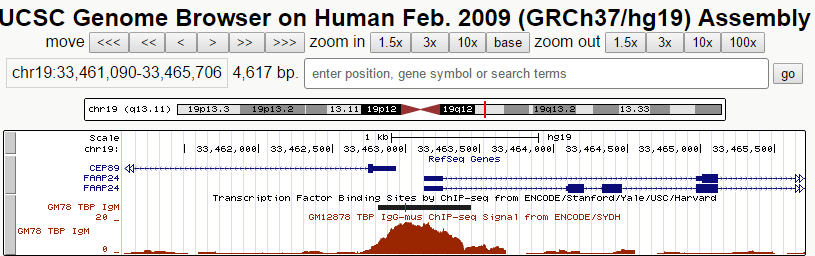
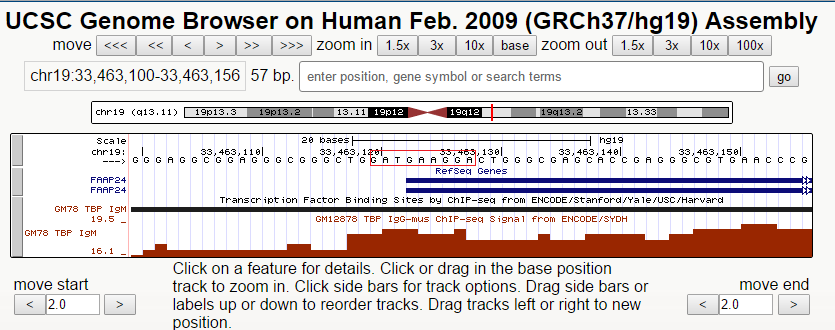
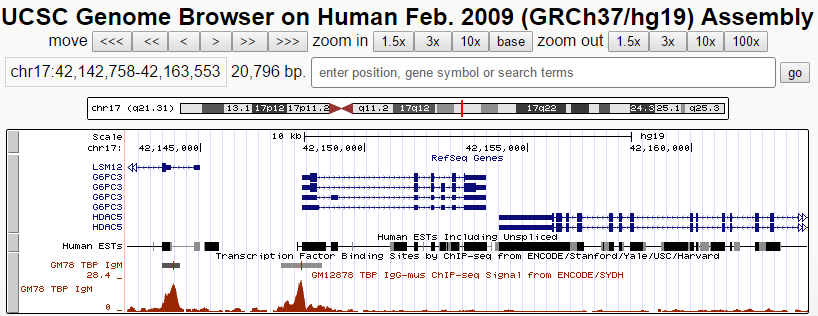
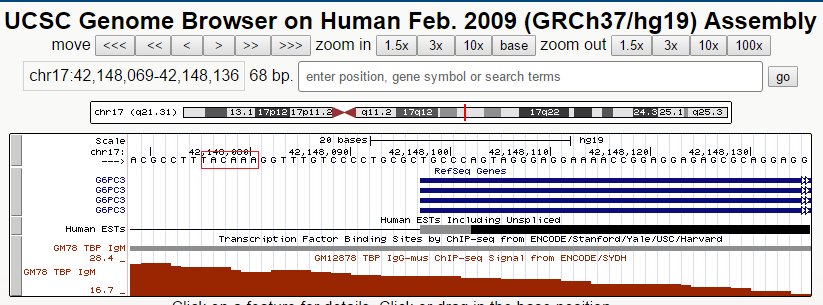
Третий ген - G6PC3. Кодирует каталитическую субъединицу глюкозо-6-фосфатазы (G6Pase). Этот белок работает в ЭПР, где катализирует гидролиз глюкозо-6-фосфата. Размер кодирующей последовательности гена - 6515 нуклеотидов. В геноме расположен на "+"-цепи 17 хромосомы на позиции 42148098-42153712. Имеет 7 экзонов.
В качестве примера гена без связывающего мотива был выбран ген LOC388436, кодирующий белок LOC388436. Размер кодирующей последовательности гена - 4349 нуклеотидов. В геноме расположен на "+"-цепи 17 хромосомы на позиции 19057837-19062185. Имеет 3 экзона.