Филогенетические деревья
Задание 1.
Для выполнения задания требовалось поработать с филогенетическим деревом восьми бактерий. Для работы были выбраны следующие штаммы бактерий:
- Ralstonia pickettii (RALPJ)
- Pseudomonas aeruginosa (PSEAE)
- Erwinia tasmaniensis (ERWT9)
- Salmonella typhimurium (SALTY)
- Yersinia pseudotuberculosis (YERPS)
- Vibrio fischeri (VIBFM)
- Pasteurella multocida (PASMU)
- Proteus mirabilis (PROMH)
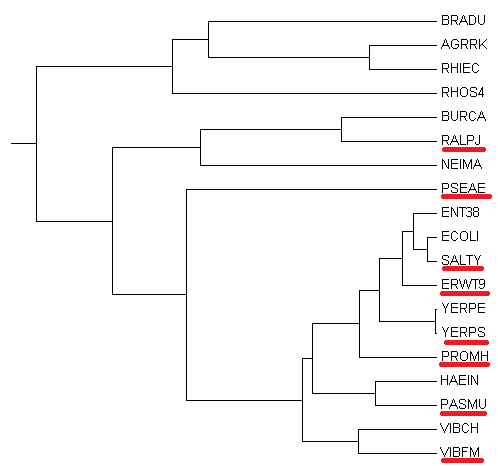
Взяв за основу дерево, приведенное на рис.1, требовалось построить филогенетическое дерево выбранных бактерий, приведя скобочную формулу и ее графический аналог:
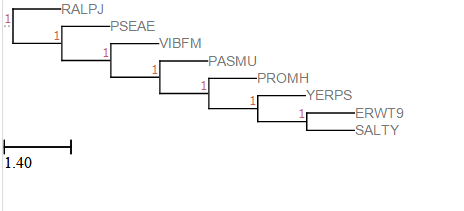
Для визуализации дерева был использован онлайн-сервис newick tree viewer:
3.Данное дерево содержит следующие нетривиальные ветви:
- (RALPJ, PSEAE) против (VIBFM, PASMU, PROMH, YERPS, ERWT9, SALTY)
- (RALPJ, PSEAE, VIBFM) против (PASMU, PROMH, YERPS, ERWT9, SALTY)
- (RALPJ, PSEAE, VIBFM, PASMU) против (PROMH, YERPS, ERWT9, SALTY)
- (RALPJ, PSEAE, VIBFM, PASMU, PROMH) против (YERPS, ERWT9, SALTY)
- (RALPJ, PSEAE, VIBFM, PASMU, PROMH, YERPS) против (ERWT9, SALTY)
Задание 2.
Используя таксономический сервис NCBI, я определил таксономию выбранных ранее организмов:

Как мы видим, все организмы принадлежат к типу Proteobacteria. При этом нетривиальная ветвь (RALPJ, PSEAE, VIBFM, PASMU) против (PROMH, YERPS, ERWT9, SALTY) отделяет таксон Enterobacteriales.
Задание 3.
При выполнении прошлого задания было построено филогенетическое дерево белка RSMH (16S рРНК-метилтрансфераза H) семи выбранных ранее бактерий, приведённое ниже на рисунке 4.
После этого требовалось переукоренить его в среднюю точку. Для этого тбыла использована программа retree пакета PHYLIP. Полученное переукорененное дерево открыто в программе MEGA:
Назад к странице семестров