Реконструкция филогении по нуклеотидным последовательностям. Паралоги.
Задание 1. Построение дерева по нуклеотидным последовательностям
Требовалось построить филогенетическое дерево тех же бактерий, что в предыдущих заданиях, используя последовательности РНК малой субъединицы рибосомы (16S rRNA). Для этого:
- Последовательности 16S rRNA бактерий нужных видов были взяты из файлов с расширением .frn в записи, принадлежащей хромосоме каждой из бактерий, на
сайте NCBI; последовательности были помещены в
fasta-файл с отредактированными названиями (оставлена только мнемоника вида). - Далее, последовательности были импортированы в Jalview и выровнены программой Muscle.
- Дерево построено в программе MEGA6 методом Neigbour-Joining:
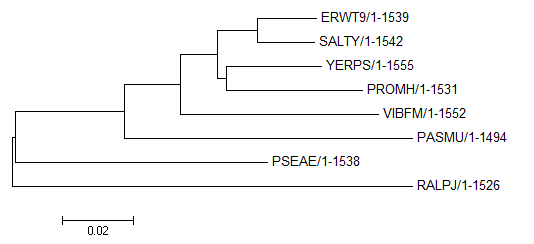
Теперь сравним полученное дерево с деревом, построенным по белковой последовательности:
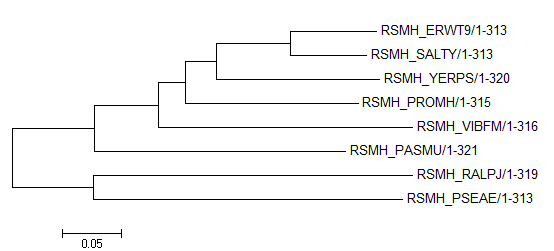
При сравнении двух рисунков видно, что деревья не отличаются расположением ветвей, различны только их длины. Это даёт более точную информацию, следовательно, точность реконструкции в случае анализа последовательностей РНК выше. Учитывая этот факт, а также то, что гены рРНК одни из наиболее консервативных, можно сделать вывод, что систематическое положение организма и время расхождения с близкими видами следует определять именно на основании анализа сходств и различий в последовательностях рРНК.
Задание 2. Построение и анализ дерева, содержащего паралоги.
Требовалось найти в протеомах бактерий достоверные гомологи белка CLPX_ECOLI и построить их дерево. Для начала я объединил протеомы всех бактерий
в один файл:
cat *.fasta >> prot.fasta
После этого был проведен blastp последовательности белка CLPX_ECOLI по полученному файлу с протеомами:
makeblastdb -in prot.fasta -dbtype prot -out blastdb.fasta
blastp -query ecoli.fasta -db blastdb.fasta -evalue 0.001 -outfmt 6 -out out.fasta
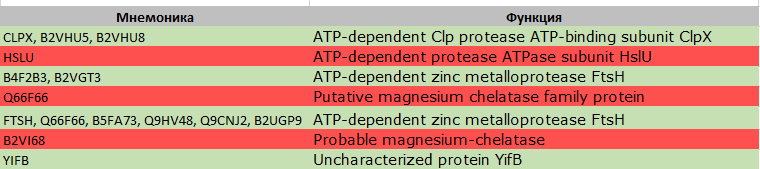
Всего в выдаче blast'а 36 находок. Их мнемоники и функции представлены на
рисунке 3:
Все 36 последовательностей были импортированы в Jalview и выровнены программой Muscle; дерево построено в программе MEGA6 методом Neighbor-Joining. Результат приведен на рисунке 4.
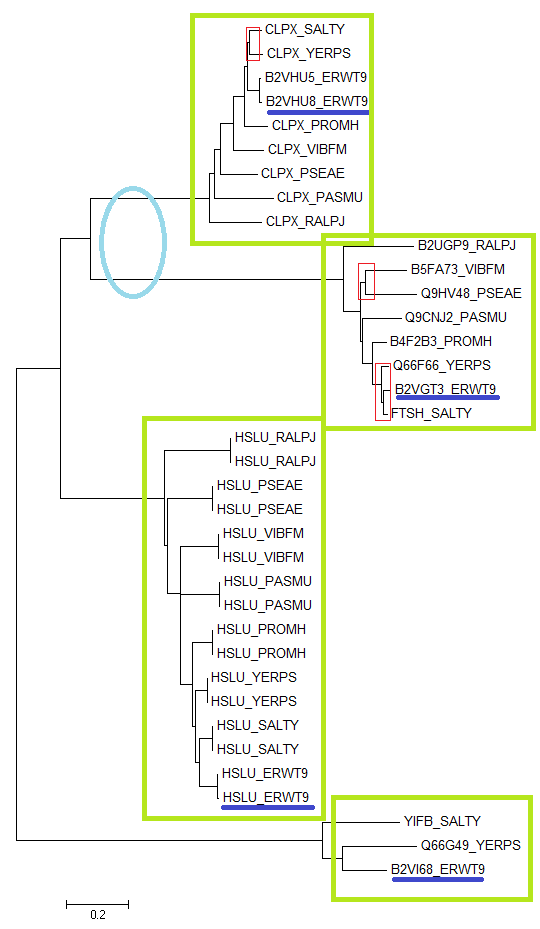
Учитывая, что под паралогами понимаются два гомологичных белка из одного организма, а под ортологами - гомологичных белка, если они: а) из разных организмов; б) разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования, приведем примеры следующих эволюционных событий на дереве:
- Салатовыми рамками выделены ортологичные группы;
- Голубой обруч - место дупликации гена - два белка выполняют разные функции и/или просто являются разными, а присутствуют у почти всех организмов;
- Пример паралогов - синее подчёркивание;
- Красные рамки - пример разделения путей эволюции белков в результате видообразования (небольшие ортологичные группы).
Назад к странице семестров