Построение и визуализация электронной плотности
Белок состоит из четырех цепей, длина одной цепи 438 аминокислотных остатков, теоретический вес белка - 48 КДа. Разрешение структуры 1.84Å.
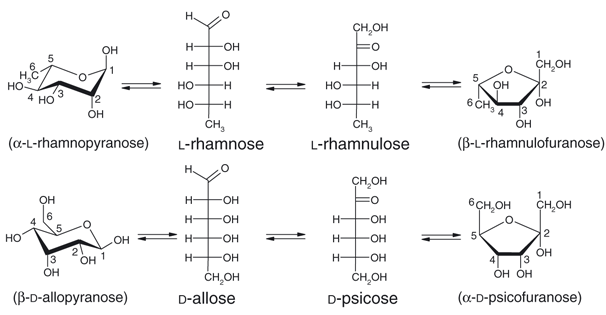
Изомераза L-рамнозы катализирует обратимую изомерацию L-рамнозы в L-рамнулозу, и является одним из ферментов пути метаболизма рамнозы у многих микроорганизмов, например у E. coli. Исследуемый в данной работе гомолог из Pseudomonas stutzeri интересен тем, что помимо этой реакции также может катализировать обратимую изомеризацию D-aллозы в D-псикозу (см. рис. 1).
Структура 3ITY была получена в результате рентгеноструктурного анализа японскими учёными и описана в работе 2010 года. Целью данного исследования было изучение механизма катализируемой ферментом реакции.
Задание 1.
Задания выполнены в программе Pymol версии 1.7.2.1.
В сессии Pymol были одновременно открыты .ent файл со структурой и .ccp файл с картой электронной плотности. Просле этого для визуализации электронной плотности вокруг остова полипептидной цепи белка на разных уровнях подрезки были применены следующие команды:
select backbone, (name ca,n,c,o) color brightorange, backbone show sticks, backboneВыделили атомы остова в переменную backbone, покрасить их в оранжевый цвет и визуализовать "палками".
isomesh grid, 3ity, x, backbone, carve=2- создает объект grid с сеткой электронной плотности из карты 3ity с уровнем подрезки х для атомов backbone, на расстоянии не дальше 2Å от центра атома. На рисункe 2 представлено изображение электронной плотности вокруг полной структуры 3ITY на уровне подрезки 0.5:
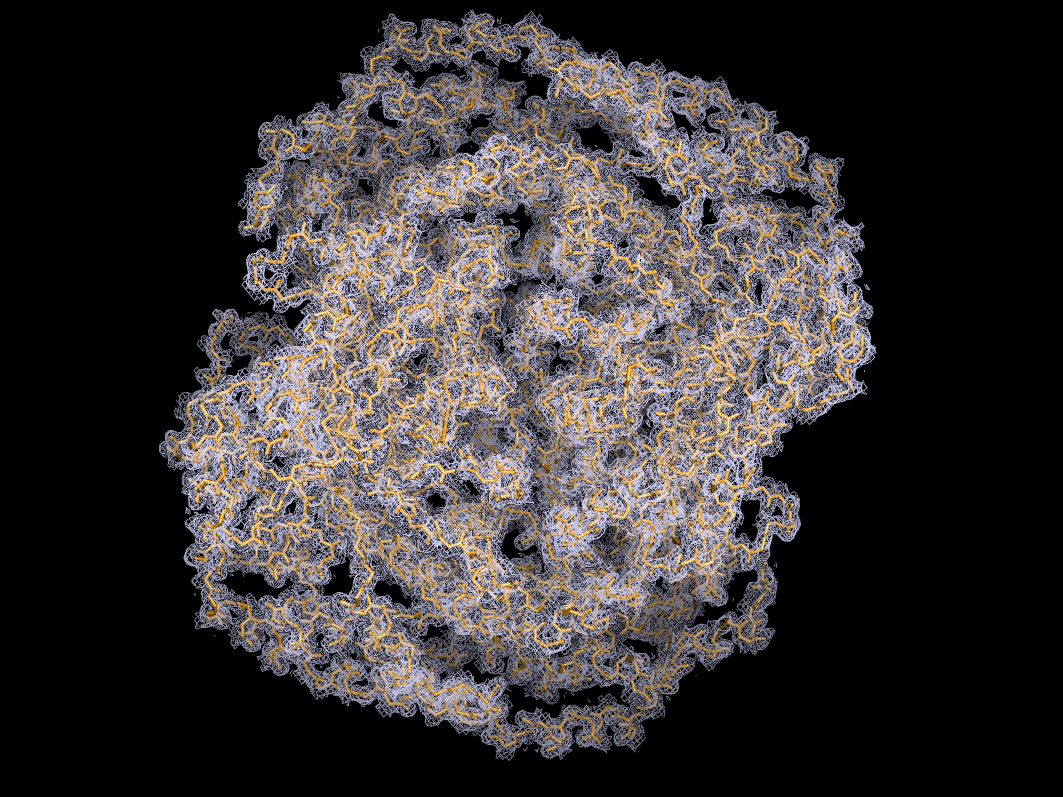
Структура состоит из четырех идентичных цепей, и смотреть на все сразу неудобно, поэтому для визуализации уровней подрезки эл.плотности на разных уровнях подрезки была выбрана цепь А:
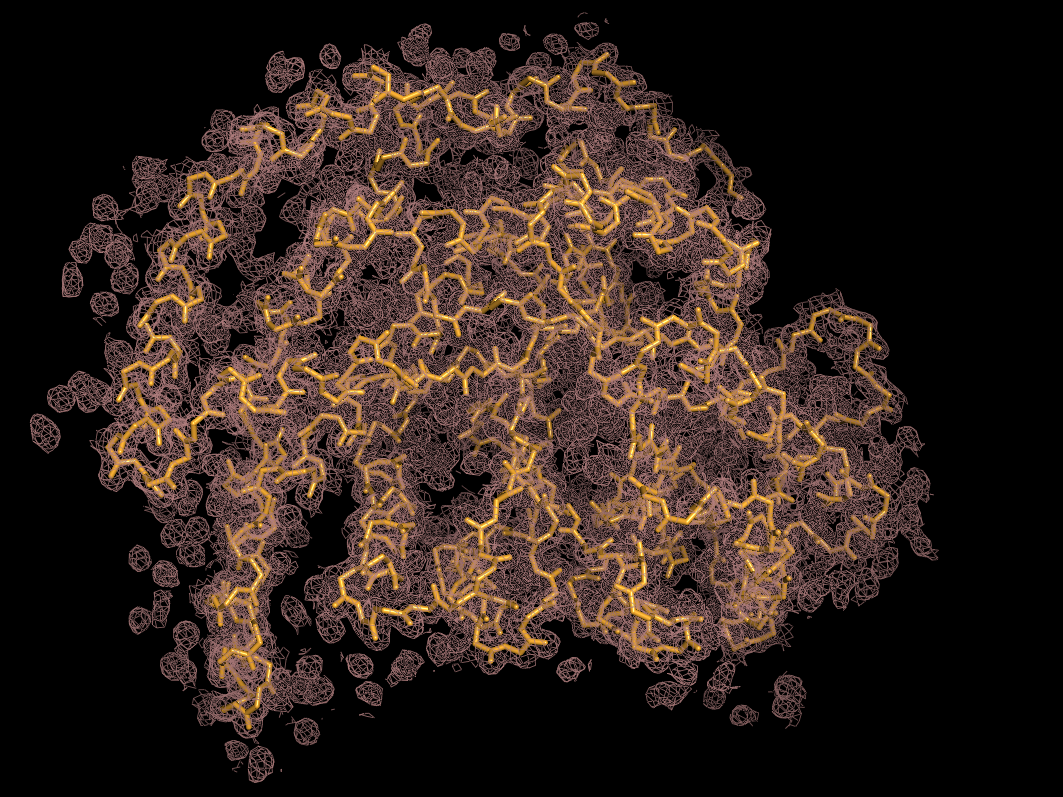
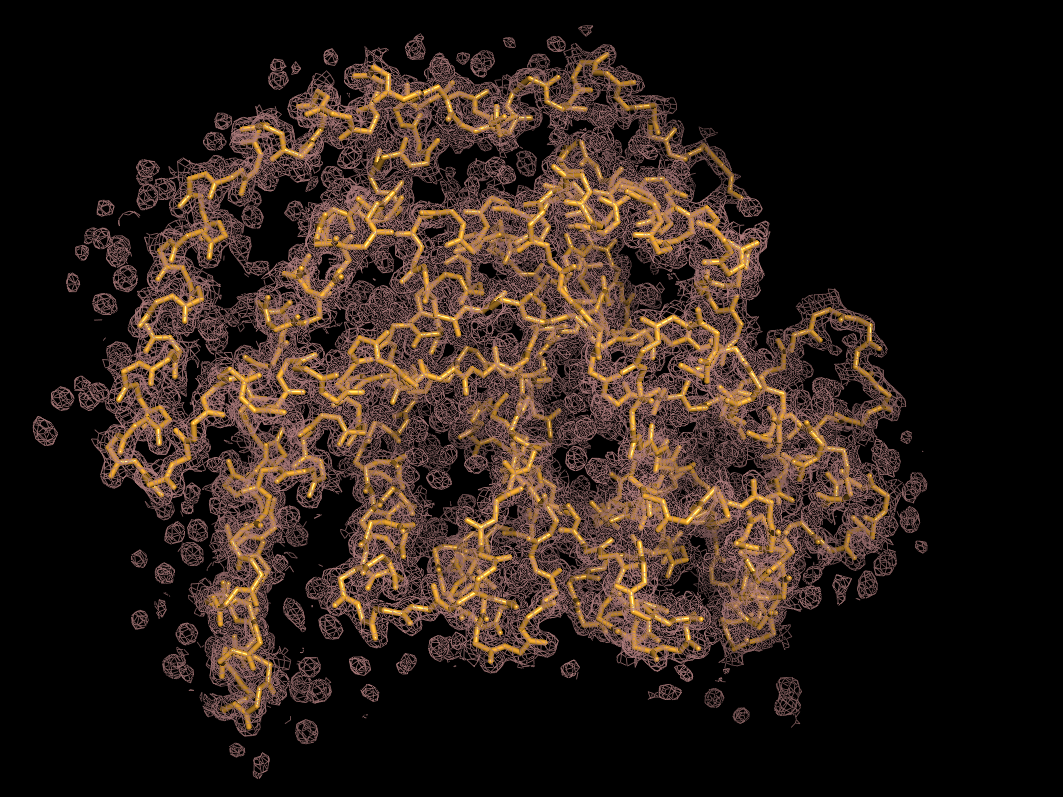
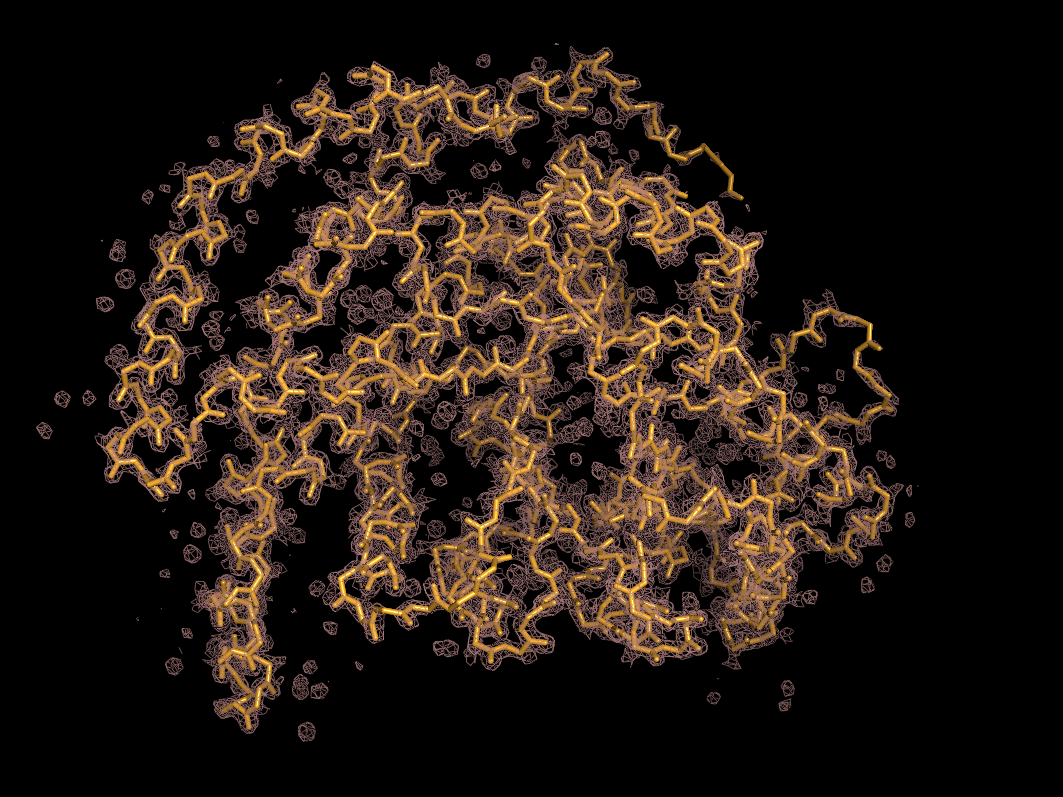
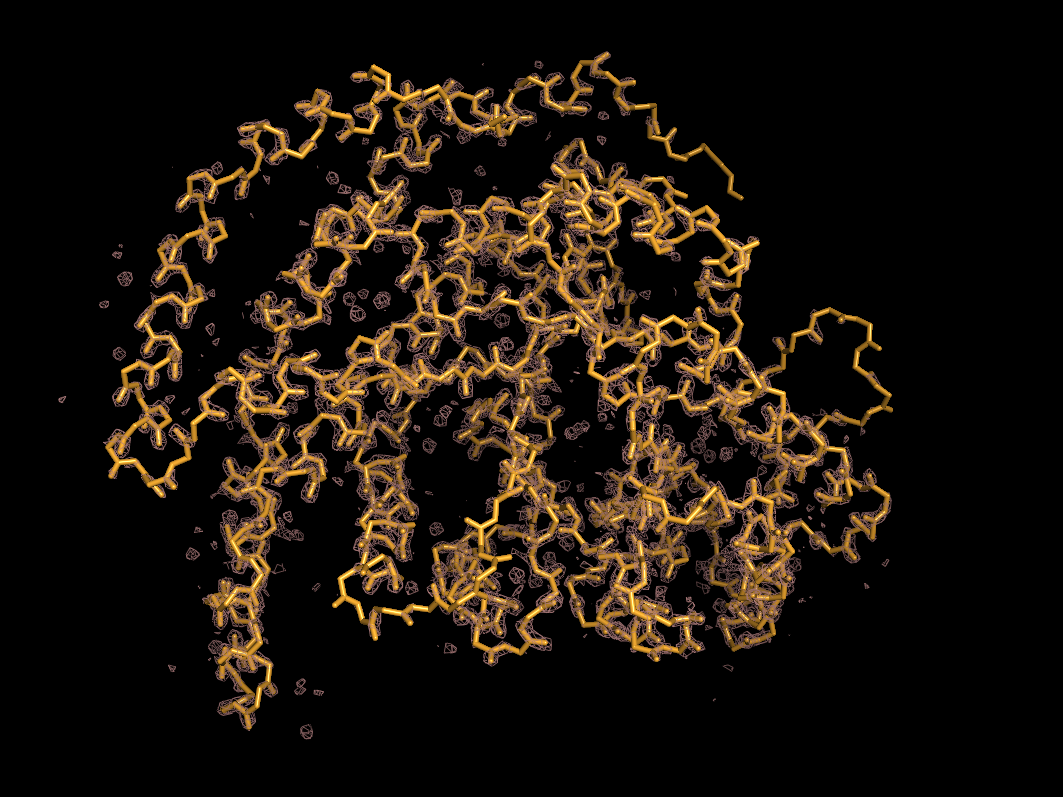
Электронная плотность покрывает полипептидную цепь и достаточно достоверно отображает расположение атомов, однако уже на уровне подрезки 1.0 часть боковых цепей остатков не покрыта плотностью, а на уровне 2.0 плотность не покрывает и часть атомов остова.
Задание 2.
Для визуализации были выбраны различные по функциональным группам остатки trp57, met325, asp 327. Электронная плотность вокруг них была визуализирована следующими командами:
select residues, (resi 57,327,325) and chain A hide all show sticks, residues isomesh grid, 3ity, x, residues, carve=2 color smudge, grid- выделить три интересующих остатка в группу residues, оставить на экране только их, показать вокруг них карту электронной плотности с уровнем подрезки х и покарсить в цвет smudge.
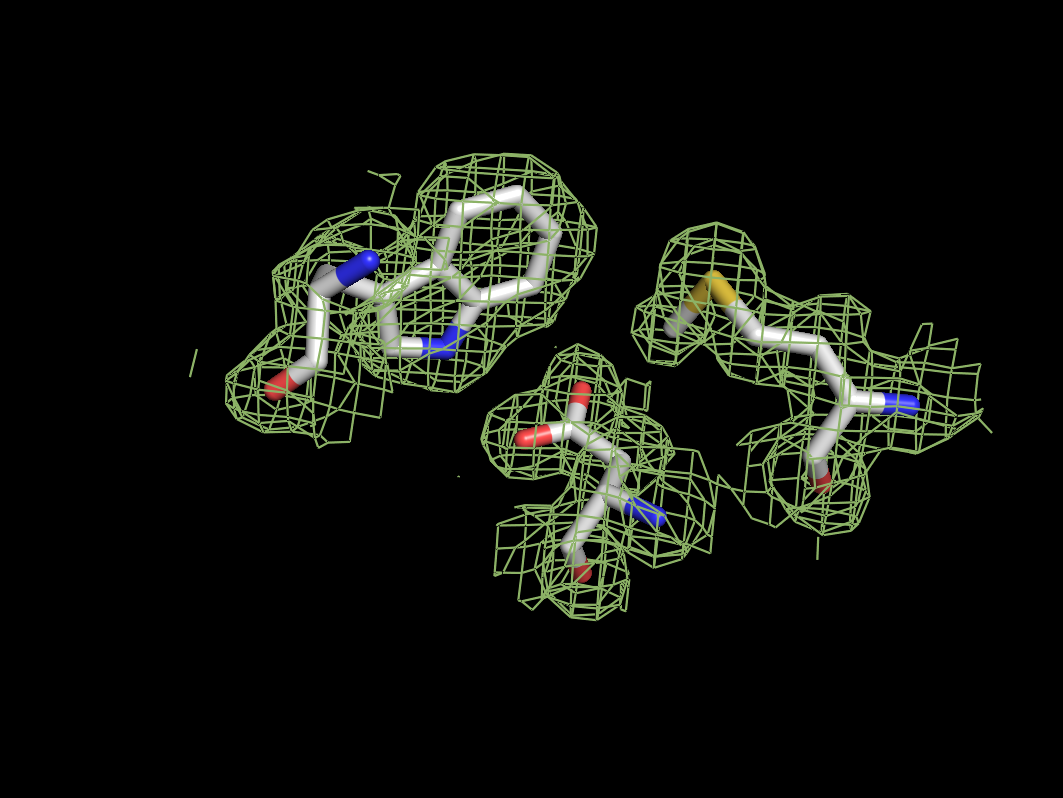
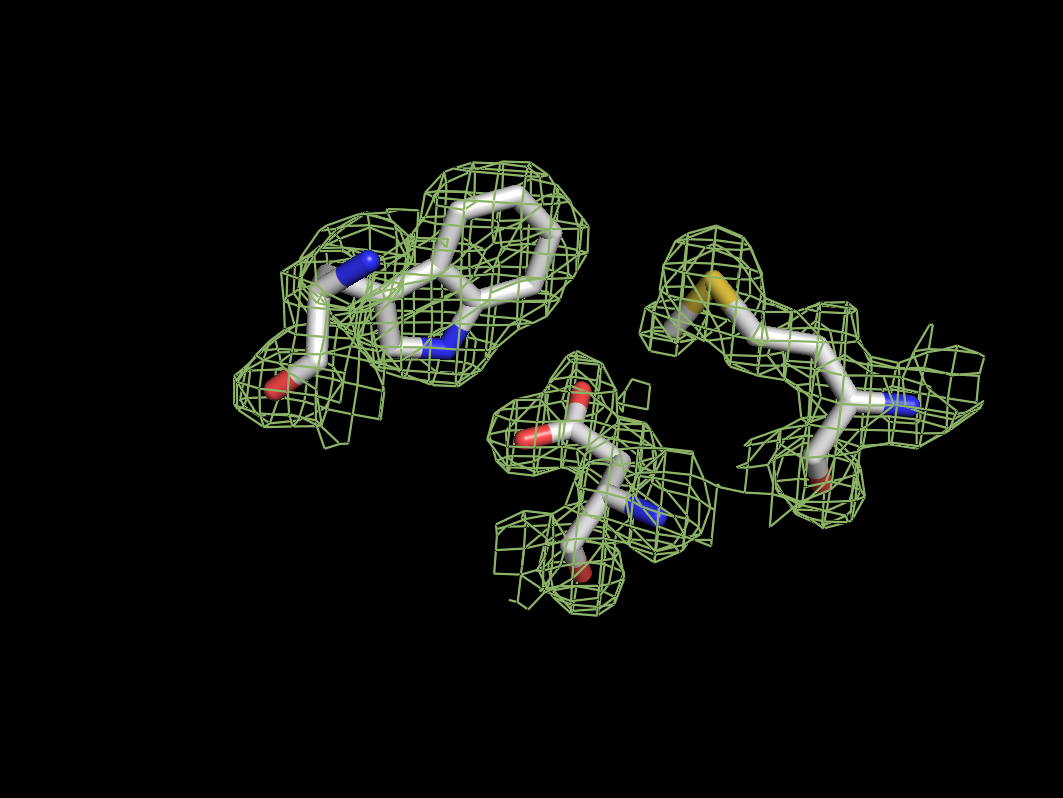
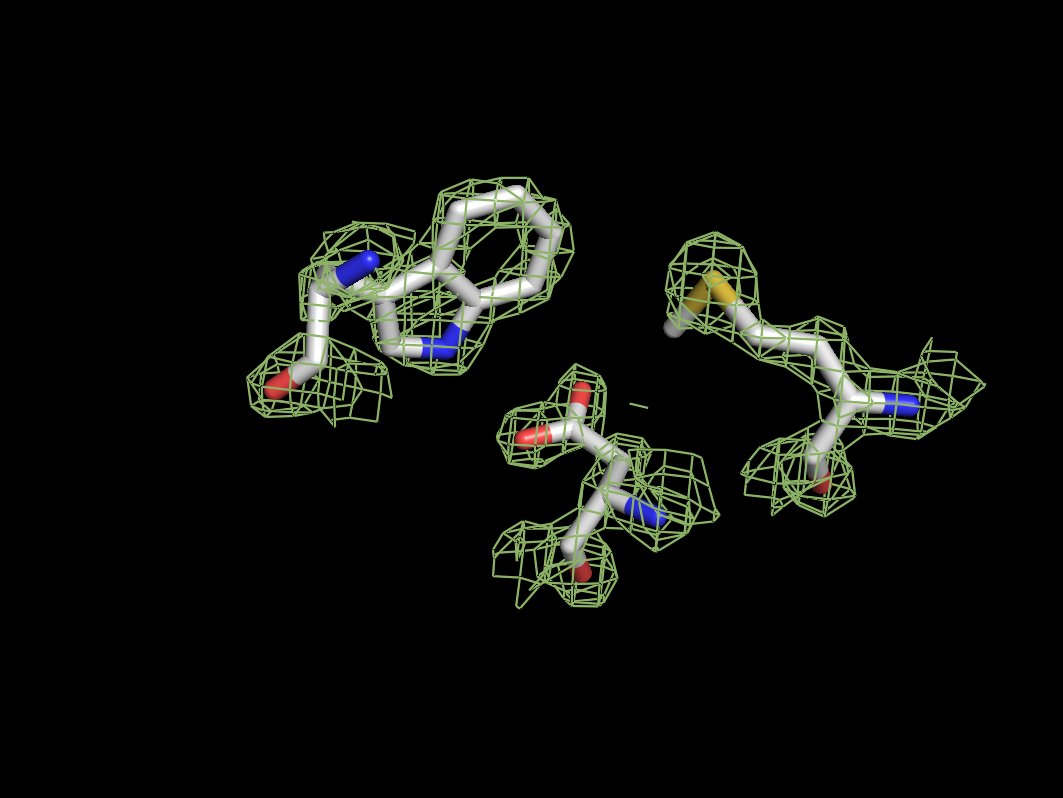
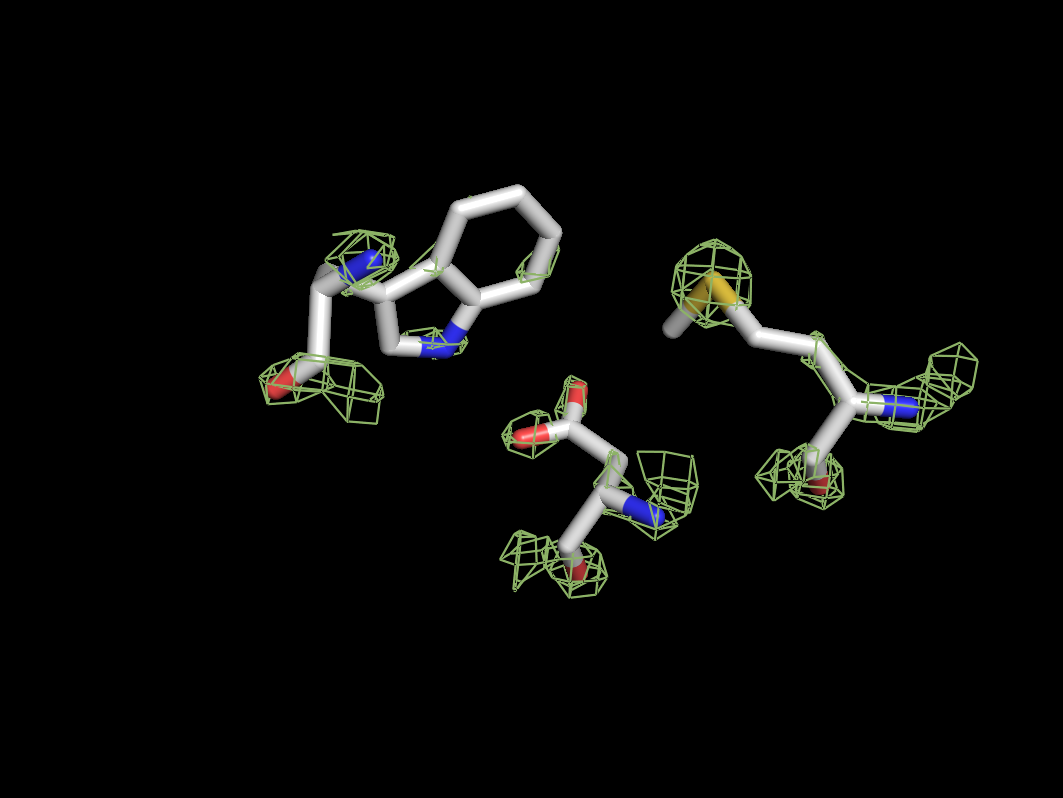
На низких уровнях подрезки электронная плотность хорошо покрывает аминокислотные остатки, и по ней можно довольно легко определить форму и тип аминокислоты. С ростом уровня подрезки электронная плотность исчезает в первую очередь с С-атомов боковых групп, концентрируясь на более электроотрицательных атомах кислорода, азота и серы.
Назад к странице семестров