Анализ ЯМР-файла
Задание. Сравнение трех водородных связей в структурах белка, расшифорованных с помощью ЯМР и РСА.
В качестве объекта исследования я выбрал структуры циклофилина А из E. coli: полученную в результате ЯМР 1OCA (20 структур) и полученную в результате РСА: 1W8L (разрешение 1.8Å). Получим пространственное выравнивание РСА-структуры и всех состояний ЯМР-структуры с помощью следующих команд:
align 1oca, 1w8l split_states 1oca set cartoon_transparency, 0.7, 1oca*Структуры довольно хорошо выравниваются (см. рис.1).
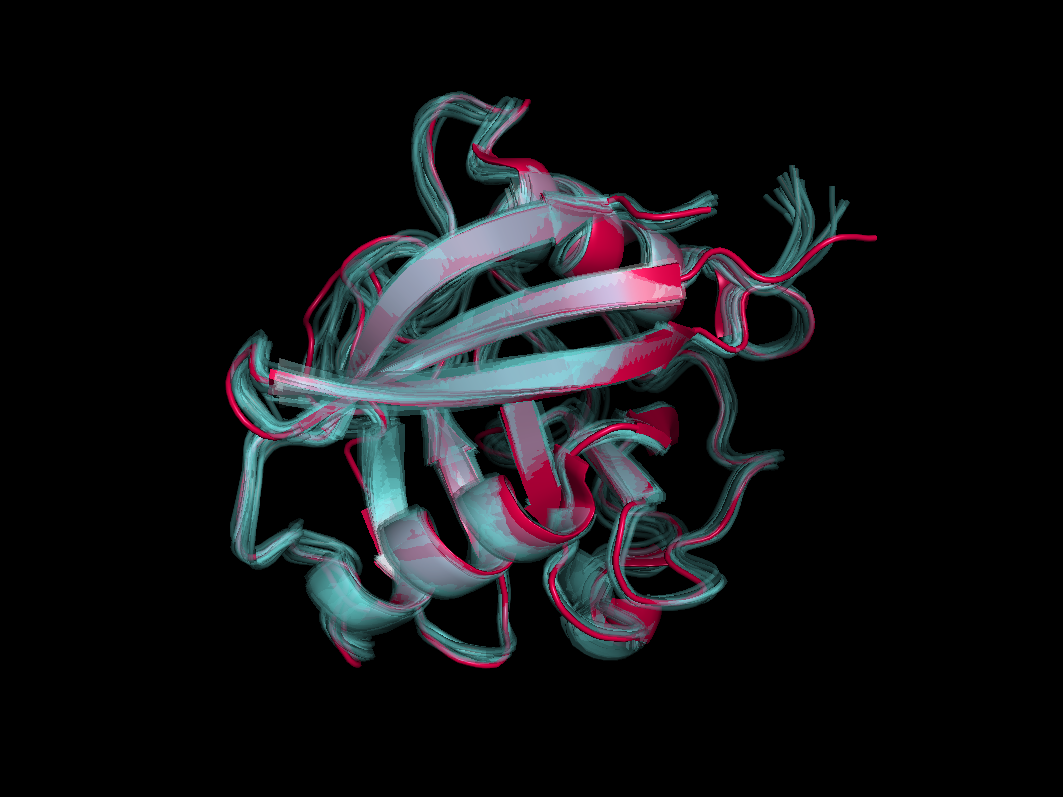
Рис.1. Пространственное выравнивание структуры 1w8l и всех состояний 1oca.
Для анализа были выбраны следующие связи:
- Остовная в β-листе: ASP9'N - ASP160'O;
- В петле на поверхности белка: CYS115'O - GLY96'N;
- С участием боковых цепей: ASP66'OD2 - GLY72'N;
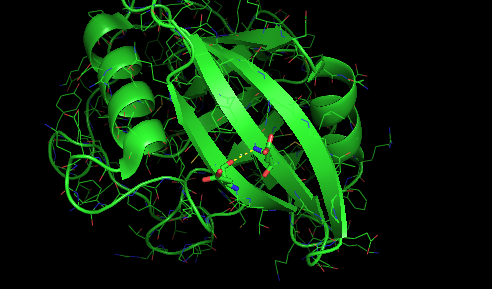 Рис.2а. Остовная водородная связь в β-листе в РСА-структуре. |
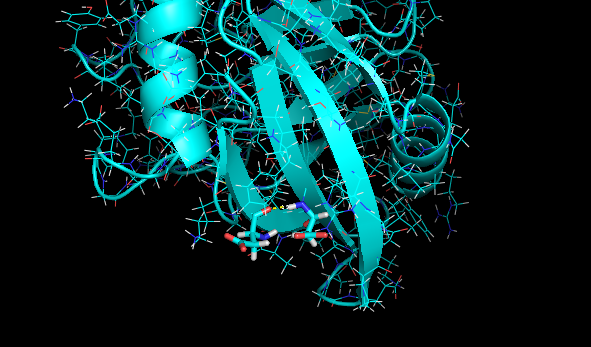 Рис.2б. Остовная водородная связь в β-листе в ЯМР-структуре. |
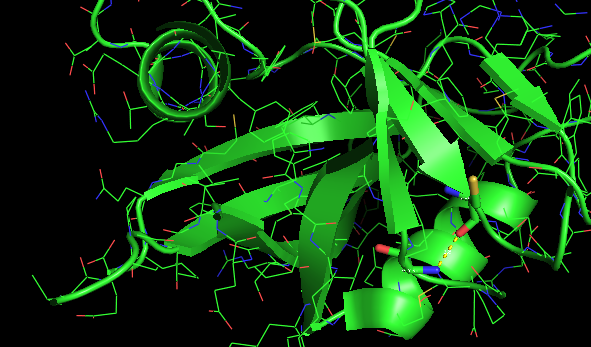 Рис.3а. Остовная водородная связь в петле на поверхности РСА-структуры. |
 Рис.3б. Остовная водородная связь в петле на поверхности ЯМР-структуры. |
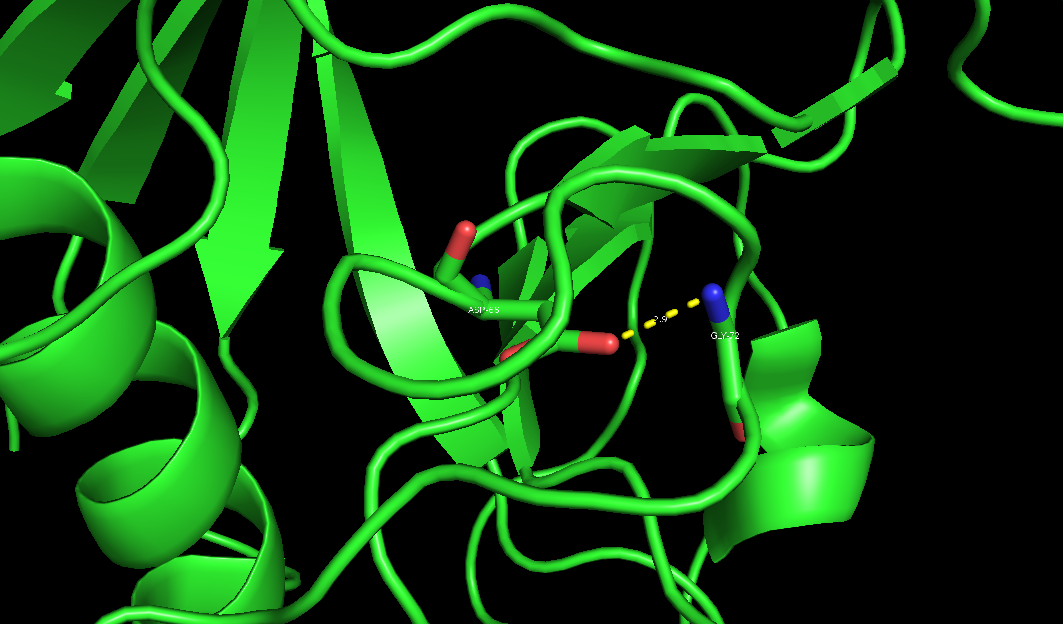 Рис.4а. Водородная связь с участием боковой цепи в РСА-структуре. |
 Рис.4б. Отсутствие водородной связи в ЯМР-структуре. |
Таблица 1. Данные о исследуемых водородных связях.
| Связь | Положение | Расстояние между атомами, Å | Встречаемость в моделях ЯМР, % | |||
| РСА | ЯМР | |||||
| Mинимум | Mедиана | Mаксимум | ||||
| ASP9'N - ASP160'O | Остовная в β-листе | 2.7 | 2.8 | 2.8 | 3.5 | 100 |
| CYS115'O - GLY96'N | Остовная в петле на поверхности белка | 2.8 | 2.7 | 2.8 | 2.8 | 100 |
| ASP66'OD2 - GLY72'N | С участием бокового радикала | 2.9 | 2.8 | 2.9 | 3.3 | 60 |
Выводы.
Различия между структурами, полученными при помощи РСА и ЯМР, скорее всего, обусловлены тем, что в растворе некоторые части белка более подвижны, из-за чего положения в пространстве некоторых остатков (И, следовательно, расстояния между ними) могут отличаться. Остовные связи элемента вторичной структуры (β-листа) и в поверхностной петле оказались относительно стабильными за исключением редких выбросов , тогда как поверхностная связь с участием бокового радикала оказалась куда менее стабильной, а в 8 из 20 структур вообще не встречается - остатки развернуты в разные стороны.
Дополнительно.
Pymol-команда для генерации изображений 2-4, пример:
hide everything, all select residues, 1w8l and (resi 66,72) show sticks, residues select O, xray and resi 66 and name od2 select N, xray and resi 72 and name N distance hbond, O, N, 3.5 show cartoon, 1w8l show lines, 1w8l and not residues
Назад к странице семестров