Пространственное выравнивание и совмещение структур
Задание 1. Совмещение структр гомологичных белков.
Для выполнения этого задания я решил взять цепь А бифункционального белка биосинтеза пуринов ATIC (он же фосфорибозиламиноимидазолкарбоксамид
формилтрансфераза инозинмонофосфат циклогидролаза), который я использовал в практикуме по определению гидрофобных ядер. PDB-id структуры
4ehi. При помощи сервиса PDBeFold были найдены структурные гомологи белка, из которых
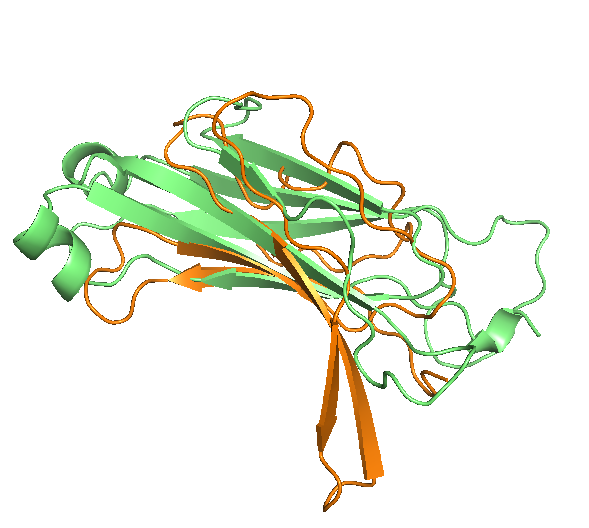
я отобрал 3 (см. рис. 1) - два бактериальных и один птичий. Далее было построено множественное выравнивание отобранных гомологов с исходным белком (см. рис. 2).

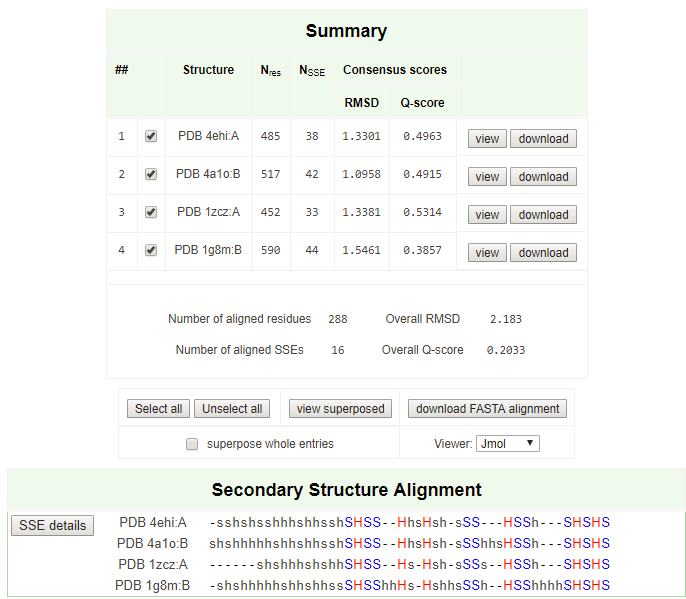
Для скачивания были доступны структурное выравнивание в формате fasta и PDB-файл с пространственным выравниванием. На рисунке 3 представлен внешний вид пространственного выравнивания. Как и следовало ожидать, три бактериальных белка выровнялись между собой относительно хорошо (хотя некоторые петли и даже элементы вторичной структуры всё равно выбиваются), а вот птичий белок выровнялся с остальными только частью своего формилтрансферазного домена и бета-листовой структурой, связывающей этот домен с циклогидролазным.
Сравним структурное выравнивание, полученное с помощью PDBeFold, и множественное выравнивание последовательностей, полученное с помощью программы Muscle. Выранивания, открытыe в JalView и покрашенные ClustalW, воспроизведены на рисунках 4 и 5, соответственно.


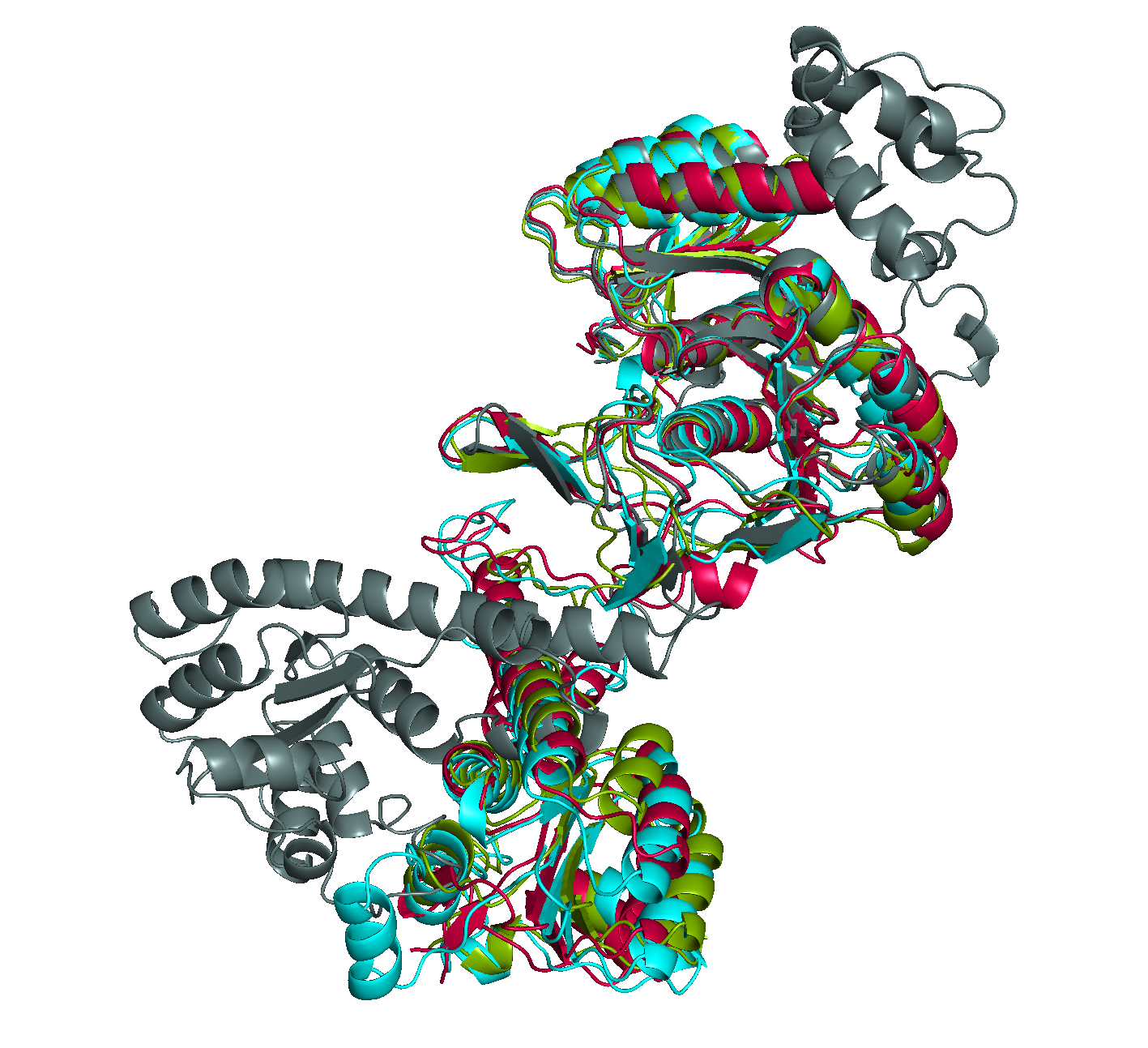
Выравнивания действительно очень сильно различаются. Они практически идентичны в области С-концевого формилтрансферазного домена (что подтверждается выравниванием структур - как консервативные блоки, так и большой участок, в котором конструкция из 4 альфа-спиралей птичьего белка не совпадает ни с одним бактериальным), однако по приближению к N-концу полученное с помощью muscle выравнивание имеет примерно однородное распределение гэпов и консервативных участков, и на нём не наблюдается громадного участка несовпадения циклогидролазного домена птичьего белка со всеми остальными, который отражен на структурном выравнивании; также последнее имеет участок несовпадения N-конца структуры 4ehi:А со всеми остальными, что тоже можно заметить по пространственному выравниванию (см. рис. 6), хоть оно и не так заметно, как рассмотренное ранее несовпадение (весь этот участок отмечен в выравнивании маленькими буквами, то есть он не считается выровненным, но начало цепи 4ehi:А выбивается из общего ряда слишком явно). Таким образом, можно сделать вывод, что структурное выранивание оказалось точнее.
Задание 2. Построение совмещения по заданному выравниванию
Для выполнения этого задания я выбрал домен α-цепи d:115-204 из структуры
2bnq и домен β-цепи e:114-242 из структуры 2bnr. Домены
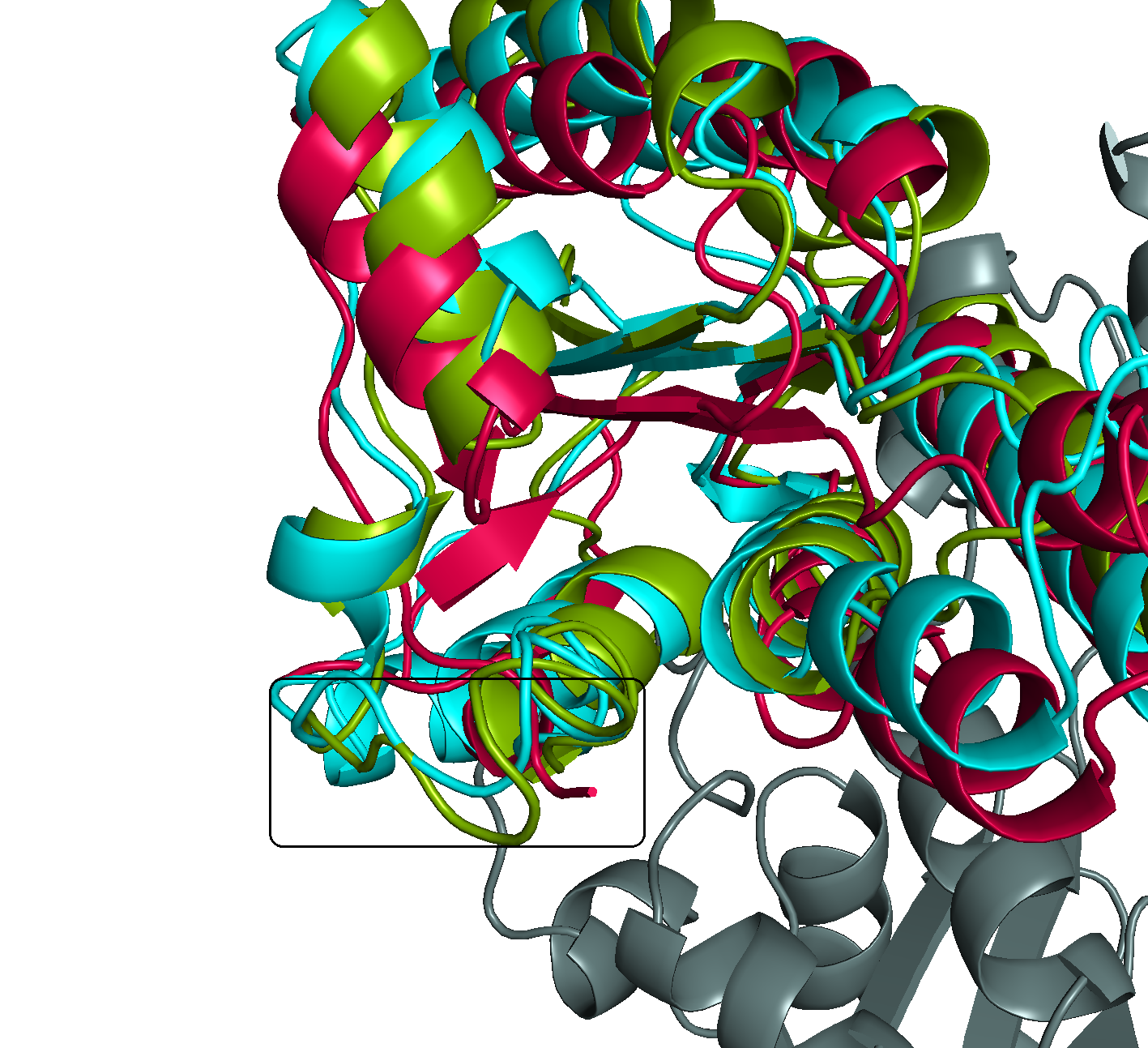
были вырезаны из структур и сохранены в отдельные PDB-файлы. При совмещении доменов видно, что выравниваются они не очень хорошо (см. рис. 7):
RMS полученного пространственного выравнивания 4.441. Попробуем немного улучшить его. При помощи сервиса Sheep для доменов были получены карты β-листов (рис.8):

Видно, что обе карты β домена не соответствуют таковой у α домена; тем не менее, я решил, что больше схож нижний лист; с ним и будем пытаться выровнять α-домен. Для этого выберем консервативные цистеины из обоих листов (137 из α и 143 из β) и их ближайших консервативных соседей и попробуем совместить структуры по ним:
select set1, alpha and resi 124-126+136-138+177-179 and name ca select set2, beta and resi 124-126+142-144+189-191 and name ca pair_fit set1, set2В результате команды по совмещению двух групп атомов RMS между ними составило 0.518 ("затравки" выравнивания совместились очень хорошо), однако для остальных частей доменов картина осталась удручающей: домены не совершенно не выровнялись друг относительно друг друга (см. рис. 9). Это можно объяснить тем, что длины доменов в заявленных границах различаются почти на 30%, и в α домене отсутствуют несколько бета-тяжей, образующих в β - домене дополнительный лист. Файл с совмещением можно скачать здесь.
Назад к странице семестров