Работа с Pymol
В этом практикуме мы будем осваивать возможности PyMol как пакета для моделирования. Начнем с простого: загрузим структуру белка 1LMP и попробуем изменить её при помощи Sculpting:
In [1]:
import __main__
__main__.pymol_argv = [ 'pymol', '-x' ]
import pymol
pymol.finish_launching()
from pymol import cmd, stored
In [2]:
cmd.do('''
fetch 1lmp, async=0
remove solvent
''')
In [3]:
cmd.png(filename='pic1.png',width='8cm',dpi=300, ray=1)
In [4]:
cmd.png(filename='pic2.png',width='8cm',dpi=300, ray=1)
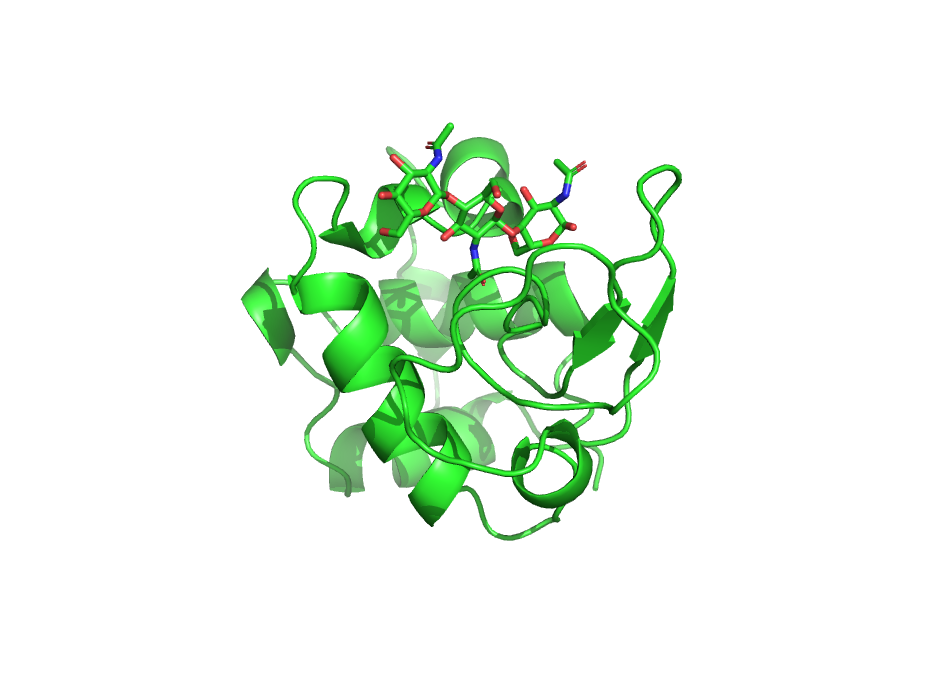 |
 |
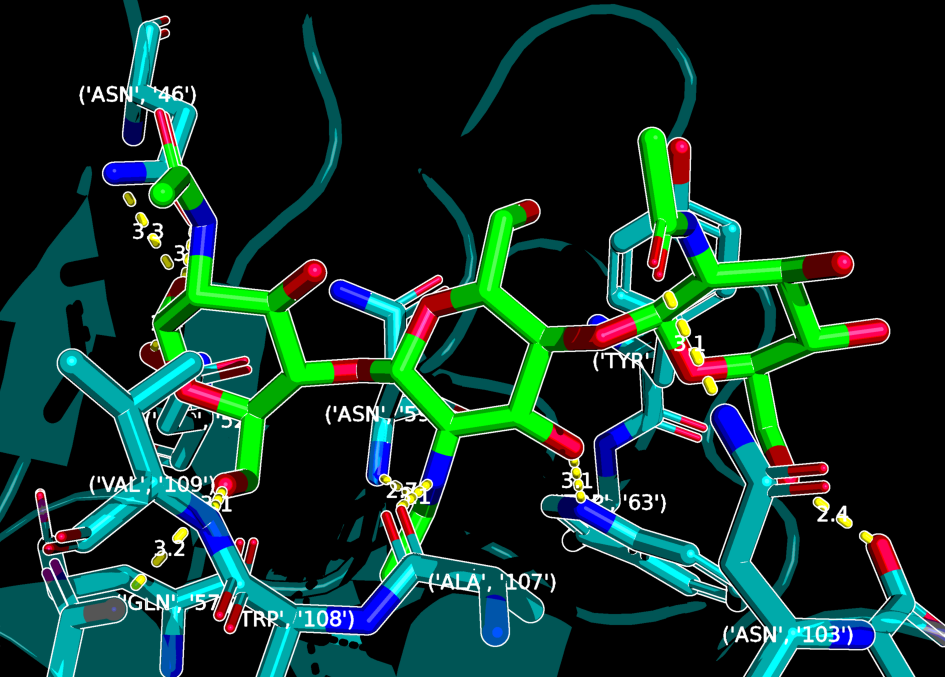
Теперь найдем остаток, принимающий участие в связывании лиганда, и проведем точечный мутагенез, ведущий к потере этого взаимодействия.
In [3]:
cmd.reinitialize()
cmd.set('ray_opaque_background', 1)
cmd.bg_color(color="black")
In [4]:
cmd.do('''
set antialias, 2
set ray_trace_mode, 3
fetch 1lmp, async=0
remove solvent
extract ligand, het
as cartoon, 1lmp
set cartoon_transparency, 0.5
as sticks, ligand
center ligand
origin ligand
zoom ligand
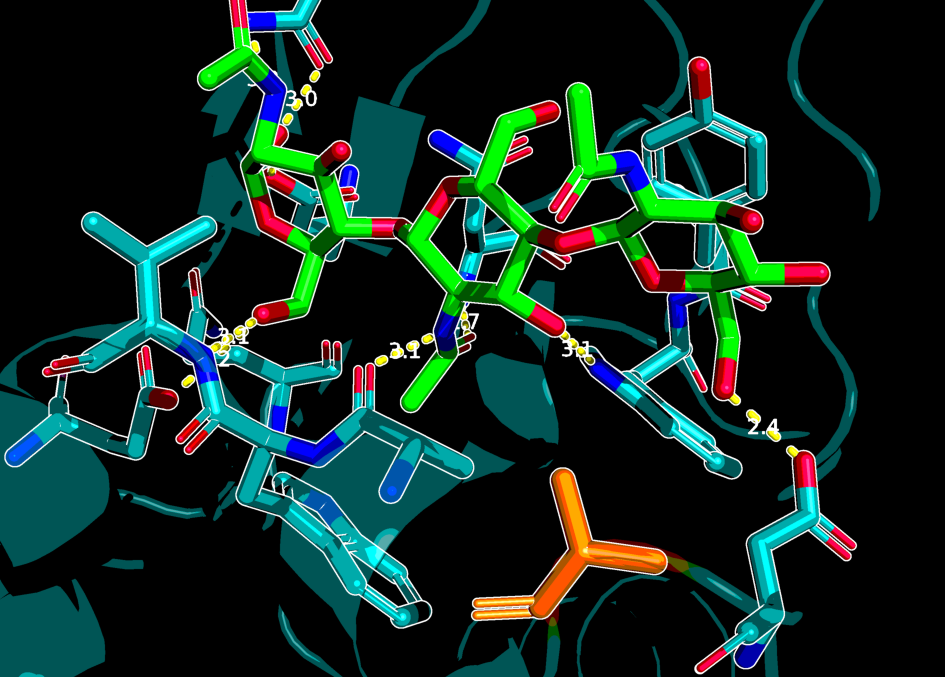
select residues, byres /1lmp//A and (ligand around 3.5)
show sticks, residues
''')
In [5]:
cmd.distance('hbonds', '(/1lmp//A)', 'ligand', '3.5', '2')
cmd.do('''
set label_color, white
label residues and name ca, resn,resi
color green, ligand
color teal, 1lmp
color seaborgium, (/1lmp//A or ligand) and name o*
color blue, (/1lmp//A or ligand) and name n*
''')
In [ ]:
cmd.png(filename='pic3.png',width='8cm',dpi=300, ray=1)
Скорее всего, в связывании лиганда точно принимает участие остаток ASN103. Проведем мутацию, заменив его на аланин:
In [6]:
from pymol import wizard
In [7]:
cmd.hide('labels')
cmd.do('''
delete hbonds
delete residues
''')
In [8]:
cmd.wizard("mutagenesis")
cmd.do("refresh_wizard")
In [9]:
cmd.get_wizard().set_mode("ALA")
cmd.get_wizard().do_select("/1lmp//A/103")
# Select the first rotamer, which is most probable
cmd.frame(1)
# Apply the mutation
cmd.get_wizard().apply()
# Close wizard
cmd.set_wizard("done")
In [10]:
cmd.color("tv_orange", '/1lmp//A/103')
cmd.distance('hbonds', '(/1lmp//A)', 'ligand', '3.5', '2')
Out[10]:
In [ ]:
cmd.png(filename='pic4.png',width='8cm',dpi=300, ray=1)
Теперь загрузим неизмененный белок и совместим две структуры:
In [11]:
cmd.do('''
delete hbonds
delete ligand
hide sticks, /1lmp//A/ and not resi 103
fetch 1lmp, init
as cartoon, init
''')
In [12]:
cmd.remove('solvent')
In [13]:
cmd.do('''
extract ligands, het
color green, ligands
color seaborgium, ligands and name o*
color blue, ligands and name n*
show sticks, ligands
color limon, init
show sticks, /init//A/ and resi 103
color magenta, /init//A/ and resi 103
''')
In [14]:
cmd.zoom()
In [15]:
cmd.do('''
set matrix_mode, 1
set movie_panel, 1
set cache_frames, 1
mset 1-190
frame 1
label resi 103, resn,resi
translate [30,30,30], object=init
mview store
mview store, object=init
mview store, object=1lmp
frame 30
translate [-30,-30,-30], object=init
mview store
mview store, object=init
mview store, object=1lmp
mview interpolate, object=init
frame 60
orient ligands
mview store
mview store, object=init
mview store, object=1lmp
mview reinterpolate, object=init
mview reinterpolate, object=1lmp
frame 90
orient resi 103
mview store
mview store, object=init
mview store, object=1lmp
frame 120
orient resi 103
mview store
mview store, object=init
mview store, object=1lmp
frame 150
orient resi 103
mview store
mview store, object=init
mview store, object=1lmp
frame 180
mview reinterpolate, object=init
mview reinterpolate, object=1lmp
frame 1
mplay
''')
In [16]:
import io
import base64
from IPython.display import HTML
In [17]:
video = io.open('movie.mp4', 'r+b').read()
encoded = base64.b64encode(video)
HTML(data='''<video alt="test" controls>
<source src="data:video/mp4;base64,{0}" type="video/mp4" />
</video>'''.format(encoded.decode('ascii')))
Out[17]:
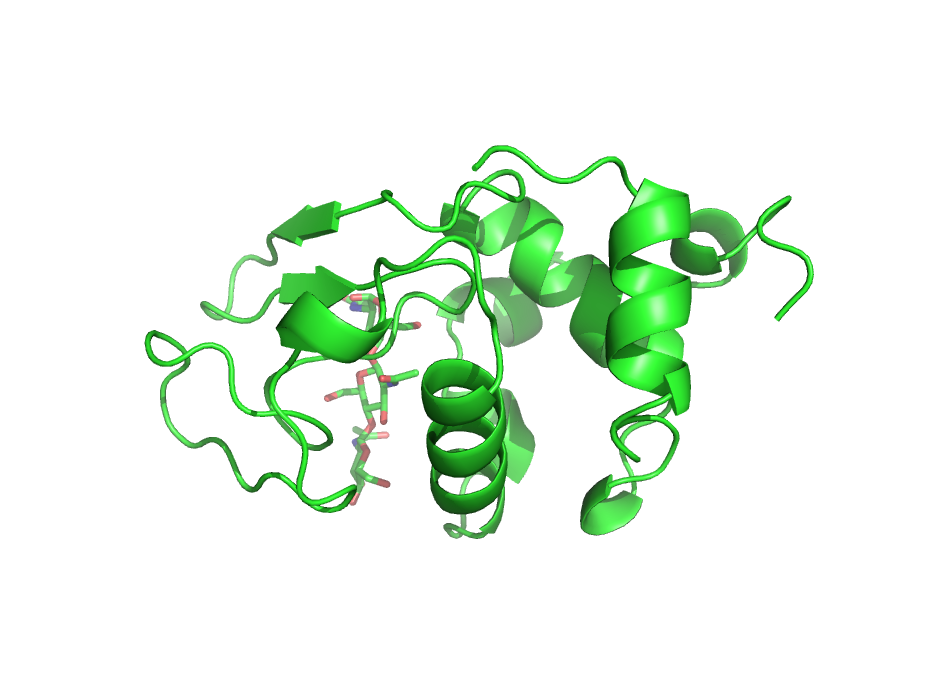
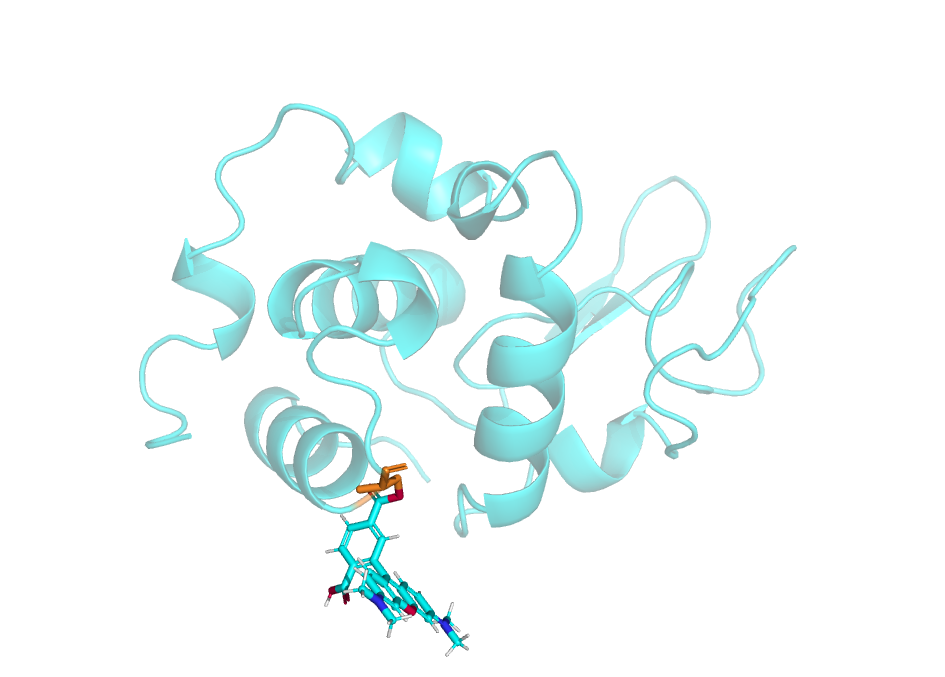
Присоединим к белку флуоресцентную метку TAMRA:
Присоединять будем к аминокислоте, имеющей -ОН группу, например к серину, чтобы образовать сложноэфирную связь. Информацию о присоединении данной метки к белкам можно найти, например, здесь.
In [18]:
cmd.reinitialize()
In [19]:
cmd.load('tamra.sdf')
cmd.remove('tamra and id 4')
cmd.select('at', 'id 32')
Out[19]:
In [20]:
cmd.do('''fetch 1lmp, async=0''')
cmd.remove('solvent')
cmd.select('ser', '/1lmp//A/SER`15')
cmd.hide('sticks', '1lmp')
cmd.show('cartoon', '1lmp')
cmd.show('sticks', 'ser')
cmd.color('tv_orange', 'ser')
cmd.select('og','/1lmp//A/SER`15/OG')
cmd.color('seaborgium', 'og')
cmd.set('cartoon_transparency','0.5')
In [21]:
cmd.fuse('at', 'og', 1)
cmd.torsion(-40.0)
In [22]:
cmd.zoom('1lmp')
cmd.turn("y", "80.0")
cmd.unpick()
In [30]:
cmd.png(filename='pic5.png',width='8cm',dpi=300, ray=1)
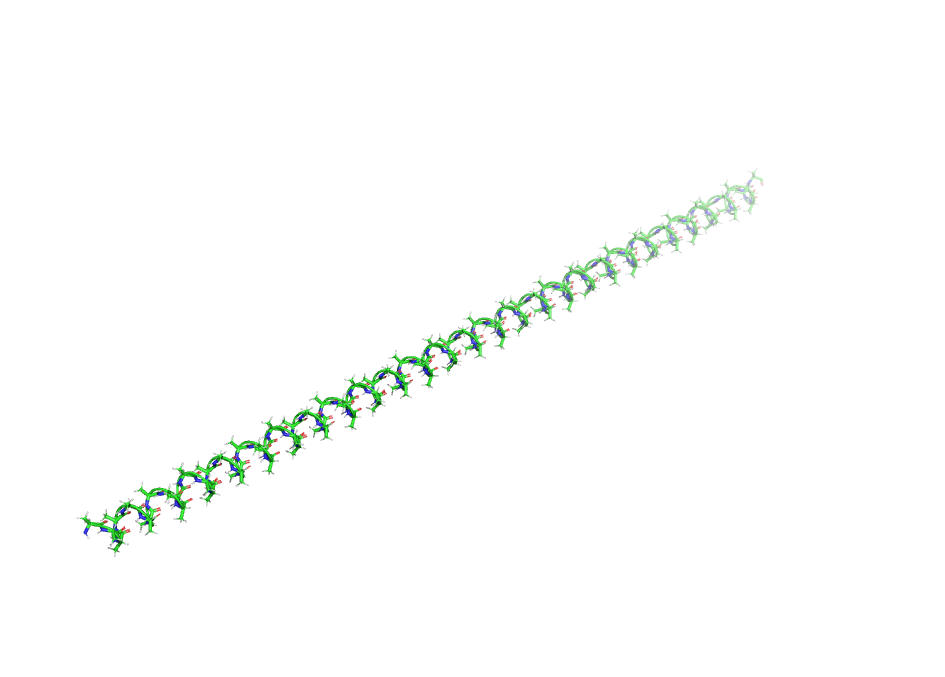
Построим полиаланиновую альфа-спираль длиной 100 аминокислот:
In [23]:
cmd.reinitialize()
phi = -60
psi = -45
cmd.fragment('ala')
acid = 'ala'
for i in range (2,101):
cmd.edit("i. %i & n. C" % i)
cmd.do('editor.attach_amino_acid("pk1", "%s")' % acid)
cmd.set_dihedral("i. %i & n. N" % i, "i. %i & n. CA" % i,
"i. %i & n. C" % i, "i. %i & n. N" % (i+1),
phi)
cmd.set_dihedral("i. %i & n. C" % i, "i. %i & n. N" % (i+1),
"i. %i & n. CA" % (i+1), "i. %i & n. C" % (i+1),
psi)
In [24]:
cmd.zoom()
cmd.unpick()
In [70]:
cmd.png(filename='pic6.png',width='8cm',dpi=300, ray=1)