Докинг низкомолекулярных лигандов в структуру белка
В этом задании мы будем проводить докинг лигандов в структуру белка, которая была сможелирована на прошлом практикуме. Докинг будем проводить при помощи Autodock Vina и oddt.
import copy
import numpy as np
# Отображение структур
import ipywidgets
import IPython.display
from IPython.display import display,display_svg,SVG,Image
# Open Drug Discovery Toolkit
import oddt
import oddt.docking
import oddt.interactions
# Органика
from rdkit.Chem import Draw
from rdkit.Chem import AllChem
from rdkit.Chem import MolFromSmiles
from rdkit.Chem.Draw import IPythonConsole
# Модуль для манипулирования pdb
import pmx
# из созданных на прошлом практикуме структур возьмем ту,
# у которой было наивысшее значение скор-функции - пятую.
pdb=pmx.Model('HYACE5.pdb')
Разделим белок и лиганд на разные структуры:
# посмотрим остатки и локализуем лиганд:
for r in pdb.residues[135:]:
print r
# создадим новые объекты:
# только белок и только лиганд
newpdb = pdb.copy()
for r in newpdb.residues[-3:]:
newpdb.remove_residue(r)
lig = pdb.copy()
for r in lig.residues[:-3]:
lig.remove_residue(r)
Теперь найдем геометрический центр лиганда:
coords = np.zeros((len(lig.atoms),3))
for c,a in enumerate(lig.atoms):
coords[c,:] = a.x
geom_center = np.mean(coords, axis = 0)
geom_center
newpdb.writePDB('prot.pdb')
lig.writePDB('lig.pdb')
Подготовка белка для докинга
prot = oddt.toolkit.readfile('pdb','prot.pdb').next()
print prot.OBMol.AddPolarHydrogens()
print prot.OBMol.AutomaticPartialCharge()
print 'is it the first mol in 1lmp is protein?',prot.protein,':) and MW of this mol is:', prot.molwt
Забавно, что белок не был распознан.
Лиганды для докинга
Создадим несколько лигандов на основе NAG и придумаем какие-нибудь еще для разнообразия:
smiles = ['OC1C(C(C(OC1O)CO)O)O', '[NH3+]C1C(C(C(OC1O)CO)O)O',
'C1C(C(C(OC1O)CO)O)O', 'C1=CC=C(C=C1)C1C(C(C(OC1O)CO)O)O',
'c1(O)cc(c2cc(F)cc(I)c2)cc(O)c1',
'C1=C(C=C(C(=C1[N+](=O)[O-])[O-])[N+](=O)[O-])[N+](=O)[O-]',
'C1=CC(=CC=C1O)OC2=CSC=C2', 'COP(=O)(O)OC1=CC=C(C=C1)C=O']
Отличные вещества! Теперь посмотрим на них и подготовим их структуры к докингу:
mols = []
images =[]
for s in smiles:
m = oddt.toolkit.readstring('smi', s)
if not m.OBMol.Has3D():
m.make3D(forcefield='mmff94', steps=150)
m.removeh()
m.OBMol.AddPolarHydrogens()
mols.append(m)
### with print m.OBMol.Has3D() was found that:
### deep copy needed to keep 3D, write svg make mols flat
images.append((SVG(copy.deepcopy(m).write('svg'))))
display_svg(*images)
Докинг
# create docking object
# центром докинга дадим геометрический центр лиганда,
# найденный нами ранее - приблизительное положение сайта связывания
dock_obj= oddt.docking.AutodockVina.autodock_vina(
protein=prot,size=(20,20,20),center=geom_center,
executable='/usr/bin/vina',autocleanup=True, num_modes=20)
print dock_obj.tmp_dir
print " ".join(dock_obj.params)
# do it
res = dock_obj.dock(mols,prot)
Результаты докинга
import pandas as pd
# отсортируем результаты по аффинности (параметр vina_affinity):
res_sorted = sorted(res, key = lambda x : float(x.data['vina_affinity']))
hbtot = []
hbstr = []
phob = []
for i,r in enumerate(res_sorted):
int1, int2, strict = oddt.interactions.hbonds(prot,r)
hbtot.append(len(int1))
hbstr.append(strict.sum())
ph1, ph2 = oddt.interactions.hydrophobic_contacts(prot,r)
phob.append(len(ph1))
dat = pd.DataFrame({'Formula':[r.formula for r in res_sorted],
'Affinity':[r.data['vina_affinity'] for r in res_sorted],
'RMSD':[r.data['vina_rmsd_ub'] for r in res_sorted],
'HB_tot':hbtot,'HB_strict':hbstr, 'Hydphobic':phob})
dat[:]
Анализ докинга
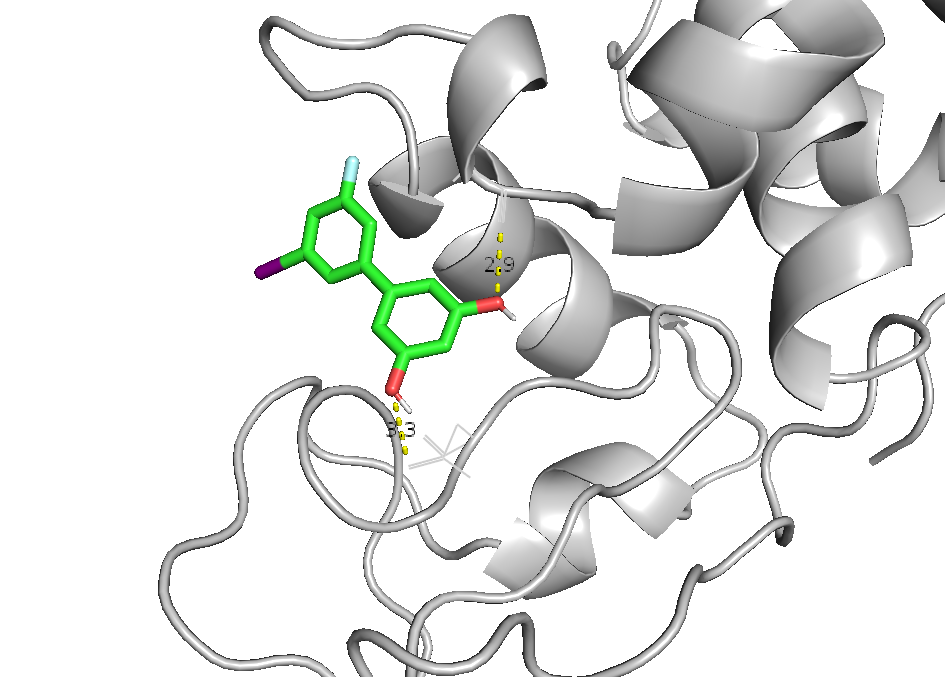
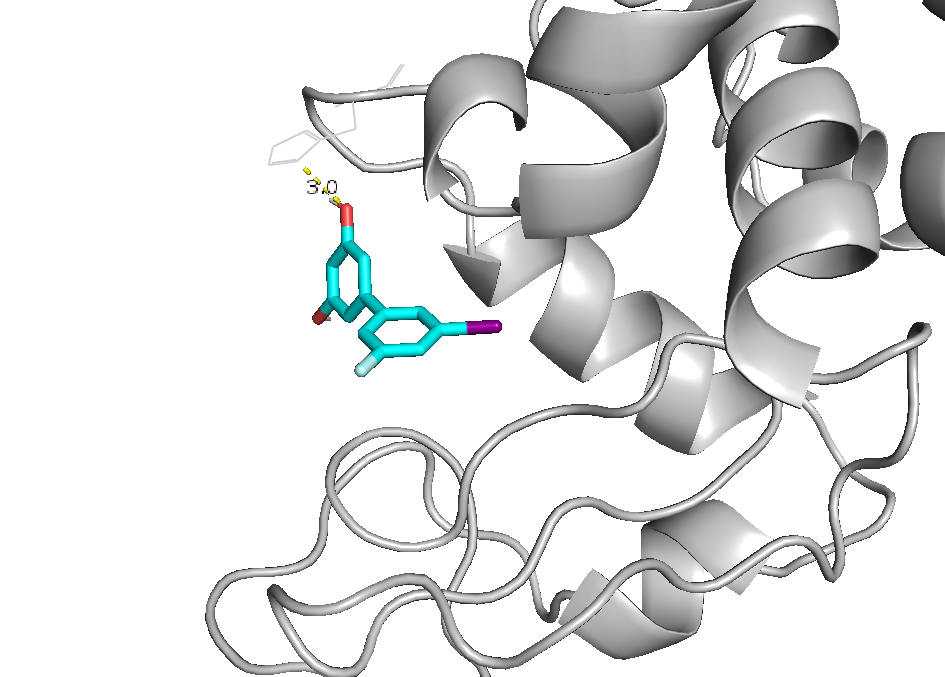
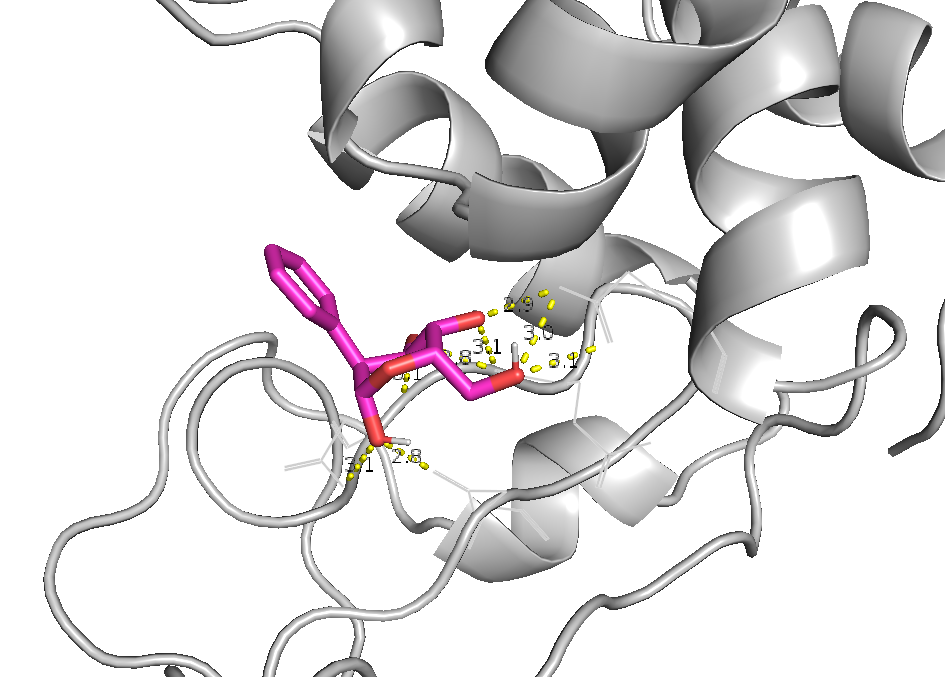
Забавно, что первые позиции заняла не модификация NAG, а придуманное мною галогенпроизводное. Рассмотрим поближе его структуры 0 (лучшая по RMSD), 3 (4 водородные связи, и все - строгие) а также лучшее из производных NAG - номер 9.
for i in (0, 4, 9):
res_sorted[i].write(filename='%s.pdb' % str(i), format='pdb')
Интересно, откуда у галогенпроизводного взялось аж 4 водородных связи учитывая что ОН групп у негов всего две. У производного NAG их тоже многовато.
def print_hbonds(res, i):
int1, int2, strict = oddt.interactions.hbonds(prot, res[i])
for x,y in zip(int1, int2):
print "%s:%s-->%s:%s" % (x[8] or 'PROT', x[5], y[9] or "LIG", y[5])
print_hbonds(res_sorted, 3)
print_hbonds(res_sorted, 9)
Видно, что многие связи дублируют друг друга; Также довольно забавно наблюдать, как в водородные связи попала связь между ароматическим азотом и фтором.
 |
 |
 |