Протонирование
PROPKA
Дан белок (PDB ID 5MGI), структура которого была получена при pH=4. Воспользовавшись
инструментом PROPKA, получаем модель с предсказанными протонами. Рассмотрим .log файл.
Наиболее примечательные строки оттуда:
Group pKa model-pKa ligand atom-type
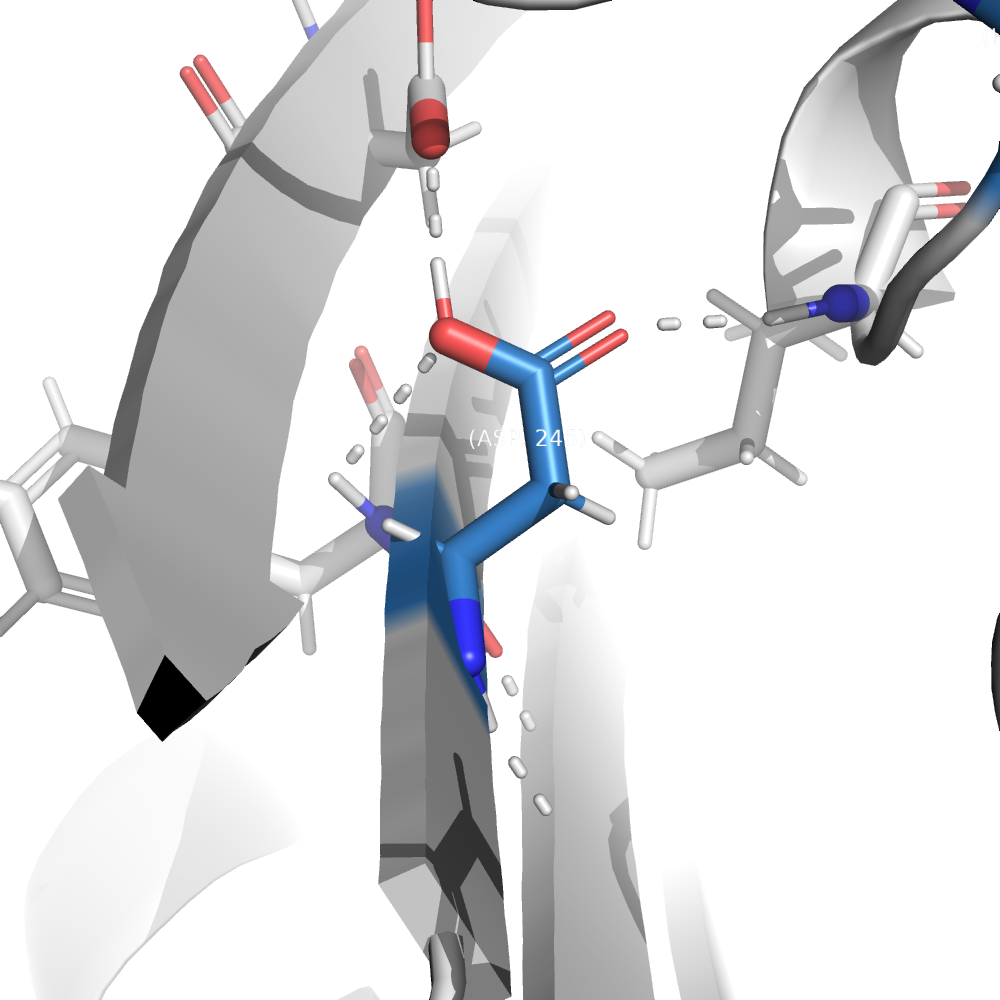
ASP 246 A 11.31 3.80
GLU 31 A 4.71 4.50
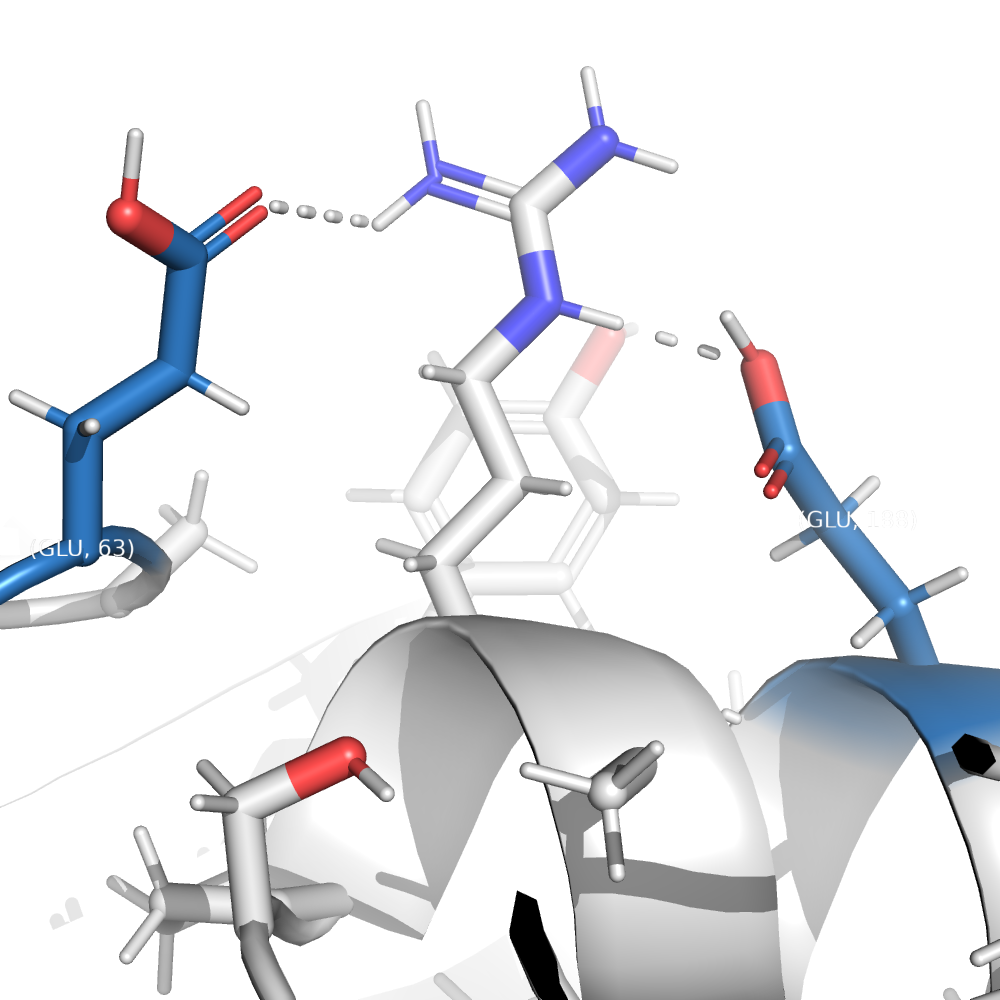
GLU 63 A 4.41 4.50
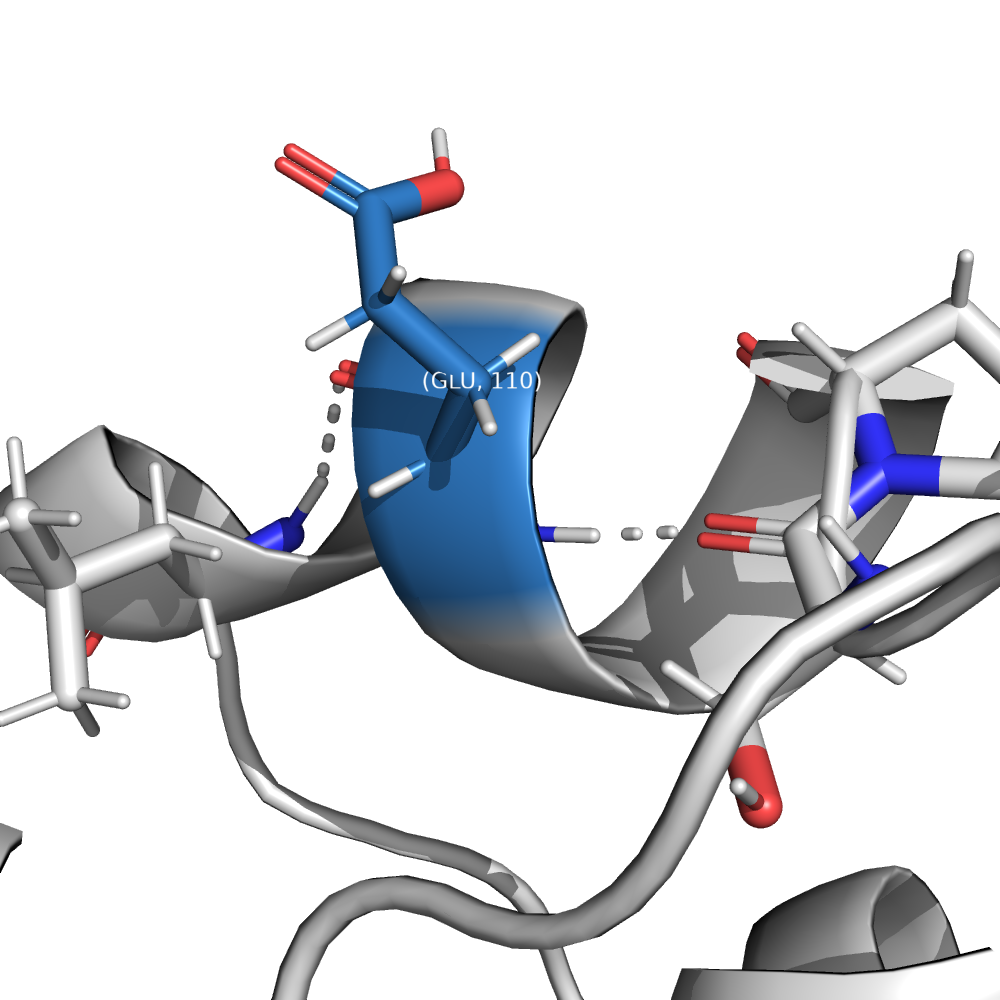
GLU 110 A 4.70 4.50
GLU 141 A 4.80 4.50
GLU 168 A 4.17 4.50
GLU 188 A 4.57 4.50
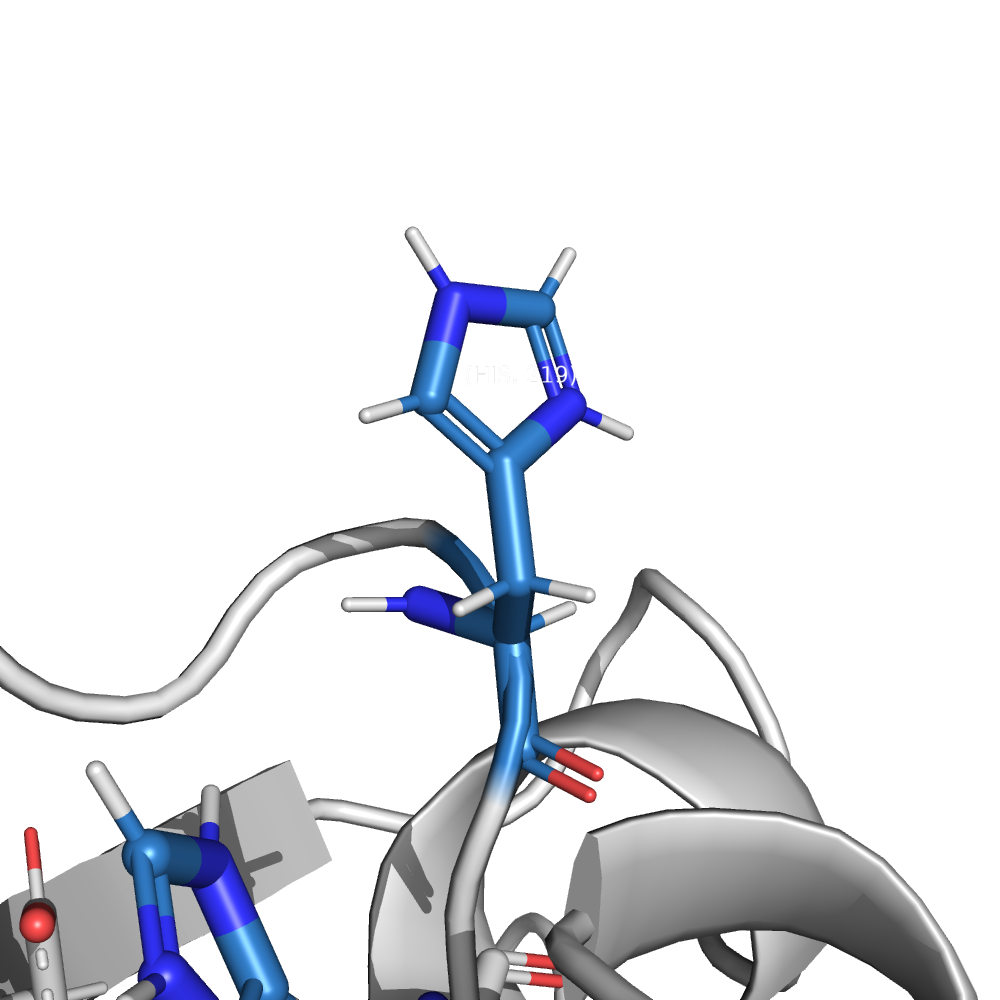
HIS 219 A 6.53 6.50
HIS 274 A 5.29 6.50
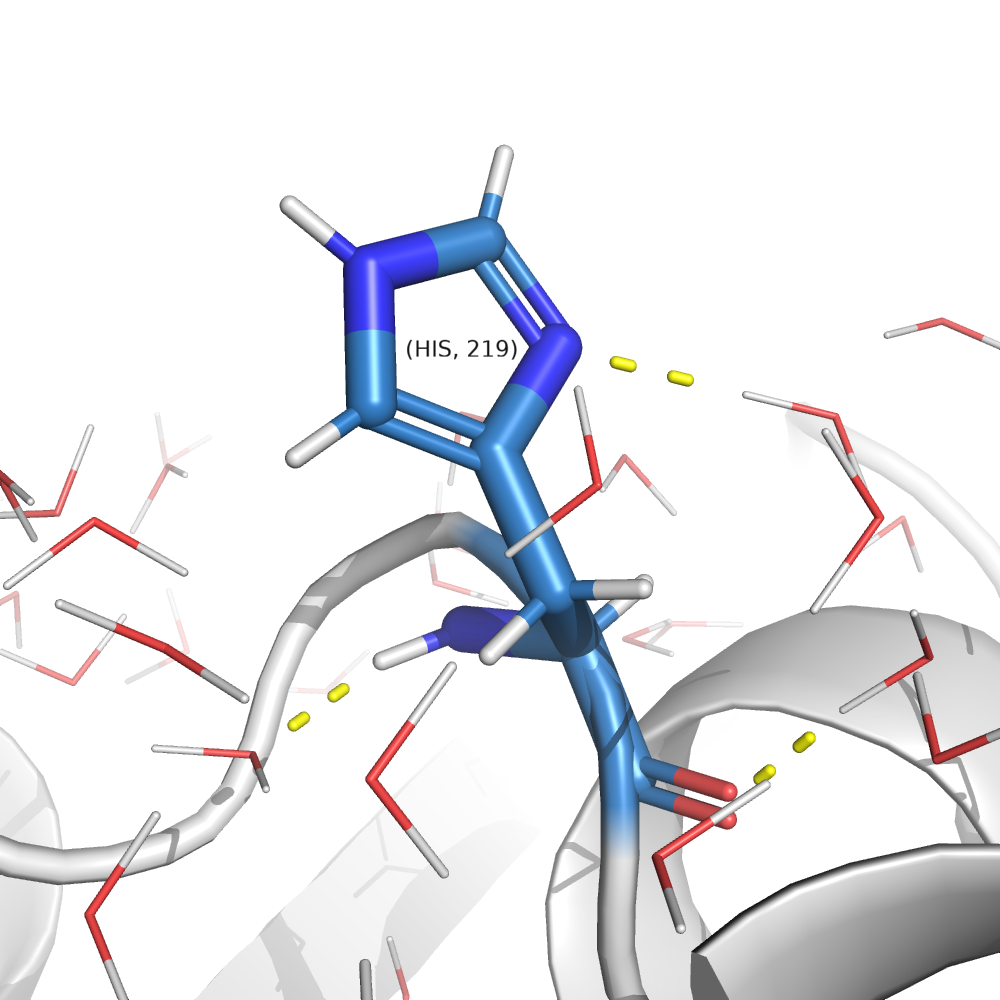
В случае гистидинов вариантов, как протонировать, нет, в отличие от глутаматов и аспартатов (рис. 1, A). В ряде случаев понять, как именно протонировать, не получится (см. рис. 1, Б). Но если протонируемая группа образует водородную связь, можно что-то понять (см. рис. 1 В, Г).
Прошу прощения за тёмный фон, на белом просто, оказывается, белые надписи видно не очень хорошо.
PROTOSS
Рассчитали протонирование при помощи другого инструмента PROTOSS. Он при потонировании учитывает возможность образования водородных связей.
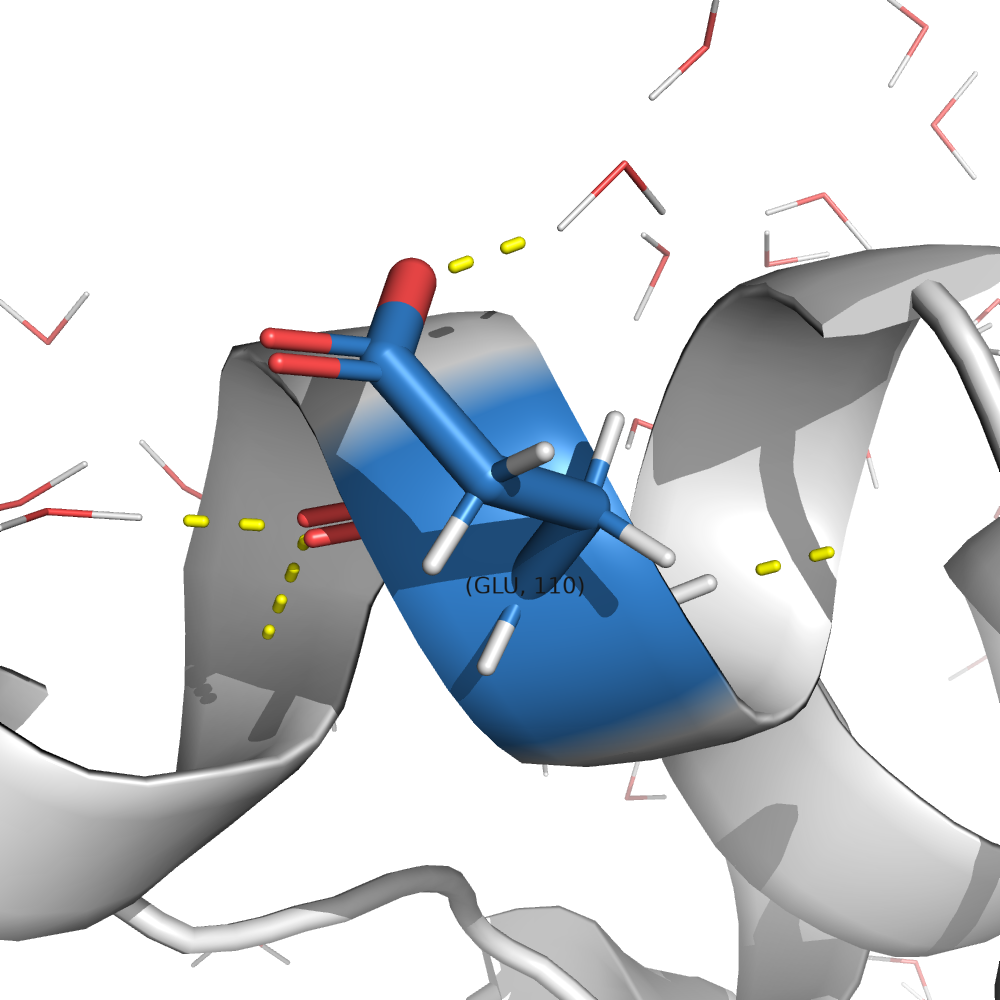
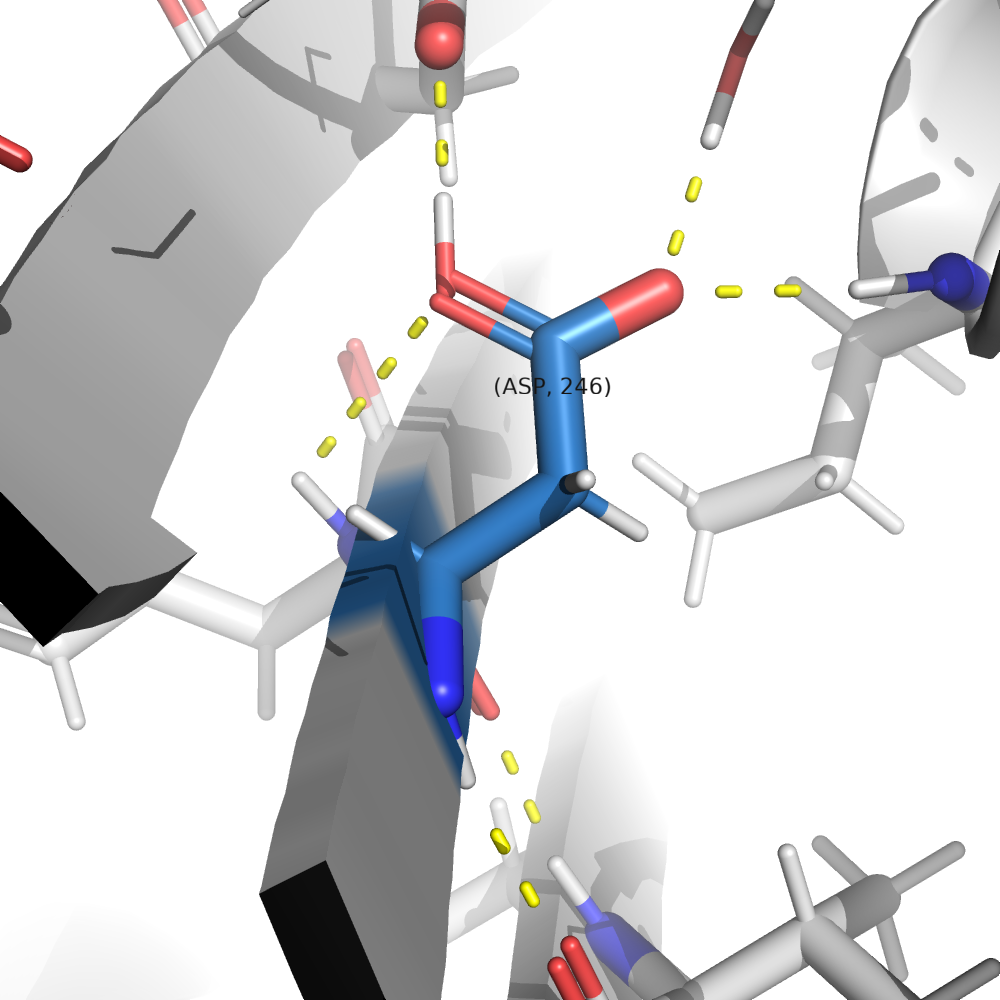

PROTOSS ничего не знает о том, при каком pH получена структура, и смотрит только на водородные связи. В результате этого видим протонированным только один остаток из рассмотренных выше пяти (ASP246). Видимо, программа считает по умолчанию, что остаток не протонирован, и только в том случае, когда это даёт выигрыш в количестве водородных связей, протонирует остаток. Можно заметить, что у остатка ASP246 как раз и было какое-то просто аномально большое значение pKa в выдаче PROPKA (11.31), что обусловлено микроокружением.
Видимо, наиболее катастрофическими последствия будут при депротонировании именно ASP246. Скорее всего, самое неприятное при этом будет даже не потеря одной водородной связи, а появление нескомпенсированного отрицательного заряда внутри окружения с низкой диэлектрической проницаемостью приводит к увеличению энергии системы (если не вру, потому что с физикой сложно).