Мини-обзор Microbacterium sediminis
Данный мини-обзор рассматривает геном Microbacterium sediminis с помощью python и Google Sheets. Рассматриваются расстояния между cds и стандартные данные о геноме.
1 Введение
У микроорганизмов, выделенных из экстремальных сред, есть уникальные физиология и метаболизм, позволяющие адаптироваться к среде обитания, из-за чего экстремофилы очень разнообразны (Jebbar et al., 2015). Глубоководные обитатели особенно интересны, так как они подвергаются воздействию сложных условий среды, включающих крайне низкую температуру (в основном 2 ◦C-3 ◦C), высокое давление (в среднем 38 МПа), отсутствие света и недостаток питательных веществ (Sanders and Hessler, 1969; Poli et al., 2017)
Microbacterium sediminis YLB-01T - бактерия, выделенная из образца осадка, собранного в юго-западной части Индийского океана (E/S, 49.8405° /37.8111°) на глубине 2327 метра во время рейса исследовательского судна Da-Yang Yi-Hao в ноябре 2008 года (Yu et al., 2013). Ее полный геном был секвенирован для выяснения молекулярных основ адаптации к низким температурам и высокому давлению.
M. sediminis - аэробный, грамположительный, палочковидный актиномицет и экстремофил (холодоустойчивый барофил), она может расти при температурах от 4°С до 50°С (оптимально 28°С), а также при давлении от 0.1 МПа до 80 Мпа. В геноме M. sediminis очень много генов вовлеченных в основной метаболизм. 249 генов связанных с метаболизмом аминокислот, охватывая пути деградации и/или синтеза 16 видов аминокислот, и 207 генов связанных с углеводным метаболизмом (Yi et al., 2020)
2 Материалы и методы
Данные по геному исследуемой бактерии были взяты с сайта Национального Центра Биотехнологической информации (NCBI):
- GCF_004564075.1_ASM456407v1
- GCF_004564075.1_ASM456407v1_cds_from_genomic.fna.gz
- GCF_004564075.1_ASM456407v1_genomic.fna.gz
Для анализа данных использовались электронные таблицы Google Sheets, и программы, написанные на языке Python и bash. Все графики, таблицы и рисунки получены с их помощью.
3 Результаты
3.1 Стандартные данные о геноме
Геном M. sediminis содержит одну кольцевую хромосому и линейную плазмиду (Таблица 1). GC состав довольно высокий (71.76% и 68.49%). Это может быть связано со способностью бактерии выдерживать высокие температуры, так как большее количество связей между гуанином и цитозином делает ДНК более устойчивой к денатурации.
Таблица 1. Стандартные данные о геноме
| ДНК | Длина (п.н.) | CG-состав |
|---|---|---|
| chromosome | 2 792 195 | 71.76% |
| plasmid | 127 669 | 68.49% |
Результаты представленные в Таблице 2 подтверждают выполнение второго правила Чаргаффа - количество аденина (A) примерно равно количеству тимина (Т), а количество гуанина (G) примерно равно количеству цитозина (C) в одной цепи ДНК. A составляет 50.07% от A+T, G составляет 49.97% от G+C.
Таблица 2. Нуклеотидный состав генома
| ДНК | A | T | C | G |
|---|---|---|---|---|
| chromosome | 39416 | 394234 | 1001464 | 1002337 |
| plasmid | 20712 | 19516 | 42751 | 44690 |
| всего в геноме | 414872 | 413750 | 1046154 | 1045088 |
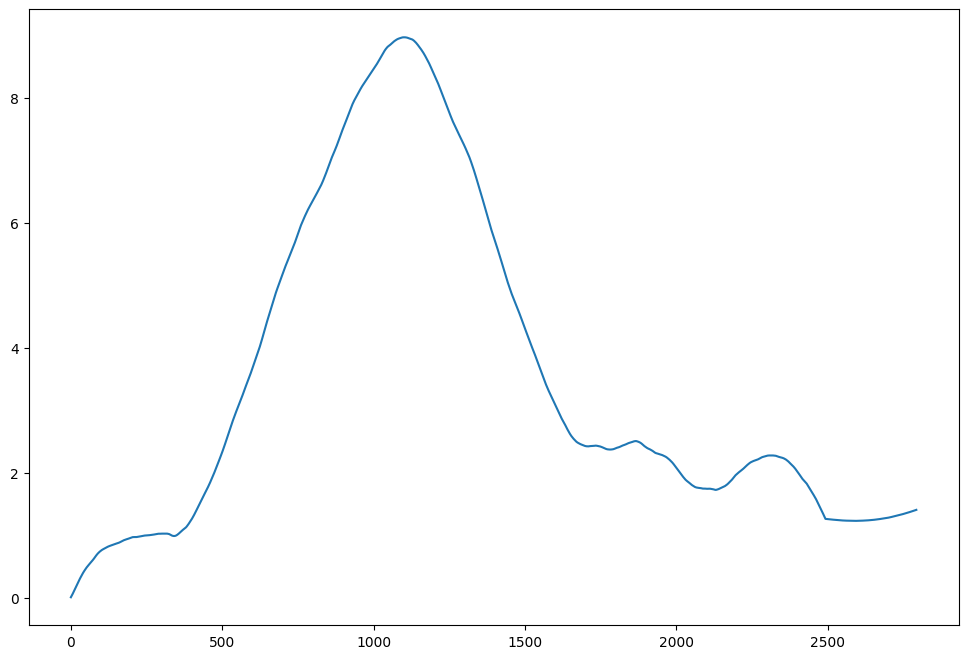
На Рис. 1. представлен cumulative GC-skew. Обычно, минимальное значение соответствует начальной точке репликации, а максимум конечной точке в прокариотическом геноме. В данном графике максимальное значение - 9.71 при х = 1172000, а минимальное значение - 0.01, достигается оно при х = 0. Соответственно начало репликации совпадает с началом файла.
3.2 Данные о белках протеома
На Рис. 2. представлена гистограмма длин белков.
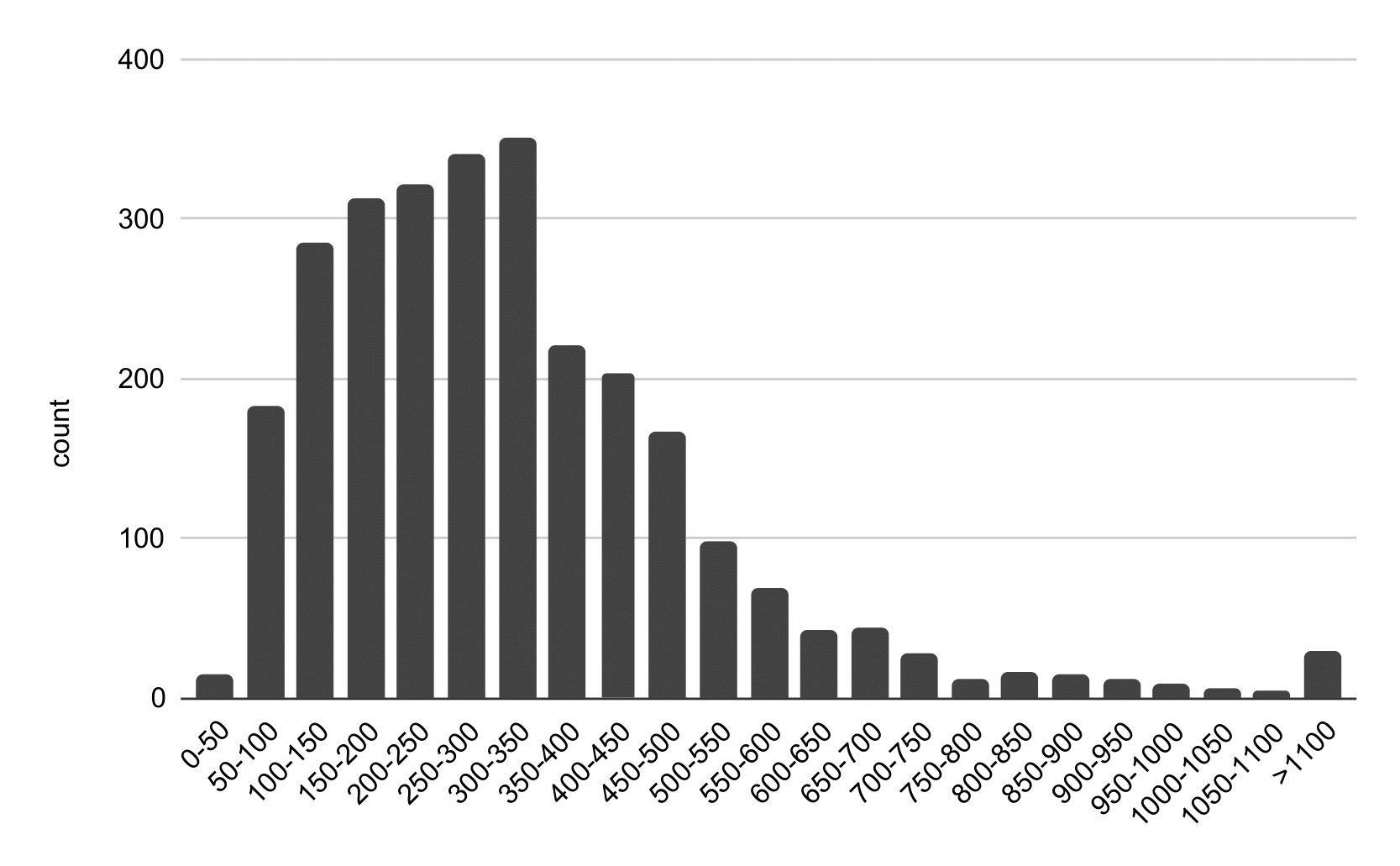
Минимальная длина белка - 32 аминокислот, это AURKAIP1/COX24 domain-containing protein.
Еще один короткий белок - 50S ribosomal protein L36 длиной в 38 аминокислот, это белок большой рибосомальной субъединицы bL36, его ген в неизменном виде встречается у многих бактерий (Escherichia coli, Cronobacter sakazakii, Salmonella typhimurium и paratyphia и т.д.).
Два самых длинных белка это Ig-like domain-containing proteins длинами в 2094 и 2059. Эти белки экспортируются во внешнюю среду и нацелены на две внеклеточные органеллы: жгутики и конъюгативные пили. Они облегчают конъюгацию, уменьшая подвижность клеток и облегчая контакт клетки с клеткой.
Длина этих белков связана с их функцией.
3.3 Данные о пересечениях cds
На рисунке 3 представлена гистограмма расстояний между cds. Чаще всего cds пересекаются и длина пересечения от 1 до 10 нуклеотидов. Пересечением считается количество нуклеотидов, которые есть в обоих cds.
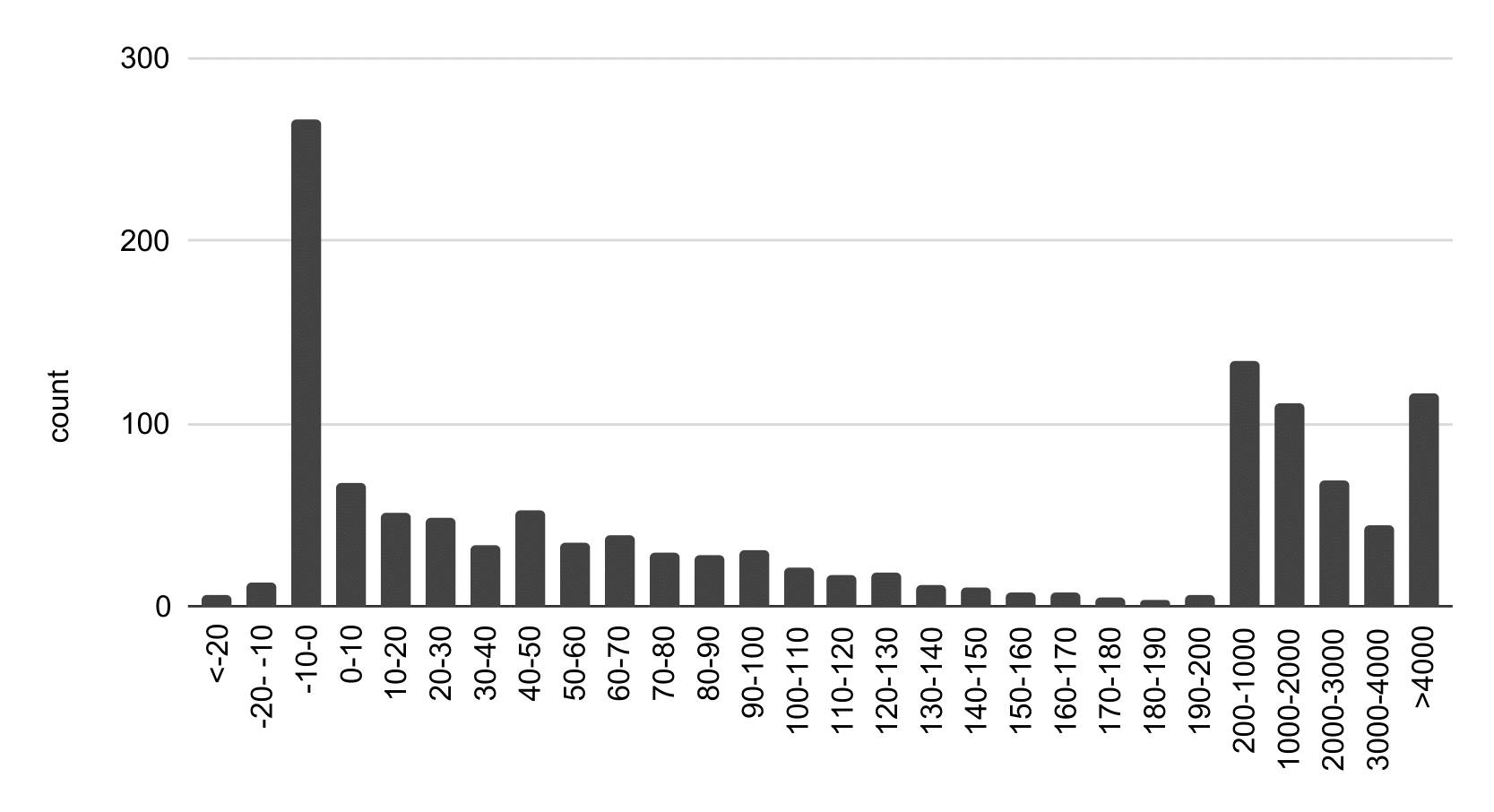
Если рассмотреть подробней на пересечения cds (рисунок 4), то видно, что чаще всего cds пересекаются на 4 нуклеотида.

Вполне логично, что все длина пересечений при делении на три дает либо остаток 1 либо 2, так как иначе стоп кодон первой последовательности останавливал бы транскрипцию второй.
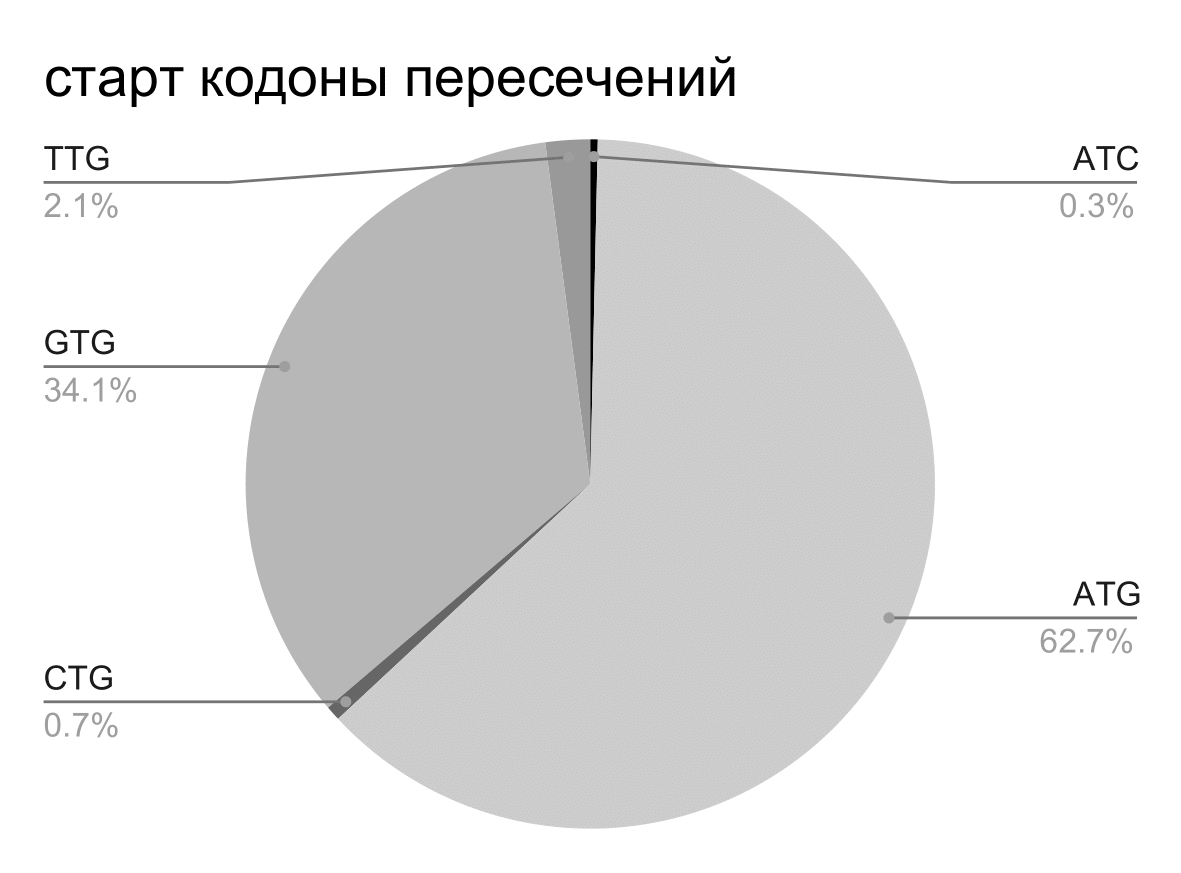
Так же рассмотрим какими нуклеотидными последовательностями являются пересечения. Все пересечения в один нуклеотид - “A”. Это связано с тем что старт кодон начинается на A, и стоп кодоны заканчиваются на А. Пересечения в 4 нуклеотида и их количества приведены в Таблице 3.
Таблица 3. Пересечения cds в 4 нуклеотида
| Пересечение | Количество |
|---|---|
| ATGA | 115 |
| GTGA | 82 |
| TTGA | 1 |
| CTGA | 1 |
Можно увидеть что самое частое пересечение - “ATGA” состоит из старт кодона ATG и стоп кодона, а второе по частоте пересечение начинается с кодона GTG, который часто выполняет функцию старт кодона.
CDS пересечения которых на 20 нуклеотидов и больше приведены в Таблице 4. Максимальное пересечени - 119 нуклеотидов, но оно между restriction endonuclease subunit S и теоретическим белком и составляет 37.78% от длины теоретического белка и 14.75% от длины restriction endonuclease subunit S. Так же есть пересечение между nrdI и nrdE в 22 нуклеотида. Эти белки имеют схожую функцию и такое пересечение может быть связано с регуляцией экспрессии, так же рядом с этим участком также находится ген nrdH и nrdF.
Таблица 4. Подробней про пересечения в 20 и больше нуклеотидов.
| название белка | пересечение |
|---|---|
| restriction endonuclease subunit S hypothetical protein - |
119 |
| IS3 family transposase tyrosine-type recombinase/integrase - |
68 |
| non-ribosomal peptide synthetase M1 family metallopeptidase - |
49 |
| class Ib ribonucleoside-diphosphate reductase assembly flavoprotein NrdI (symbol nrdI) class 1b ribonucleoside-diphosphate reductase subunit alpha (symbol nrdE) - |
22 |
| bifunctional UDP-N-acetylglucosamine diphosphorylase/glucosamine-1-phosphate
N-acetyltransferase GlmU (symbol glmU) ribose-phosphate diphosphokinase - |
23 |
| phosphonate ABC transporter, permease protein PhnE (symbol phnE)
MurR/RpiR family transcriptional regulator - |
23 |
| glutamine synthetase family protein gamma-glutamyl-gamma-aminobutyrate hydrolase 20 family protein |
20 |
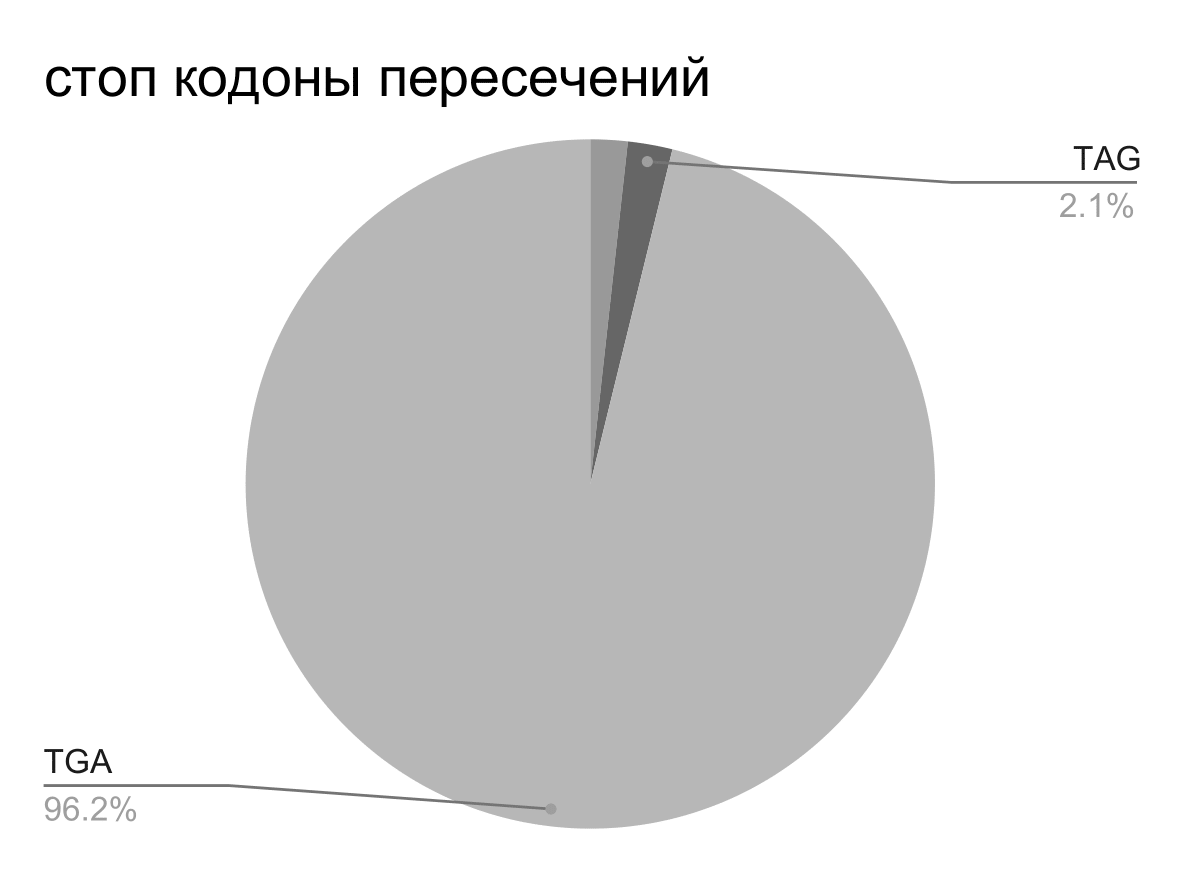
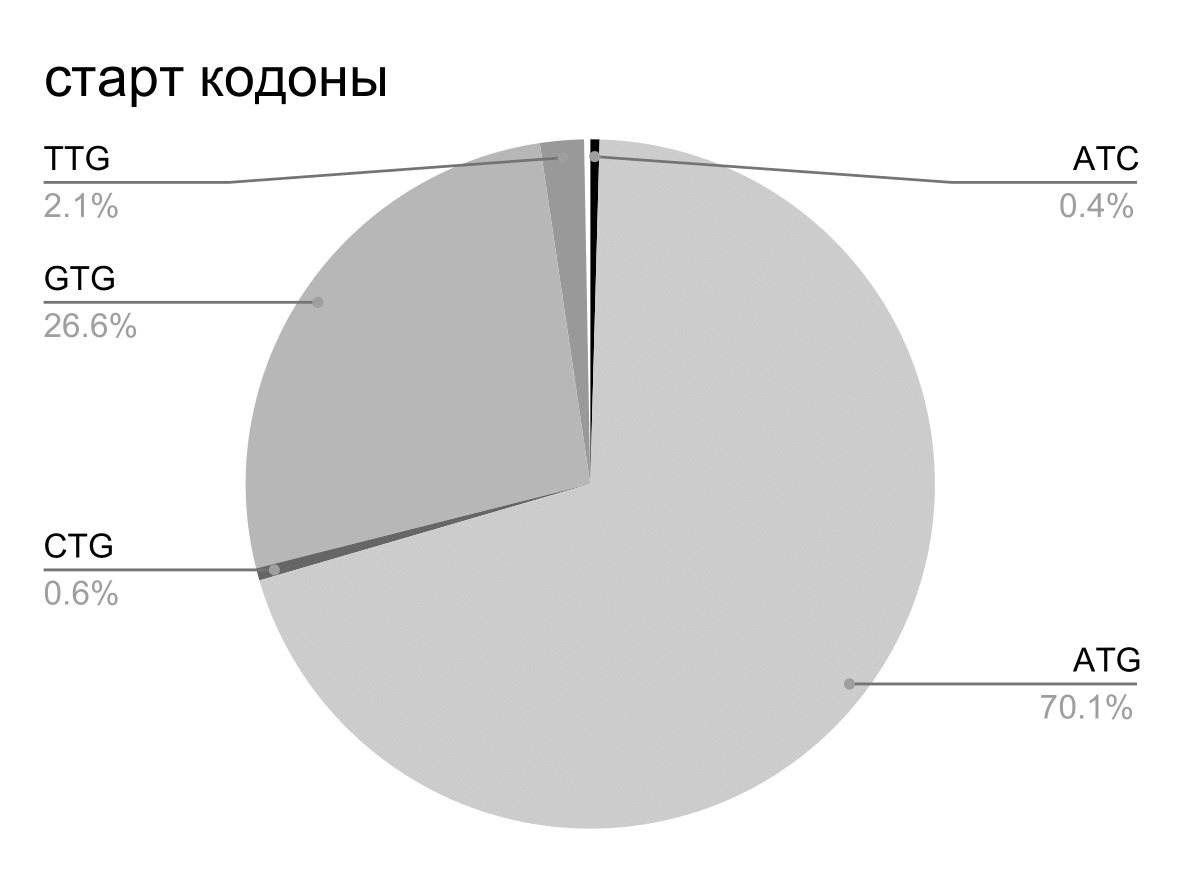
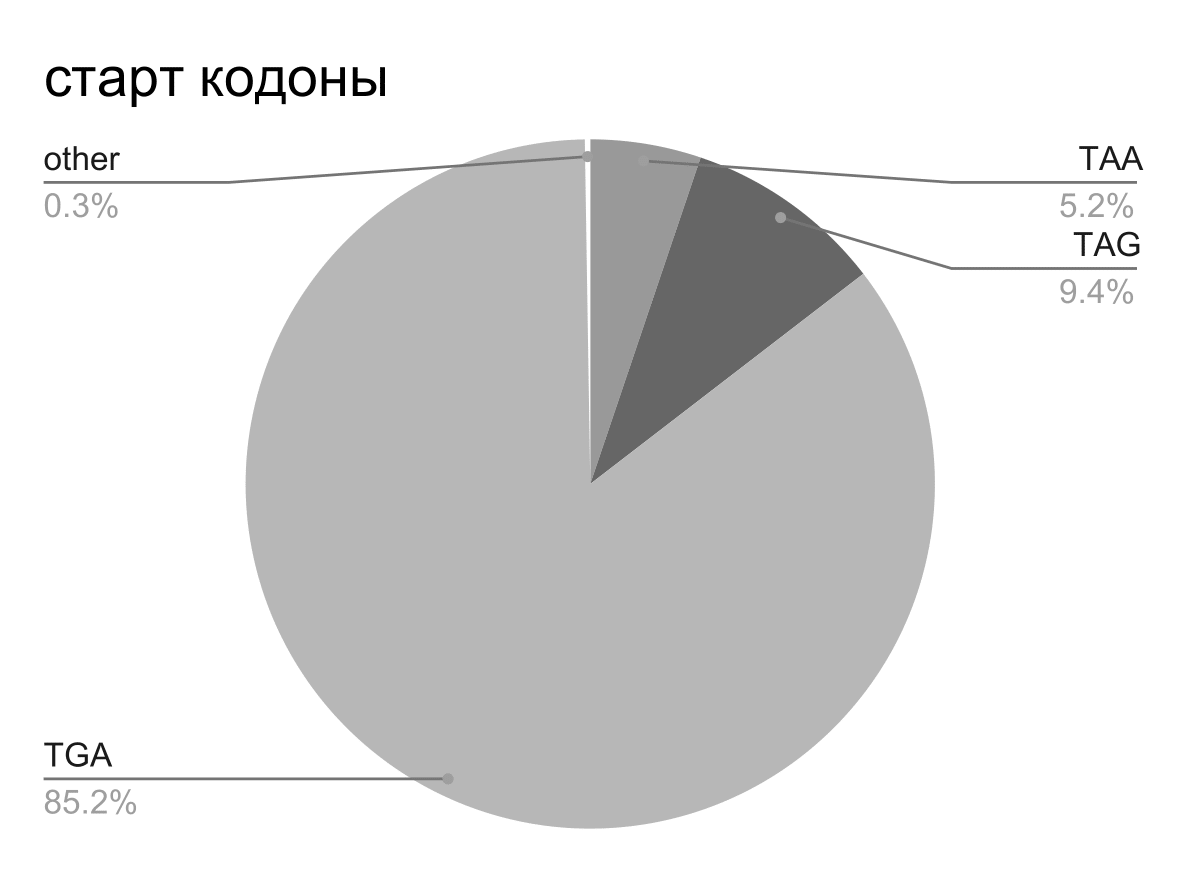
На Рисунках 5 и 6 представлены круговые диаграммы для старт и стоп кодонов которые попадают в пересечения cds, в то время как на Рисунках 7 и 8 представлены круговые диаграммы для старт и стоп кодонов для всех cds на хромосоме. Можно видеть, что почти все стоп кодоны в пересечениях - “TGA”, а также относительное количество старт кодона “GTG” больше в пересечениях. Пересечения генов могут быть связаны с регуляцией экспрессии и возможно большее относительное содержание старт кодона “GTG” также с этим связано.
Сопроводительные материалы
Список литературы
- Jebbar, M., Franzetti, B., Girard, E., Oger, P., 2015. Microbial diversity and adaptation to high hydrostatic pressure in deep-sea hydrothermal vents prokaryotes. Extremophiles 19 (4), 721–740.
- Liu, D., Lin, H., Proksch, P., Tang, X., Shao, Z., & Lin, W. (2015). Microbacterins A and B, New Peptaibols from the Deep Sea Actinomycete Microbacterium sediminis sp. nov. YLB-01(T). Organic Letters, 17(5), 1220–1223.
- Poli, A., Finore, I., Romano, I., Gioiello, A., Lama, L., Nicolaus, B., 2017. Microbial diversity in extreme marine habitats and their biomolecules. Microorganisms 2. Sanders, H.L., Hessler, R.R., 1969. Ecology of the deep-sea benthos. Science 163 (3874), 1419–1424.
- Yu, L., Lai, Q., Yi, Z., Zhang, L., Huang, Y., Gu, L., & Tang, X. (2012). Microbacterium sediminis sp. nov., a psychrotolerant, thermotolerant, halotolerant and alkalitolerant actinomycete isolated from deep-sea sediment. INTERNATIONAL JOURNAL OF SYSTEMATIC AND EVOLUTIONARY MICROBIOLOGY, 63(Pt 1), 25–30.
- Yu, L., Lai, Q., Yi, Z., Zhang, L., Huang, Y., Gu, L., Tang, X., 2013. Microbacterium sediminis sp. nov., a psychrotolerant, thermotolerant, halotolerant and alkalitolerant actinomycete isolated from deep-sea sediment. Int. J. Syst. Evol. Microbiol. 63 (Pt 1), 25–30.
- Yi, Z., Cao, X., Li, H., Jian, H., Xu, X., Yu, L., & Tang, X. (2020). Genomic analysis of Microbacterium sediminis YLB-01T reveals backgrounds related to its deep-sea environment adaptation. Marine Genomics, 100818.