Описание лизин-рацемазы
Oenococcus oeni PSU-1
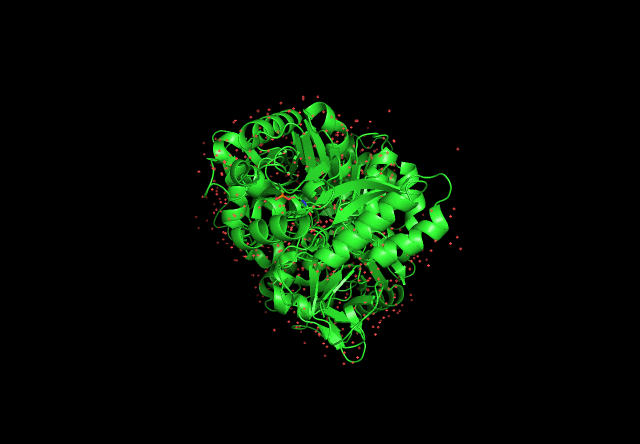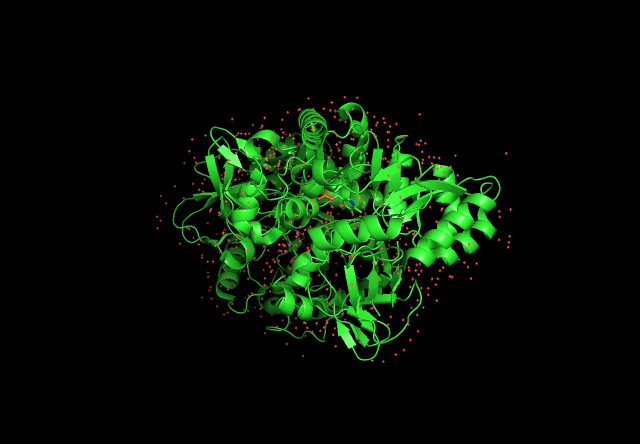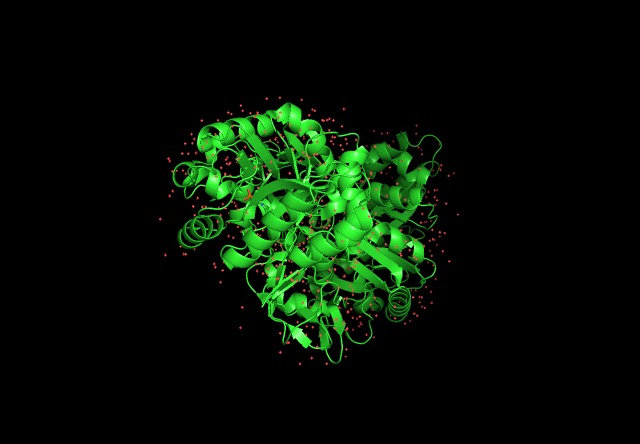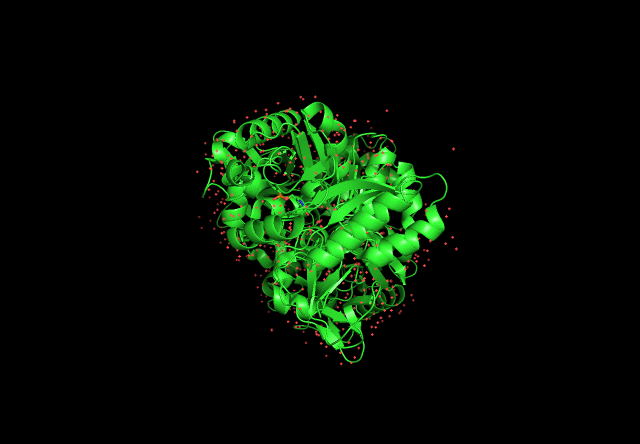
Cтруктура белка, полученная при помощи pyMOL. 'E pur si muove!' - Galileo Galilei.
Oenococcus oeni - это ацидофил, осуществляющий малолактозную ферментацию вина. PSU-1 - облигатно гетероферментативная бактерия (в результате брожения образуется несоклько продуктов), в её геноме закодированы все ферменты фосфокетолазного пути. В результате этого пути образуется лактат и этанол. Изготовление вина состоит из нескольких стадий. Первая - это ферментация винограда Saccharomyces, которые специально прививаются ещё до сбора либо присутствуют в винограде и так. Эта первичная ферментация создёт анаэробную среду, которая лимитирует рост других дрожжей, что защищает вино от скисания. Малолактозное брожение - это вторая стадия. Она делает вино более кислым, превращая малат в лактат. В основном вина повышенной кислотности производятся в регионах с холодным климатом. [1]
Лизин-рацемаза Oenococcus oeni изучается в связи с обраружением в вине D-Lys. Рацемазы в целом делятся на две группы: пиридоксаль-5-фосфат-независимые (PLP-independent) и PLP-зависимые. У PLP-зависимых рацемаз прослеживается гомология. У лизин-рацемазы два активных сайта и два связывающих сайта. Из интересного: лизин и аланин-рацемаза имеют очень схожую струтуру, однако аланин-рацемаза может катализировать рацемизацию и аланина, и лизина, а лизин-рацемаза - только лизина. [2]
На анимации 1 показана структура лизин-рацемазы Oenococcus oeni. Зелёные стрелки показывают бета-цепи, зелёные ленты со стрелками - альфа спирали. Всего структура содержит 22 бета-цепи и 18 альфа-спиралей. Также на структуре видны аминокислоты двух связывающих и двух активных сайтов (1 активный сайт: тирозин, 1 связывающий сайт: метионин, 2 активный сайт: лизин и 2 связывающий сайт: аргинин). Структура получена с сайта NCBI и обработана в PyMOL.
Базовая инофрмация о данном белке
При помощи базы данных NCBI мы узнали последовательность белка и основные его характеристики. Поиск производился по индентификатору белка в разделе Protein. Файл с последовательностью был оформлен самостоятельно. Кодировка файла - ASCII, кодировка концов строк - Unix, ширина строки - 60 символов. Основные данные о белке приведены в таблице 1.
Последовательность была получена из базы данных NCBI: скачать файл с последовательностью в формате fasta
| Идентификатор белка | ABJ56155.1 |
|---|---|
| Идентификатор генома | CP000411.1 |
| Координаты гена в геноме | 150759..151874 |
| Длина гена (п.н.) | 1116 |
| Цепь | Прямая |
| Длина белка (а.о.) | 371 |
Функция данной конкретной рацемазы ещё не полностью изучена, но в целом лизин-рацемазы вовлечены в катаболизм лизина. В бактериях он протекает двумя путями. Один из них идёт через дельта-аминовалериановую кислоту, другой через рацемизацию лизина. Ключевые метаболиты второго пути - это альфа-аминоадипат (AMA) и L-пипеколат [3]. Из этого следует интересное предположение. Несколько лет назад было показано, что метаболизм лизина и в частности повышенный уровень L-пипеколата делает бактерий более устойчивыми к осмотическому стрессу [4]. Возможно, Oenococcus oeni испытывает в вине осмотический стресс, чем и вызвана высокая специфичность его лизин-рацемазы.
Была получена информация о геномном окружении данного белка, она приведена на рисунке 1. Соседние белки: ацетилтрансфераза (ABJ56154.1, обратная цепь), ферредоксин-НАДФН-редуктаза (ABJ56156.1, прямая цепь). Чуть дальше находятся рибозо-фосфат-пирофосфокиназа (ABJ56153.1, прямая цепь) и белок, содержащий GAF-домен (ABJ56157.1, прямая цепь).

Геномное окружение гена белка лизин-рацемазы (ABJ56155.1). Изображение получено с помощью геномного браузера NCBI. Красные полосы - гены различных белков. Стрелки указывают, в каком направлении они считываются. По их направлению можно определить, что исследуемый белок закодирован на прямой цепи ДНК. Выше показаны другие возможные рамки считывания. Зелёными палочками отмечены старт-кодоны, красными - стоп-кодоны. Серые полосы обозначают открытые рамки считывания.
Итоги
- Oenococcus oeni - бактерия, важная в производстве вина и способствующая его подкислению;
- из базы данных NCBI были получены основные характеристики лизин-рацемазы Oenococcus oeni, а также её аминокислотная последовательность;
- лизин-рацемаза данной бактерии высоко специфична в отличие от других её рацемаз;
- путь расщепления лизина с участием лизин-рацемазы важен для бактерий при осмотическом стрессе. Высокую специфичность данной рацемазы можно объяснить тем, что, возможно, находясь в вине, Oenococcus oeni испытывает постоянный стресс.
Ссылки на использованную литературу
- [1] David A. Mills, Helen Rawsthorne, Kira Makarova; Genomic analysis of Oenococcus oeni PSU-1 and its relevance to winemaking, FEMS Microbiology Reviews, Volume 29, Issue 3, 1 August 2005, Pages 465–475, https://doi.org/10.1016/j.fmrre.2005.04.011
- [2] Shiro Kato, Hisashi Hemmi, Tohru Yoshimura; Lysine racemase from a lactic acid bacterium, Oenococcus oeni: structural basis of substrate specificity, The Journal of Biochemistry, Volume 152, Issue 6, 1 December 2012, Pages 505–508, https://doi.org/10.1093/jb/mvs120
- [3] I-Chien Chen, Wei-De Lin, Shin-Kuang Hsu, Venkatesan Thiruvengadam, Wen-Hwei Hsu; Isolation and Characterization of a Novel Lysine Racemase from a Soil Metagenomic Library, Appl. Environ. Microbiol. Jul 2009, 75 (15) 5161-5166; doi: 10.1128/AEM.00074-09
- [4] Izabella AP Neshich, Eduardo Kiyota, Paulo Arruda; Genome-wide analysis of lysine catabolism in bacteria reveals new connections with osmotic stress resistance, ISME J. 2013 Dec; 7(12): 2400–2410. Published online 2013 Jul 25. doi: 10.1038/ismej.2013.123