EMBOSS и протеомы
Два полных протеома были скачаны из базы данных Uniprot в fasta-формате. Количество остатков было подсчитано при помощи
команды
awk '{sum+=$2} END {print sum}' файл, полученный при помощи команды wordcount
Escherichia coli (штамм K12 / MG1655 / ATCC 47076)
Proteome ID: UP000000625
Число записей: 4352
Количество остатков: 1353357
Oenococcus oeni (штамм ATCC BAA-331 / PSU-1)
Proteome ID: UP000000774
Число записей: 1682
Количество остатков: 486326
Таблица, описывающая разность частот аминокислот в протеомах (см. Таблица 1), была составлена при помощи python-скрипта (скачать,
команда запуска: python pr7_Nogina.py), полученный
tsv-файл был конвертирован
в html- таблицу при использовании онлайн-конвертера.
| а.о. | E.coli, % | O.oeni, % | Разность (E. coli - O.oeni), % |
|---|---|---|---|
| L | 10.6763 | 9.9483 | 0.728 |
| A | 9.5072 | 8.2568 | 1.2504 |
| G | 7.3661 | 7.5419 | -0.1758 |
| V | 7.0703 | 7.3996 | -0.3293 |
| I | 6.0115 | 7.0899 | -1.0784 |
| S | 5.7991 | 6.4391 | -0.64 |
| E | 5.7623 | 6.3885 | -0.6262 |
| R | 5.5205 | 5.6429 | -0.1224 |
| T | 5.3945 | 5.3608 | 0.0337 |
| D | 5.1469 | 5.3376 | -0.1907 |
| Q | 4.4427 | 5.3359 | -0.8932 |
| P | 4.4288 | 5.0661 | -0.6373 |
| K | 4.407 | 4.0134 | 0.3936 |
| N | 3.9383 | 3.8992 | 0.0391 |
| F | 3.8944 | 3.422 | 0.4724 |
| Y | 2.8449 | 3.328 | -0.4831 |
| M | 2.8253 | 2.3316 | 0.4937 |
| H | 2.2695 | 1.8595 | 0.41 |
| W | 1.5321 | 1.0067 | 0.5254 |
| C | 1.1621 | 0.3323 | 0.8298 |
| U | 0.0002 | 0.0 | 0.0002 |
Комментарий
Чаще всего в протеоме O.oeni встречаются остатки лейцина, аланина и глицина (9.9483%, 8.2568%, 7.5419%), в протеоме E.coli также остатки лейцина, аланина и глицина (10.6763%, 9.5072%, 7.3661%) соответственно. Самыми редкими остатками у O.oeni и E.coli являются гистидин, триптофан и цистеин. Самая большая разница в содержании остатков в пользу O.oeni наблюдается для остатков изолейцина - 1.0784%. Самая большая разница в содержании остатков в пользу E.coli наблюдается для остатков аланина - 1.2504%.
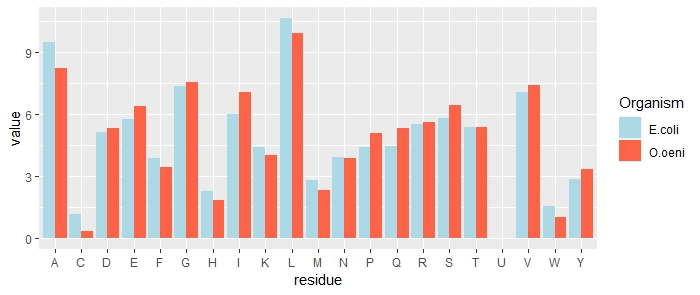
Рисунок 1. Частоты аминокислот в протеомах.
На рис.1 наглядно показаны различия встречаемости различных аминокислот. Можно увидеть, что общая картина в случае O.oeni и E.coli примерно повторяется, но при этом наблюдаются как заметные различия для отдельных аминокислот, так и очень схожие процентные содержания (например, в случае треонина).
wordcount
Подсчёт всех возможных уникальных последовательностей заданной длины в одной или нескольких последовательностях.
output: файл с двумя колонками, разделёнными '\t', в первой колонке сама последовательность, во второй - её количество в анализируемом файле.
Формат стандартного запроса приблизительно тот же, что и у compseq, указывается название файла с последовательностями [-sequence], название файла для записи [-outfile] и длина n-мера -wordsize (в случае compseq это -word). Если имя файла для записи не указывается, создаётся файл с расширением .wordcount. Длина по умолчанию от 2 до 4 символов.
С -sequence ассоциировано много полезных опций (с -outfile только одна: -odirectory2, позволяет выбрать директорию, в которую будет записан файл). Например, можно обозначить начало и конец анализируемого участка последовательностей (-sbegin1 и -send1), если имеем дело с ДНК, то считать её обратную цепь (-sreverse1), можно учитывать цикличность ДНК (-scircular1) и прочее.
У wordcount только одна дополнительная опция: -mincount, которая устанавливает нижнюю границу выдачи (по умолчанию 1).
Подсчёт отдельных аминокислот при помощи wordcount в протеоме E.coli занимает 4.448 секунды.
compseq
output отличается от такового команды wordcount. Первые несколько строчек начинаются с # и содержат общую информацию и комментарии. Затем в двух отдельных строчках даются длина n-мера (указанная в -word в input) и общее количество.
Названия колонок расположены в строчках, начинающихся на #. Сами колонки: подпоследовательность, количество, наблюдаемая частота, частота, подчитанная на основе предположения, что все найденные подпоследовательности встречаются с равной вероятностью, отношение наблюдаемой частоты к ожидаемой. Далее следует строчка Other для всего странного и непонятного.
Опции, ассоциированные с -sequence и -outfile те же, что и у wordcount. Зато дополнительных опций больше (-mincount нет). Всего их шесть.
1. -infile
Файл, который был сгенерирован ранее при помощи compseq, может использоваться для установки ожидаемых частот. Размер n-мера и тип молекулы (белок/ДНК) должны быть теми же.
2. -frame
При помощи этой опции можно задать рамку считывания
3. -[no]ignorebz
Аминокислотный код B представляет собой аспарагин или аспарагиновую кислоту, а код Z представляет собой глутамин или глутаминовую кислоту. Это не часто используемые коды, поэтому можно не считать слова, содержащие их, просто отмечая их в графе "другие" слова (Other).
4. -reverse
Анализировать также обратную цепь.
5. -calcfreq
Если задано значение true, то ожидаемые частоты слов вычисляются по наблюдаемой частоте одиночных оснований или остатков в последовательностях. Если сообщать размер слова 1 (одиночные основания или остатки), то нет смысла использовать эту опцию, потому что вычисленная ожидаемая частота будет равна наблюдаемой частоте. Если был указан входной файл ожидаемых частот слов, то вместо этого вычисления ожидаемой частоты из последовательности будут использоваться значения из этого файла, даже если "calcfreq" имеет значение true.
6. -[no]zerocount
Не записывать в файл результаты для невстречающихся слов.
Подсчёт отдельных аминокислот при помощи compseq в протеоме E.coli занимает 1.730 секунду.
В будущем я буду использовать главным образом compseq, а не wordcount, потому что: 1) он быстрее, 2) есть подсчёт процентного содержания, что очень удобно, 3) за счёт большего числа опций compseq более дружелюбен и удобен для пользователя.