Филогенетическое дерево
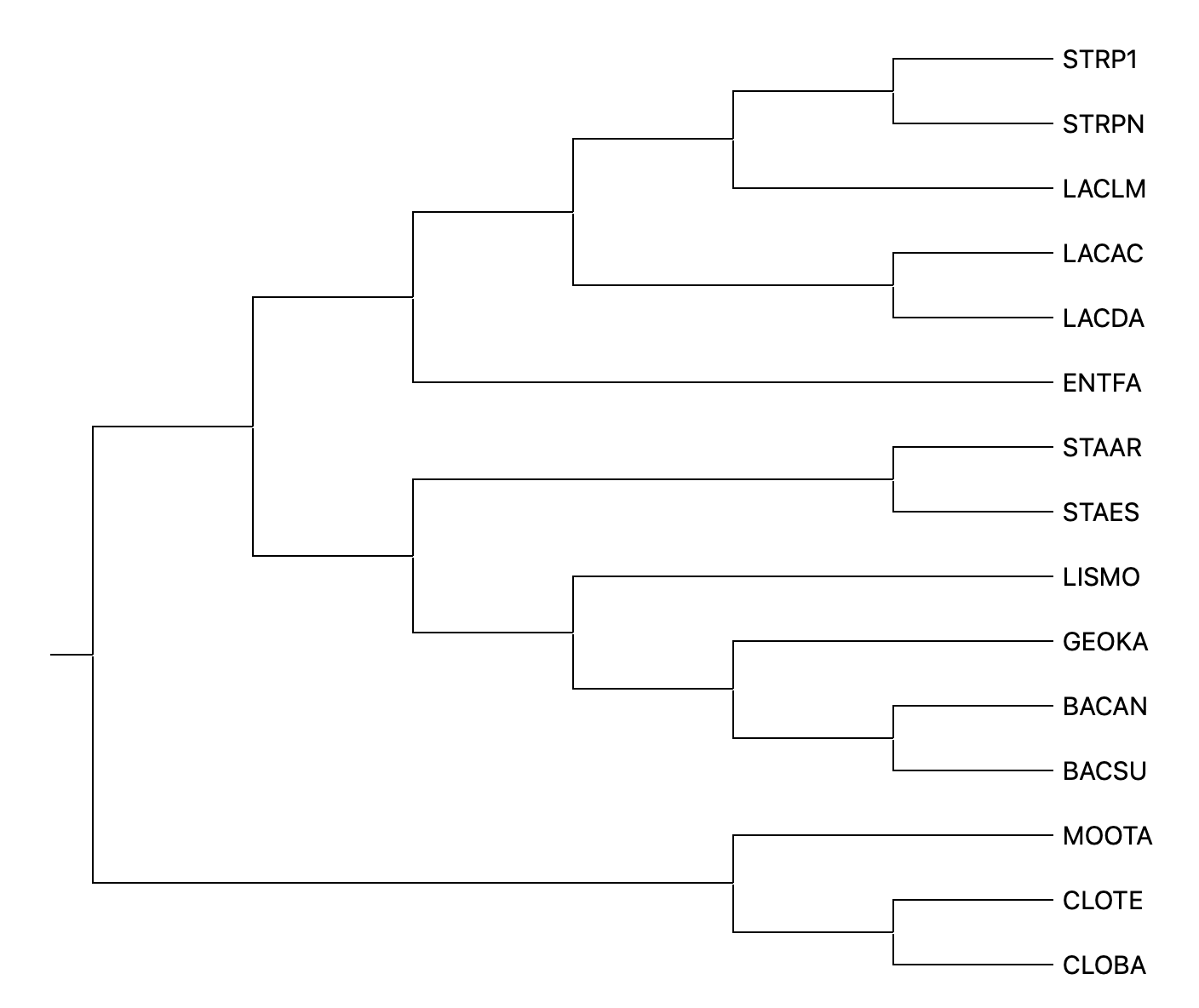
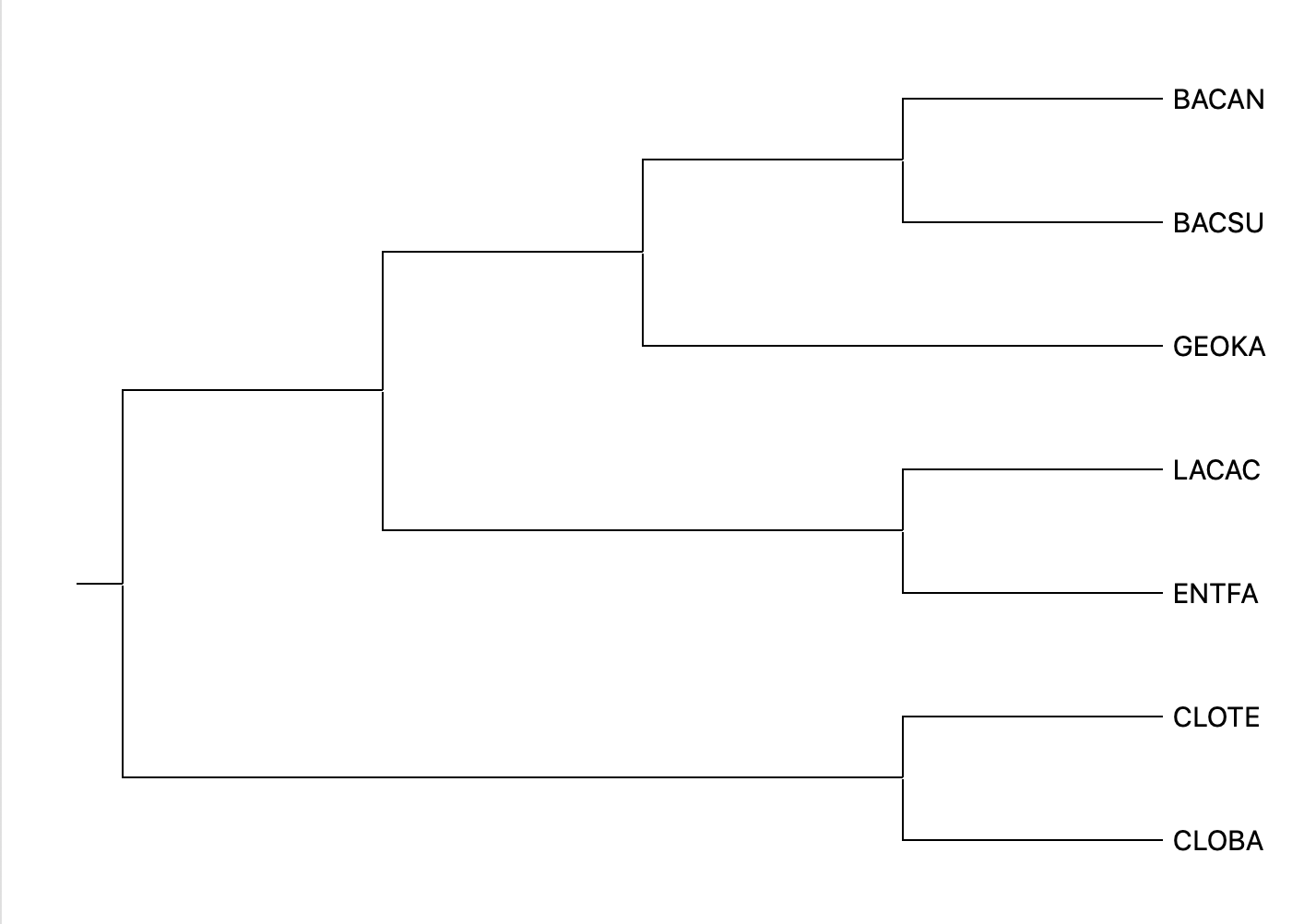
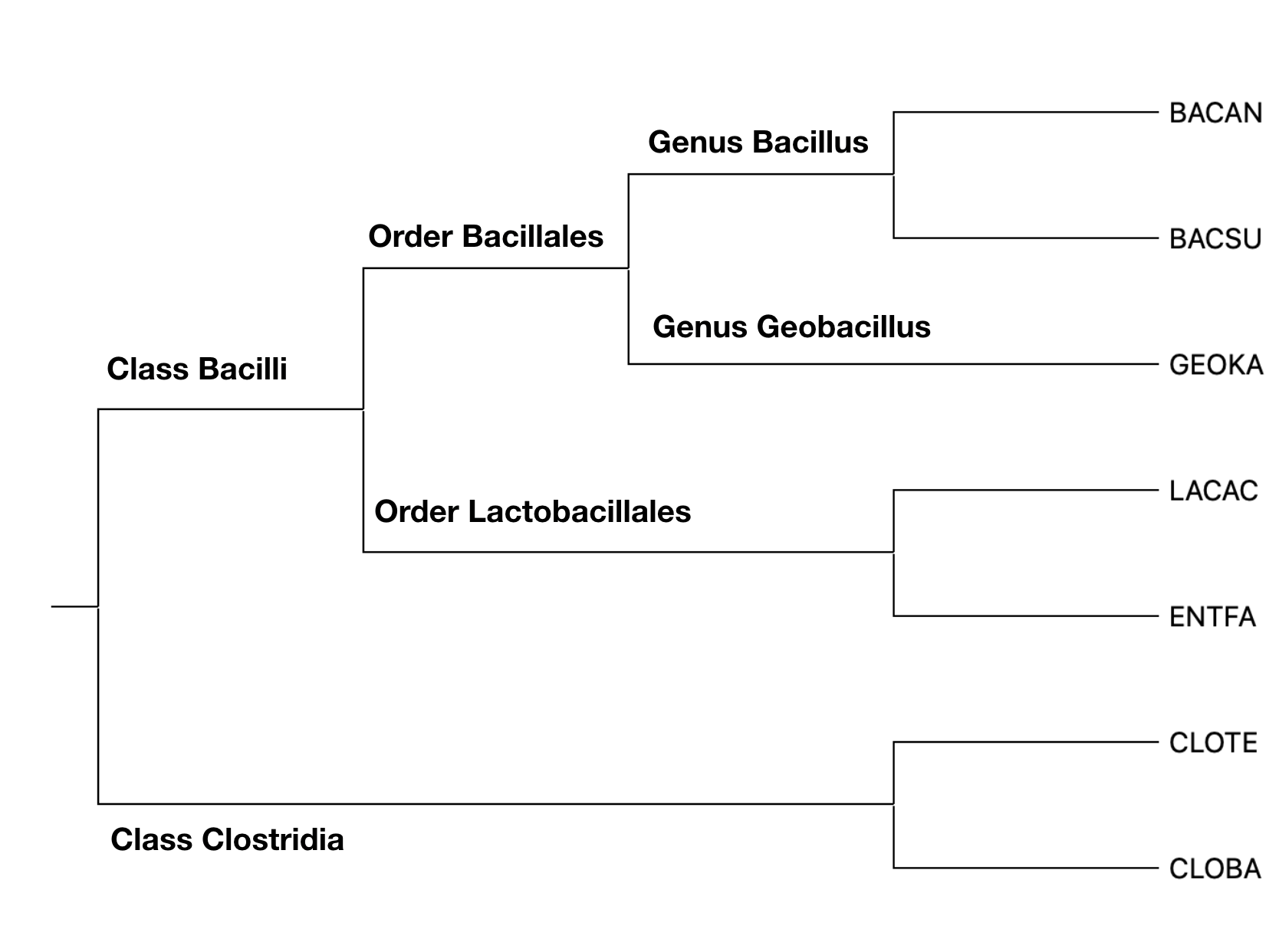
Выбранные бактерии.
| Название | Мнемоника |
| Bacillus anthracis | BACAN |
| Bacillus subtilis | BACSU |
| Clostridium botulinum | CLOBA |
| Clostridium tetani | CLOTE |
| Enterococcus faecalis | ENTFA |
| Geobacillus kaustophilus | GEOKA |
| Lactobacillus acidophilus | LACAC |
Набор нетривиальных разбиений:
{BACAN, BASCU} VS {GEOKA, LACAC, ENTFA, CLOTE, CLOBA}
{BACAN, BASCU, GEOKA} VS {LACAC, ENTFA, CLOTE, CLOBA}
{BACAN, BASCU, GEOKA, LACAC, ENTFA} VS {CLOTE, CLOBA}
Выбранный белок: 16S рРНК-метилтрансфераза H (RSMH). Было построено выравнивание белковых последовательностей в программе muscle. Затем оно было отредактировано, оставлена только мнемоника вида.
Задание 4
Все протеомы нужных бактерий были объеденены в all.fa. Потом создана база данных для локального поиска.
makeblastdb -in all.fa -dbtype prot
blastp -query CLPX_ECOLI.fa -db all.fa -out res.out -evalue 0.001
Выдача
Пары ортологов: CLPX_BACAN - CLPX_BACSU, CLPX_CLOTE - CLPX_CLOBH, HSLU_ENTFA - HSLU_LACAC
Пары паралогов: HSLU_GEOKA - Q5KUR3_GEOKA, Q5FKR6_LACAC - Q5FHW6_LACAC, CLPX_ENTFA - Q839B1_ENTFA
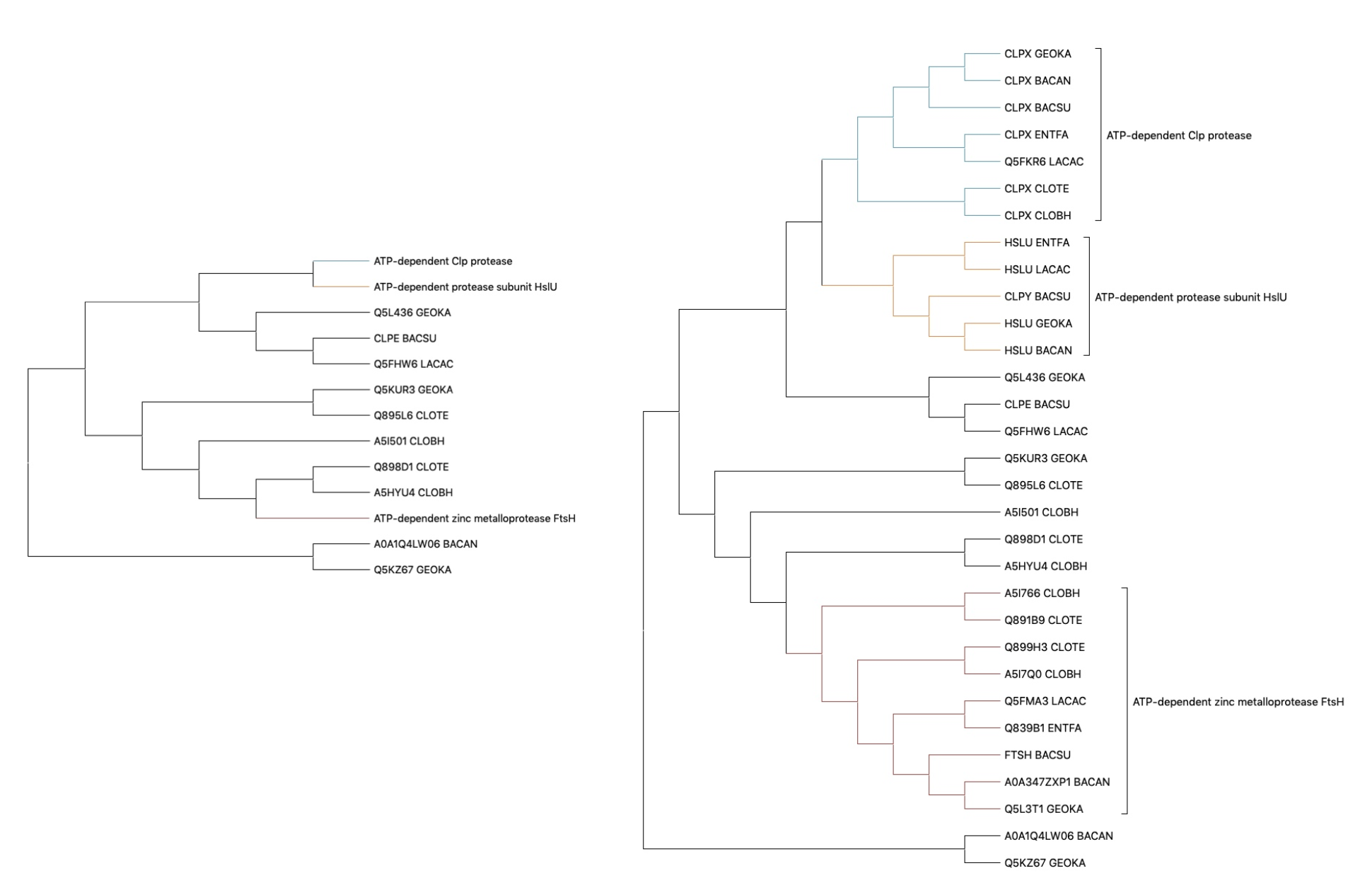
Рис.4. Филогенетическое дерево с выделенными по группам ортологами + дерево, где группы ортологичных белков схлопнуты.
summary:
1) группа CLPX - частично соответсвует филогении. Ветвь, отделяющая GEOKA и BACAN от BACSU, не соответствует филогении.
2) группа HlsU - соответствует филогении
3) группа FtsH - мало соответсвует филогении.
В верхней группе представлены белки CPLX - это компонент Clp протеазы, отвечающий за ее АТФ-зависимую специфичность. Разделение CLPX для GEOCA, в соответствие с филогенией, должно было предшествовать разделению BACAN и BACSU. Разделение между LACAC и ENTFA можно считать соответсвующим филогении.
Вторая группа состоит из белков HslVU. Во второй группе можно заметить белок CLPY_BACSU - я сначала приняла его за белок CLPY, однако, его отличие в названии объясняется просто немного другой аннотации, в целом он ортологичен остальным. То, что пары GEOKA и BACAN и ENTFA и LACAC принадлежат разным ветвям, согласуется с филогенией.
Следующая группа довольно обширна и включает в себя белки, связанные с металлами. С филогенией согласуется довольно слабо: так, CLOBH и CLOTE были два раза выделены в две отдельные ветви. BACAN и GEOKA снова отделены от BACSU, как в группе CLPX, что не соответсвует филогении.