|
g0 |
g1 |
g2 |
g3 |
g4 |
a |
b |
c |
d |
e |
f |
| g0 |
0 |
70 |
50 |
65 |
60 |
100 |
100 |
90 |
90 |
100 |
100 |
| g1 |
— |
0 |
120 |
135 |
130 |
30 |
30 |
160 |
160 |
170 |
170 |
| g2 |
— |
— |
0 |
15 |
10 |
150 |
150 |
40 |
40 |
50 |
50 |
| g3 |
— |
— |
— |
0 |
25 |
165 |
165 |
25 |
25 |
65 |
65 |
| g4 |
— |
— |
— |
— |
0 |
160 |
160 |
50 |
50 |
40 |
40 |
| a |
— |
— |
— |
— |
— |
0 |
60 |
190 |
190 |
200 |
200 |
| b |
— |
— |
— |
— |
— |
— |
0 |
190 |
190 |
200 |
200 |
| c |
— |
— |
— |
— |
— |
— |
— |
0 |
50 |
90 |
90 |
| d |
— |
— |
— |
— |
— |
— |
— |
— |
0 |
90 |
90 |
| e |
— |
— |
— |
— |
— |
— |
— |
— |
— |
0 |
80 |
| g |
— |
— |
— |
— |
— |
— |
— |
— |
— |
— |
0 |
|
Слева представлена треугольная матрица расстояний между нуклеотидными последовательностями, полученными ранее
(см. Эволюционную модель).
|
Матрица попарного различия: среднее число несовпадающих нуклеотидов на 100 позиций
|
g0 |
g1 |
g2 |
g3 |
g4 |
a |
b |
c |
d |
e |
f |
| g0 |
0.00 |
34.54 |
30.93 |
37.97 |
35.22 |
45.02 |
46.91 |
46.91 |
46.22 |
47.08 |
48.45 |
| g1 |
— |
0.00 |
49.48 |
54.30 |
52.41 |
20.10 |
19.93 |
59.11 |
59.62 |
60.65 |
61.17 |
| g2 |
— |
— |
0.00 |
11.86 |
7.56 |
54.64 |
57.39 |
27.66 |
26.12 |
28.18 |
30.58 |
| g3 |
— |
— |
— |
0.00 |
19.07 |
58.25 |
60.82 |
17.35 |
16.49 |
34.88 |
39.18 |
| g4 |
— |
— |
— |
— |
0.00 |
56.01 |
59.97 |
33.16 |
32.13 |
24.23 |
24.91 |
| a |
— |
— |
— |
— |
— |
0.00 |
35.57 |
61.34 |
63.40 |
61.51 |
64.09 |
| b |
— |
— |
— |
— |
— |
— |
0.00 |
63.06 |
63.92 |
66.15 |
65.46 |
| c |
— |
— |
— |
— |
— |
— |
— |
0.00 |
29.04 |
45.19 |
47.08 |
| d |
— |
— |
— |
— |
— |
— |
— |
— |
0.00 |
45.36 |
48.80 |
| e |
— |
— |
— |
— |
— |
— |
— |
— |
— |
0.00 |
40.03 |
| g |
— |
— |
— |
— |
— |
— |
— |
— |
— |
— |
0.00 |
|
Матрица, представленная слева, получена при помощи программы distmat пакета EMBOSS.
При этом выравнивания были сделаны без вставок и делеций (т.к. их не предусматривает наша модель
эволюции): последовательности были просто написаны друг под другом. Программа distmat
использовалась без коррекции расстояний (с параметром uncorrected distances).
|
Матрица попарных эволюционных расстояний, вычисленных по методу Джукса - Кантора
|
g0 |
g1 |
g2 |
g3 |
g4 |
a |
b |
c |
d |
e |
f |
| g0 |
0.00 |
46.28 |
39.87 |
52.94 |
47.57 |
68.76 |
73.65 |
73.65 |
71.84 |
74.11 |
77.89 |
| g1 |
— |
0.00 |
80.871 |
96.541 |
89.981 |
23.403 |
23.173 |
116.37 |
118.84 |
124.05 |
126.79 |
| g2 |
— |
— |
0.00 |
12.90 |
7.97 |
97.79 |
108.67 |
34.52 |
32.10 |
35.34 |
39.29 |
| g3 |
— |
— |
— |
0.00 |
22.01 |
112.42 |
124.95 |
19.74 |
18.63 |
46.92 |
55.41 |
| g4 |
— |
— |
— |
— |
0.00 |
103.03 |
120.54 |
43.78 |
41.95 |
29.26 |
30.28 |
| a |
— |
— |
— |
— |
— |
0.00 |
48.22 |
127.73 |
140.00 |
128.68 |
144.58 |
| b |
— |
— |
— |
— |
— |
— |
0.00 |
137.81 |
143.41 |
160.29 |
154.68 |
| c |
— |
— |
— |
— |
— |
— |
— |
0.00 |
36.73 |
69.20 |
74.11 |
| d |
— |
— |
— |
— |
— |
— |
— |
— |
0.00 |
69.63 |
78.87 |
| e |
— |
— |
— |
— |
— |
— |
— |
— |
— |
0.00 |
57.23 |
| g |
— |
— |
— |
— |
— |
— |
— |
— |
— |
— |
0.00 |
|
Эта матрица была получена на основе того же множественного выравнивания, что и предыдущая,
при помощи той же программы distmat. Но была применена формула Джукса – Кантора (JC).
|
Сравнение 2 методов определения эволюционных расстояний
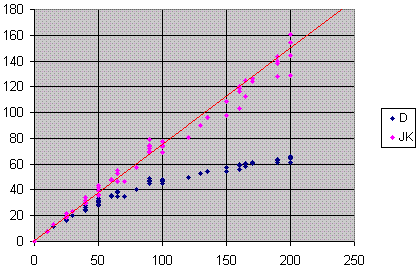
|
- По оси x отложено количество мутаций, сделанных программой msbar (что в 4/3 раза более
истиного расстояния, т.к. замену нуклеотида сам на себя программа считает мутацией)
- По оси y отложены:
- Синим цветом — расстояния между последовательностями согласно методу неоткорректированных расстояний
(D)
- Розовым — расстояния между ними же согласно формуле Джукса – Кантора (JC).
- Красным цветом — линия y = 0.75x (истинные расстояния)
Расстояния указаны в количестве мутаций на 100 п.н.
Работа проводилась в Microsoft Excel.
|
Итак, мы видим, что формула Джукса – Кантора даёт почти идеально верные результаты
при малых расстояниях между последовательностями, а при больших (до 200 мутаций) – небольшие отклонения.
Метод неоткорректированных расстояний даёт хорошие результаты на расстояниях до 40 нуклеотидов, а
при больших расстояниях сильно занижает результат, асциллируя при стремлении расстояния к бесконечности
у прямой y = 0.75.
