 |
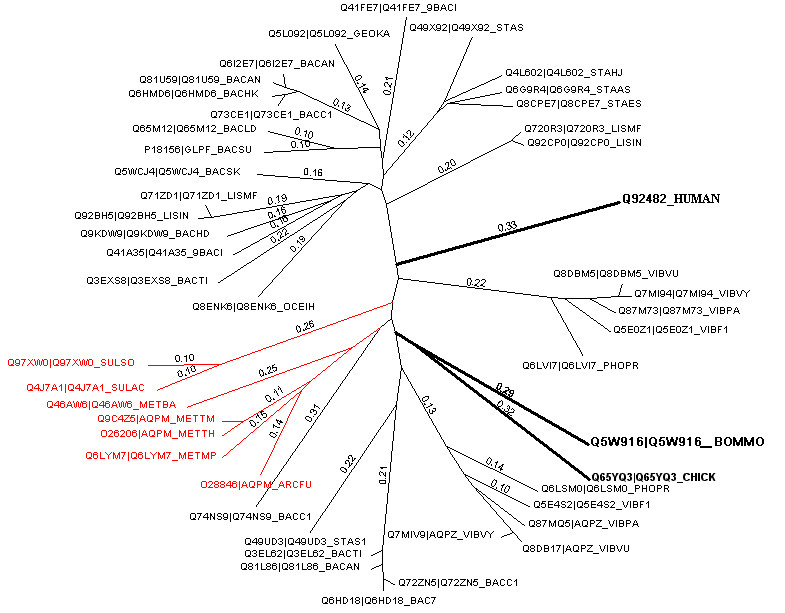
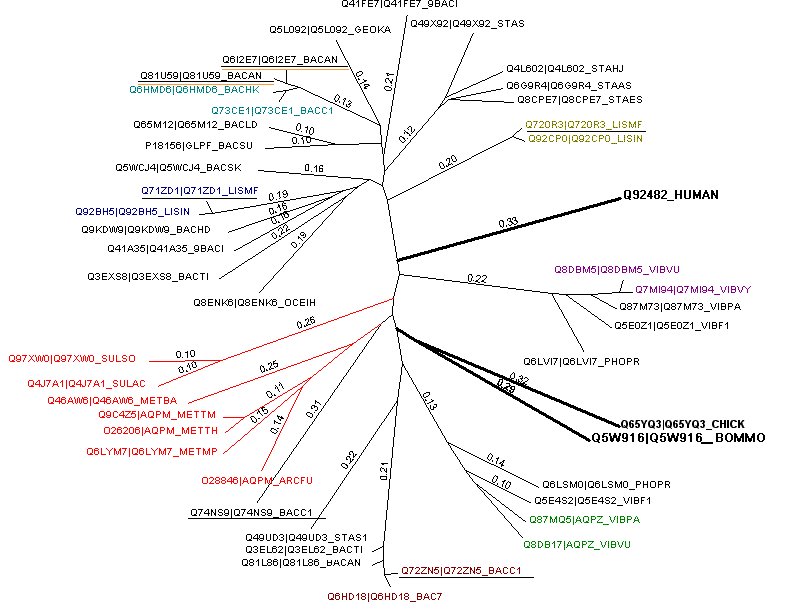
На дереве слева архейные белки выделены красным цветом , а белки аутгруппы — жирным шрифтом. Длины ветвей пропорциональны предполагаемым эволюционным расстояниям. |
Ниже приведено таксономическое дерево нашей выборки организмов, построенное на веб-сайте NCBI.
cellular organisms + Archaea + + Euryarchaeota + + + Archaeoglobi + + + + Archaeoglobus fulgidus + + + Methanobacteria + + + + Methanothermobacter thermautotrophicus + + + Methanococci + + + + Methanococcus maripaludis + + + Methanomicrobia + + + + Methanosarcina barkeri + + Crenarchaeota + + + Sulfolobus + + + + Sulfolobus acidocaldarius + + + + Sulfolobus solfataricus + Bacteria + + Firmicutes + + + Bacillales + + + + Bacillaceae + + + + + Bacillus + + + + + + Bacillus cereus group + + + + + + + Bacillus anthracis + + + + + + + Bacillus cereus + + + + + + + Bacillus thuringiensis + + + + + + + + Bacillus thuringiensis serovar israelensis + + + + + + + + + Bacillus thuringiensis serovar israelensis ATCC 35646 + + + + + + + + Bacillus thuringiensis serovar konkukian + + + + + + Bacillus clausii + + + + + + Bacillus halodurans + + + + + + Bacillus licheniformis + + + + + + Bacillus subtilis + + + + + Exiguobacterium + + + + + + Exiguobacterium sibiricum 255-15 + + + + + Geobacillus + + + + + + Geobacillus kaustophilus + + + + + Oceanobacillus + + + + + + Oceanobacillus iheyensis + + + + Listeriaceae + + + + + Listeria + + + + + + Listeria innocua + + + + + + Listeria monocytogenes + + + + Staphylococcaceae + + + + + Staphylococcus + + + + + + Staphylococcus aureus + + + + + + Staphylococcus epidermidis + + + + + + Staphylococcus haemolyticus + + + + + + Staphylococcus saprophyticus + + + + + + + Staphylococcus saprophyticus subsp. saprophyticus + + Proteobacteria + + + Vibrionaceae + + + + Photobacterium + + + + + Photobacterium profundum + + + + Vibrio + + + + + Vibrio fischeri + + + + + Vibrio parahaemolyticus + + + + + Vibrio vulnificus + Eukaryota + + Coelomata + + + Protostomia + + + + Bombyx mori + + + Deuterostomia + + + + Amniota + + + + + Sauropsida + + + + + + Gallus gallus + + + + + Mammalia + + + + + + Homo sapiens |
 |
|
Итак, на дереве выше изображены некоторые ортологи и паралоги из данного
семейства.
Конечно, далеко не все ортологи были отмечены. Ведь можно предполагать, что
все, скажем, бактериальные белки этого семейства из различных бактерий
гомологичны друг другу (относительно близкое родство, одна и та же функция
– transporter activity). Однако нами выделялись белки, удовлетворяющие 2
критериям:
1) В файле с названиями организмов ищутся повторяющиеся названия организмов, включая названия штаммов (BACILLUS THURINGIENSIS ,EXIGUOBACTERIUM SP., STAPHYLOCOCCUS SAPROPHYTICUS SUBSP. SAPROPHYTICUS ,BACILLUS ANTHRACIS, PHOTOBACTERIUM PROFUNDUM,VIBRIO VULNIFICUS, LISTERIA INNOCUA,VIBRIO FISCHERI, BACILLUS CEREUS). 2) На дереве ищутся белки, соответствующие этим организмам.
Все выше приведенные деревья были визуализированы программой GeneMaster. Программа хорошая, но ещё сыровата (например, изменить название последовательности зачастую удаётся раза с седьмого:)