Занятие 10 (зачётное)
Дано: неаннотированный фрагмент генома бактерии Yersinia mollaretii
Задача: определить, закодированы ли данном фрагменте какие-либо белки, похожие на известные белки родственной бактерии (кишечной палочки).
1) Полный протеом E. coli получим из Swiss-Prot с помощью команды
Seqret sw: *_ECOLI
получим ecoli.fasta
2) Создадим индексные файлы для поиска программами пакета BLAST
formatdb -i xc_genome.fasta -p F -n eco
3) Вырезаем нужный фрагмент генома с помощью
seqret –sask
4) Извлеките из вашего фрагмента трансляции всех открытых рамок считывания длиной не менее 240 нуклеотидов. Воспользуйтесь программой getorf из пакета EMBOSS. При этом используйте стандартный для бактерий (bacterial) генетический код, открытой рамкой считайте последовательность, начинающуюся со старт-кодона и заканчивающуюся стоп-кодоном.
getorf –find 1 –table 11
Finds and extracts open reading frames (ORFs)
Input nucleotide sequence(s): aald01000001.embl
protein output sequence(s) [aald01000001.orf]:
5) Создадим книгу Excel list.xls, включающую информацию обо всех открытых рамках считывания в данном фрагменте генома. Для каждой рамки указано: начало во фрагменте, конец во фрагменте, направление (прямое или обратное), число сходных последовательностей, найденных у E. coli при условии E-value<0,001, идентификатор самого близкого из найденных белков E. coli и E-value находки. Приведены только ненулевые результаты.
| | | | | | | |
| имя |
Обозначение на схеме |
начало |
конец |
направление |
число находок |
лучшая находка |
| >AALD01000001_1 |
Yer1 |
15 |
314 |
прямое |
3 |
YDCR_ECOLI |
3,00E-19 |
| >AALD01000001_2 |
Yer2 |
777 |
2129 |
прямое |
7 |
ASTC_ECOLI |
4,00E-167 |
| >AALD01000001_4 |
Yer4 |
2138 |
3196 |
прямое |
1 |
ASTA_ECOLI |
3,00E-121 |
| >AALD01000001_6 |
Yer6 |
3196 |
4695 |
прямое |
12 |
ASTD_ECOLI |
0.0 |
| >AALD01000001_7 |
Yer7 |
4634 |
6061 |
прямое |
1 |
ASTB_ECOLI |
7,00E-172 |
| >AALD01000001_8 |
Yer8 |
6043 |
6999 |
прямое |
1 |
ASTE_ECOLI |
3,00E-76 |
6)Схема расположения этих генов в геноме Yersinia mollaretii
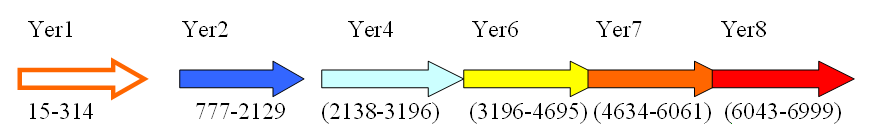
Yer1 Yer2 Yer4 Yer6 Yer7 Yer8
15-314 777-2129 (2138-3196) (3196-4695) (4634-6061) (6043-6999)
7) Составим схему лучших находок в геноме E.coli, для чего сначала соберем информацию о генах, кодирующих эти белки
| | | | | |
| Белок |
ген |
начало |
конец |
направление |
описание |
YDCR_ECOLI
|
ydcR
|
1508027
|
1509433
|
Прямое
|
fused predicted DNA-binding transcriptional regulator/predicted amino transferase; putative regulator; Not classified; |
ASTC_ECOLI
|
astC
|
1828786
|
1830006
|
Обратное
|
succinylornithine transaminase, PLP-dependent; 2.6.1.17; 2.6.1.11; enzyme; Amino acid biosynthesis: Arginine; |
| ASTA_ECOLI |
astA |
1827755 |
1828789 |
Обратное |
arginine succinyltransferase; 2.3.1.109; |
| ASTD_ECOLI |
astD |
1826280 |
1827758 |
Обратное |
succinylglutamic semialdehyde dehydrogenase |
| ASTB_ECOLI |
astB |
1824940 |
1826283 |
Обратное |
succinylarginine dihydrolase [EC:3.5.3.23] |
| ASTE_ECOLI |
astE |
1823979 |
1824947 |
Обратное |
succinylglutamate desuccinylase (EC:3.5.1.-) |
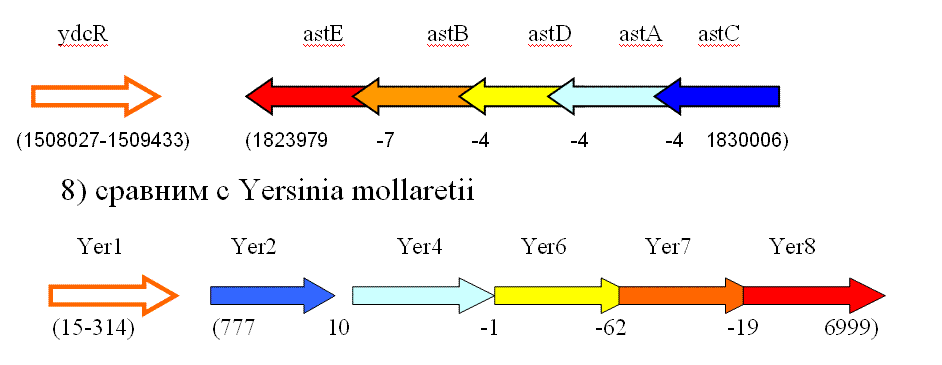
Исходя из положений и описаний генов, можно предположить, что они объединены в один оперон, при этом первый ген кодирует регуляторный белок, связывающийся с ДНК, а остальные – ферменты, участвующие в метаболизма аминокислот – аргинина и пролина.
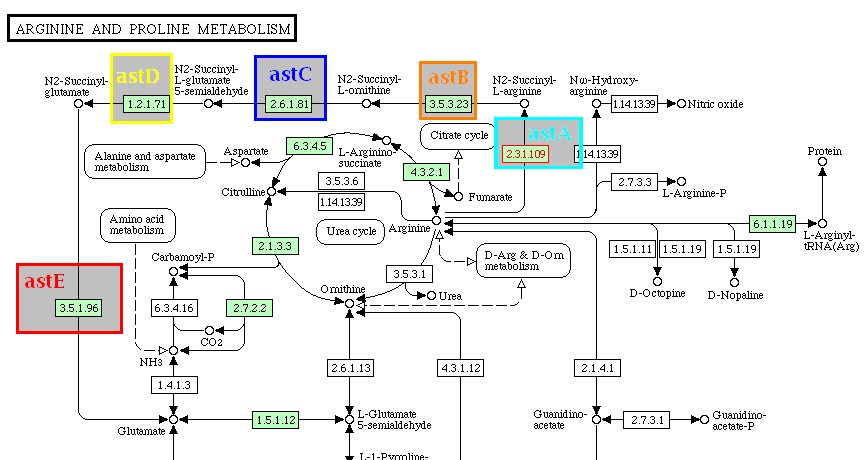
Фрагмент схемы этого процесса из базы данных KEGG
Карта генов оттуда же (тоже фрагмент)
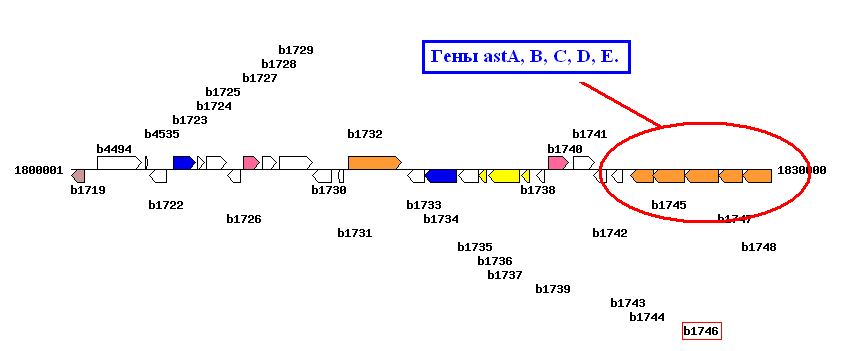
Оранжевый цвет значит группу процессов - Amino Acid Metabolism (метаболизм аминокислот)
Таким образом можно предположить наличие оперона метаболизма аргинина и пролина у Yersinia mollaretii , так как сходные 5 генов Ecoli лежат в том же порядке и перекрываются сходным образом, хотя и направлены в другую сторону. Кроме того, существует и ген регулятора этого оперона, причем в отличие от генома ecoli, здесь он сближен с генами ферментов. Перекрытия генов в данном случае объясняются тем, что продукты генов участвуют в одном процессе, непосредственно друг за другом (см. схему) и, следовательно, должны экспрессироваться согласованно.
Замечание: В процессе поиска с помощью SRS я вышла на базу данных KEGG: Kyoto Encyclopedia of Genes and Genomes, которая позволила наглядно предоставить связь как между генами (карта генома ecoli) так и между их продуктами (схема процесса). За что ей и благодарность, и ссылка
http://www.genome.jp/kegg/

