Исследование структуры тРНК
- Краткое описание структуры в файле
4TNA.pdb
В файле приведены координаты атомов молекулы тРНК для фенилаланина,
из данных об организме - только тривиальное название - yeast - дрожжи.Потому что
источник - synthetic construct, то есть искусственно синтезированная молекула.
Для исследования была выбрана цепь А (единственная), представляющая фенилаланин-тРНК со следующей последовательностью:
[1] 5' -GCGGAUUUAGCUCAGUUGGGAGAGCGCCAGACUGAAGAUCUGGAGGUCCUGUGUUCGAUCCACAGAAUUCGCACCA- 3' [76],
где 1 и 76 - номера первого и последнего нуклеотида.
На 3'-конце есть триплет CCA, к которому присоединяется аминокислота.Этот триплет есть и в PDB файле - Исследование вторичной структуры
С помощью программы find_pair пакета 3DNA были определены возможные
водородные связи
между азотистыми основаниями
Получили файл pairs.inp
В соответствии с полученными данными
акцепторный стебель состоит из участка 1-7 и комплементарного ему участка 72-66.
Т-стебель - 49-53 и 65-61
D-стебель - 10-14 и 25-21
антикодоновый стебель - 27-31 и 43-39
Структуру стеблевых дуплексов поддерживают 19 канонические и 1 неканонические пары оснований:Рис.1. Вторичная структура фенилаланин-тРНК из дрожжей
Скрипт для получения изображения select all wireframe off backbone 50 color black background white select 1-7 or 66-72 color red backbone 100 select 49-53 or 61-65 backbone 100 color green select 10-14 or 21-25 backbone 100 color blue select 27-31 or 39-43 color orange backbone 100
U-G (4-69)
Неканоническая пара G-U
Вариабельная пеля отсутствует, тимидин в Т-петле отсутствует
Дигидроуридины в D-петле (16 и 17 нуклеотиды)
Антикодон в антикодоновой петле - A-A- (35-37 нуклеотиды)
YG - WYBUTOSINE;Систематическое название 1H-IMIDAZO(1,2-ALPHA)PURINE-7-BUTANOIC ACID,4,9-DIHYDRO-ALPHA-((METHOXYCARBONYL)AMINO)- 4,6-DIMETHYL-9-OXO-METHYL ESTER.Практически, это сильно модифицированный гуанозин. Это основание соединяется с рибозой посредством стандартной N9-C1* связи.
- Исследование третичной структуры
1)Для изучения стекинг взаимодействия используем файлы, полученные с помошью команды
find pair -t 4TNA.pdb stdout | analyze
В файле 4TNA.out Находим информацию о перекрытиях оснований (поиск F7 по слову overlap). Выбираем перекрытие с наибольшей площадью, и для сравнения возьмем перекрытие с площадью поменьше.Им соответствуют структуры 13 и 3 из файла stacking.pdb. Чтобы получить картинки сначала вырезаем нужные фрагменты
ex_str -3 stacking.pdb step3.pdb
ex_str -13 stacking.pdb step13.pdb
Потом делаем из них картинки
stack2img -cdolt step3.pdb step3.ps
stack2img -cdolt step13.pdb step13.ps
Между основаниями конца акцепторного
стебля и начала Т-стебля
#0007 5MU 1MA / G A
Площадь перекрытиямежду антикодоновым и D-стеблем
#0020 G G / U C
Площадь перекрытия2)Дополнительные водородные связи между основаниями D- и T-петель
Реальная и предсказанная вторичная структура тРНК из файла 4TNA.pdb
Участок структуры
Позиции в структуре
(по результатам find_pair)Результаты предсказания
с помощью einvertedРезультаты предсказания
по алгоритму ЗукераАкцепторный стебель 5'1-7 3'
5' 66-72 3'
Всего 7 парни оной пары не предсказано из этого промежутка 6 из 7 пар D-стебель 10-13
22-25
4 парынуклеотиды 22-25 "спарены" с другой цепью все 4 правильные T-стебель 49-53
65-61
5 парвсе 5 пар правильные все 5 пар правильные Антикодоновый стебель 39-43
31-27
5 парвсе 5 пар предсказаны, есть лишние все 5 пар правильно Общее число канонических пар нуклеотидов 21 всего 15, правильно 11 всего 20 пар,все правильно Изображение, полученное с помощью mfold
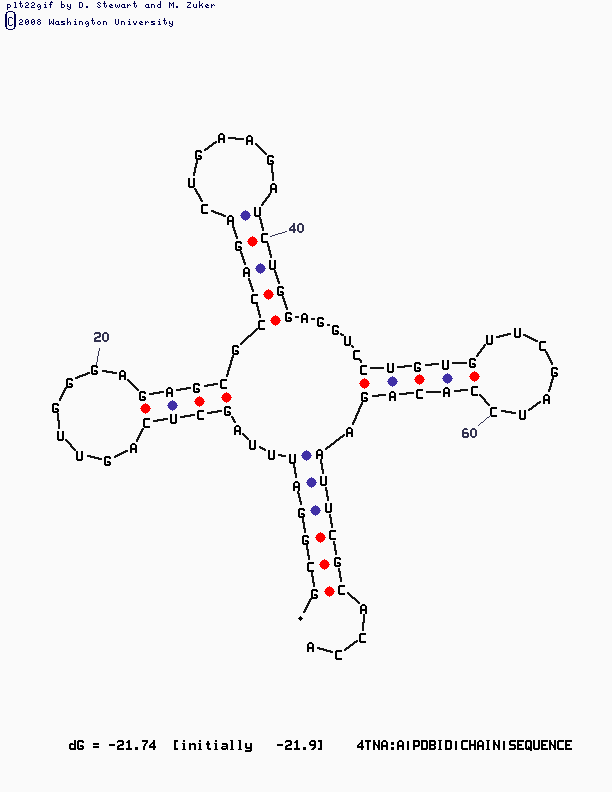
Использовались значения параметров: -для программы einverted уменьшали параметр threshold до 10, пока не появился осмысленный результат, изменение параметра gap не дало видимых результатов, другие параметры не трогали. -для программы mfolt взяли P=15, хотя если оставить этот параметр по умолчанию, все равно есть хорошая картинка, которая и представлена в отчете.