Вернуться на главную |
Страница проектов |
Заметки |
Таким образом теоретически слово fever должно встретиться 191670831*3,86*6,75*6,75*6,87*5,53*10^(-10)=128,06.
Слово fever может встречаетя в базе данных 128 раз.
Проверка встречаемости слова fever через PROSITE показала, что реально в базе встречается 101 раз.
Полученные данные говорят, что к базе белков применимы модели для случайных выборок (т.к. формула для теор.рассчета сделана исходя из этого.)
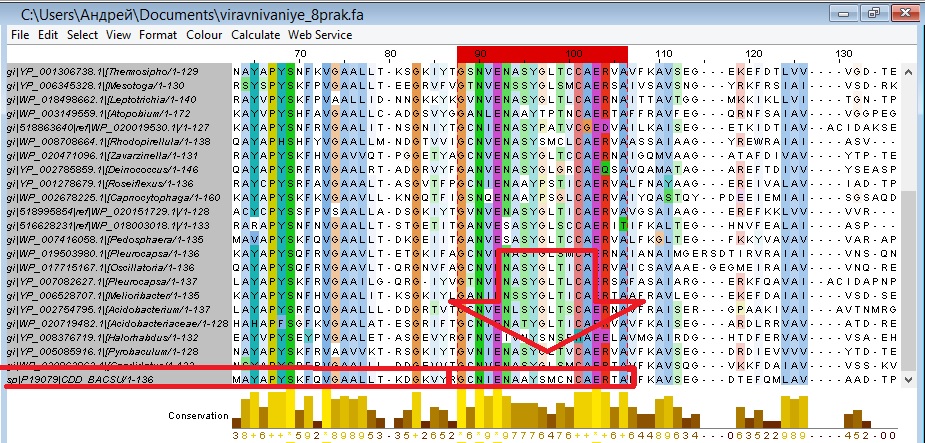
Участок выравнивания, по которому я строил паттерн:

Для поиска гомологов были составленны паттерны представленные в Таблице 1.
| Характеристика паттерна | Паттерн | В скольких последовательностях банка Swiss-Prot найден мотив, удовлетворяющий паттерну? | Все ли последовательности из множественного выравнивания найдены? | e-value паттерна |
| Сильный | G-[ACSTV]-N-x-E-[NS]-x-[AST]-x-[GPST]-x-[ACGST]-x-C-[AG]-E-[DQR]-x-[AT] | 9 | не все, это обусловлено разными базами данных, по которым велся поиск | 1.155866e-05 |
| Слабый | G-[ACSTV]-N-x-E-[NS]-x-[AST] | 163 | почти все, это обусловленно раличием баз, по которым велся поиск | 56 |
Поиск проведен через веб интерфейс, результат представлен в Таблице 2.
| Идентификатор документа Prosite (AC) | Название мотива | Краткое описание мотива | Тип подписи | Паттерн (если это паттерн) | Специфична ли подпись? | Сколько мотивов нашлось в белке? |
| PS00903 | CYT_DCMP_DEAMINASES | сайт связывания цинка | паттерн | [CH]-[AGV]-E-x(2)-[LIVMFGAT]-[LIVM]-x(17,33)-P-C-x(2,8)-C-x(3)-[LIVM] | да | 1 |
| PS00008 | MYRISTYL | Сайт N-миристоилирования | паттерн | G-{EDRKHPFYW}- x(2)- [STAGCN]-{P} | нет | 1 |
| PS00006 | CK2_PHOSPHO_SITE | Сайт фосфорилирования казеинкиназой II | паттерн | [ST]-x(2)-[DE] | нет | 3 |
| PS00007 | TYR_PHOSPHO_SITE | Сайт фосфорилирования тирозина | паттерн | [RK]-x(2)-[DE]-x(3)-Yor[RK]-x(3)-[DE]-x(2)-Y | нет | 1 |