На странице "Library page" среди доступных баз данных выбрал "UniPRotKB/Swiss-Prot".
Затем слева, в блоке "Search Options", выбрал "Standard Query Form".
В одном из полей запроса выбрал
Из полученного списка белков выбрал мой белок - CDD_BACSU.
Перешёл в раздел "Features" вверху страницы, где выбрал произвольные три особенности (например, вторую альфа-спираль, третий поворот и второй бета-тяж).

Связывание деаминазы(фермент) с цинком.
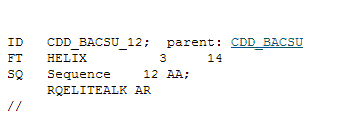
Альфа спираль, образованная 12 аминокислотными остатками(с 3 по 14).

Поворот, образованный 3 аминокислотными остатками(с 20 по 22).
2.Получение и сохранение описания всех записей банка трёхмерных структур PDB, относящихся к моему белку
Находясь на странице с информацией о белке, в левой части страницы в блоке "Entry Options" нажал на красную кнопку "Link", после чего перешёл на страницу находок.
Выбрал раздел "Protein function, structure and interaction databases", в котором отметил галочкой базу данных "PDB". Затем нажал на красную кнопку "Search" в верхней левой части страницы.
Получив список документов PDB, нажал на красную кнопку "Save" в графе "Result Options" в верхней левой части страницы.
Перейдя на страницу параметров сохранения информации, отметил "File (text)" в блоке "Output To:", после чего нажал на красную кнопку "Save" в нижней правой части страницы.
Таким образом, сохранив полученную информацию, я получил файл pdb_info_CDD_BACSU.txt, в котором записана информация о всех записях, которые были в "находках".
3.Поиск полноразмерных белков из Firmicutes, имеющих функцию, сходную с функцией белка CDD_BACSU из Bacillus subtilis
В одном из полей запроса я выбрал "Taxonomy" и внёс в окошко "Firmicutes"
В следующем поле выбрал "Comments: Comment" и стал вносить в окошко различные описания функций или отдельные слова из описания.
После чего нажал кнопку поиска "Search"
Запрос всех последовательностей
| Формулировка функции белка | Строка запроса | Количество найденных документов |
|---|---|---|
Для поиска только полноразмерных последовательностей я выбрал в новом поле запроса "Description" и записал в окошко "*!fragment"
Запрос только полноразмерных последовательностей
| Формулировка функции белка | Строка запроса | Количество найденных документов |
|---|---|---|
Полноразмерные последовательности отличаются несильно, но таково задание.
4.Сохранение полноразмерных последовательностей найденных белков в fasta формате После поиска последовательностей я вернулся к наиболее подходящему из запросов. Затем выбрал 10 последовательностей (только 10, так как находок было 1287) и сохранил их в следующем файле similar_fasta_files_CDD_BACSU.fasta.
При сохранении в разделе "Output To" я отметил "File (text)", а в разделе "Save as" вместо "UniprotView" выбирал "FastaSeqs"
5.Сохранение списка последовательностей из задания 4, в котором указаны ID, AC последовательностей, организм (Species), название белка (Description) и длина последовательности (Sequence Length)
Далее я вернулся к первому запросу (из таблицы в задании 3) и в разделе "Create view" выбрал поля: ID, AccessionNumber, "Save"
На открывшейся странице с параметрами сохранения в разделе "Output To" отметил "File (text)", а в разделе "Save As" выбрал "SWISSPROT"
Нажал на кнопку "Save" и переименовал сохранённый файл в файл table_of_similar_fasta_files_CDD_BACSU.xls .
© Прозоров Данила