Примечание: вибутозин=1H-имидазол(1,2-альфа)пурин-7-масляная кислота,4,9-дигидро-альфа-((метоксикарбонил)амино)-2,4,6-диметил-9-оксометиловый эфир. Фактически это очень сильно модифицированный гуанозин. Связан с рибозой через N9-C1* связь.
Структура
Strand I Strand II
Helix 1
1 (0.073) A:...1_:[..G]G-----C[..C]:..72_:A (0.048)
2 (0.039) A:...2_:[..C]C-----G[..G]:..71_:A (0.071)
3 (0.077) A:...3_:[..G]G-----C[..C]:..70_:A (0.035)
4 (0.084) A:...4_:[..G]G-*---U[..U]:..69_:A (0.057)
5 (0.070) A:...5_:[..A]A-----U[..U]:..68_:A (0.056)
6 (0.048) A:...6_:[..U]U-----A[..A]:..67_:A (0.068)
7 (0.061) A:...7_:[..U]Ux----A[..A]:..66_:A (0.067)
Helix 2
8 (0.043) A:..49_:[5MC]c-----G[..G]:..65_:A (0.075)
9 (0.061) A:..50_:[..U]U-----A[..A]:..64_:A (0.085)
10 (0.076) A:..51_:[..G]G-----C[..C]:..63_:A (0.038)
11 (0.053) A:..52_:[..U]U-----A[..A]:..62_:A (0.060)
12 (0.085) A:..53_:[..G]G----xC[..C]:..61_:A (0.041)
Helix 3
15 (0.049) A:..39_:[PSU]P-*---A[..A]:..31_:A (0.071)
16 (0.035) A:..40_:[5MC]c-----G[..G]:..30_:A (0.090)
17 (0.050) A:..41_:[..U]U-----A[..A]:..29_:A (0.069)
18 (0.069) A:..42_:[..G]G-----C[..C]:..28_:A (0.050)
19 (0.080) A:..43_:[..G]G-----C[..C]:..27_:A (0.044)
20 (0.086) A:..44_:[..A]Ax*---g[M2G]:..26_:A (0.086)
Helix 4
21 (0.084) A:..10_:[2MG]g-----C[..C]:..25_:A (0.047)
22 (0.038) A:..11_:[..C]C-----G[..G]:..24_:A (0.086)
23 (0.056) A:..12_:[..U]U-----A[..A]:..23_:A (0.062)
24 (0.043) A:..13_:[..C]C----xG[..G]:..22_:A (0.084)
Разными цветами выделены различные спирали (1-я синим, 2-я зеленым, 3-я сиреневым, 4-я красным)
|
G C G G A U U U A 2MG C U C A G H2U H2U G G G A G A G C M2G C C A G A OMC U OMG A A YG A PSU 5MC U G G A G 7MG U C 5MC U G U G 5MU PSU C G 1MA U C C A C A G A A U U C G C A C C A |
Вид структуры в RasMol.
Цепь тРНК в остовной модели, найденные спирали покрашены в разные цвета, при 5'-концевом и 3'-концевом нуклеотидах подписаны номера остатков.
Изображение создано с помощью следующих команд:
background white
define helix1 1-7, 66-72
define helix2 49-53, 61-65
define helix3 26-31, 39-44
define helix4 10-13, 22-25
restrict none
select *:a
backbone 100
select helix1
color blue
select helix2
color green
select helix3
color violet
select helix4
color red
select *.p and (1,76)
label %n %r
set fontsize 14
Анализ структуры тРНК
Внеспиральные стекинг-взаимодействие между основаниями:
- азот 5,6-дигидроуридин-а и кислород цитидина: h2u16a.n3 - c60a.o2, расстояние 2.896 А
- азот 7N-метил-8-гидрогуанозина и кислород гуанидина: 7mg46a.n2-g22a.o6, расстояние 2.641 А
- азот гуанозина и кислород 2N-метилгуанозина: g45a.n2-2mg10a.o6, расстояние 2.699
Примеры водородных связей между основаниями, не сводящихся к Уотсон-Криковскому спариванию комплементарных оснований:
- гуанозин с уридином: g4 и u69;
- 5-метилуридин с 1N-метиладенозином: 5mu55 и 1ma58;
- псевдоуридин с гуанозином: psu55 и g18;
- псевдоуридин с аденозином: psu39 и a31;
- аденозин с N2-метилгуанозином: a44 и m2g26;
- аденозин с уридином: u8 и a14, но расположены друг относительно друга так, что взаимодействуют атомы u8a.o4 и a14a.n7 (3.336 A), u8.o2 и a14a.n6 (2.99 A), то есть взаимодействие не по стандартой схеме;
- гуанозин с цитидином: g15 и c48, основания смещены друг относительно друга и взаимодействуют атомы g15a.n1-c48a.o2 (3.09 A);
- уридин с аденозином: u33 и a36, взаимодействуют u33a.o2-a36a.n7, u33a.n3-a36a.o2p, где последний указанный атом - кислород остатка фосфорной кислоты, то есть это не взаимодействие основание-основание.
С помощью конвеера find_pair | analyze была получена таблица:
Minor Groove Major Groove
P-P Refined P-P Refined
1 GC/GC --- --- --- ---
2 CG/CG --- --- --- ---
3 GG/UC 17.3 --- 16.2 ---
4 GA/UU 16.8 15.2 16.3 13.0
5 AU/AU 17.6 15.5 14.9 12.2
6 UU/AA 17.8 16.0 16.7 13.8
7 Uc/GA 16.7 15.2 17.9 15.5
8 cU/AG 16.8 15.3 18.0 17.1
9 UG/CA 17.2 16.0 15.1 12.0
10 GU/AC 16.9 15.6 15.3 12.1
11 UG/CA 16.4 --- 14.2 ---
12 Gu/aC --- --- --- ---
13 uP/Ga --- --- --- ---
14 PP/AG --- --- --- ---
15 Pc/GA --- --- --- ---
16 cU/AG --- --- --- ---
17 UG/CA 16.7 --- 15.5 ---
18 GG/CC 18.8 15.9 14.0 7.5
19 GA/gC 19.1 15.0 13.0 10.1
20 Ag/Cg 17.3 14.5 11.5 10.0
21 gC/GC 17.2 15.5 11.0 10.8
22 CU/AG 17.6 16.5 15.3 10.0
23 UC/GA 16.9 --- 8.6 ---
24 CA/UG --- --- --- ---
25 AG/CU --- --- --- ---
26 GG/CC --- --- --- ---
27 GU/AC --- --- --- ---
Из данной таблицы мы видим, что ширина малой бороздки больше, чем ширина большой, из чего можно сделать вывод, что данная РНК похожа на A-форму ДНК.
Работа с программой einverted из пакета EMBOSS.
Программа была запущена с параметром "Minimum score threshold" равным 10, в результате нашлись 2 спирали:
Score 16: 8/10 ( 80%) matches, 0 gaps
22 gagcgccaga 31
|| | |||||
48 ctggaggtct 39
Score 15: 5/5 (100%) matches, 0 gaps
49 ctgtg 53
|||||
65 gacac 61
Вторая спираль совпадает с найденной мной (у меня она называется Helix 2), первая спираль не совпадает ни с одной из ранее найденных. Программа нашла меньше спиралей, чем приведено выше. Для работы прогараммы в fasta-файле пришлось заменить модифицированные основания на наиболее близкие к ним канонические. Т.к. прграмма einverted работает лишь с последовательностью нуклеотидов, а не с пространственной структурой, она не может учесть неканонические взаимодействия, поэтому находит не все существующие спирали.
Анализ структуры с помощью программы mfold
Программа была запущена с параметром P= 15, в fasta-файле модифицированные основания заменены на наиболее близкие к ним канонические. Максимально близкое к реальности изображение структуры было вторым.
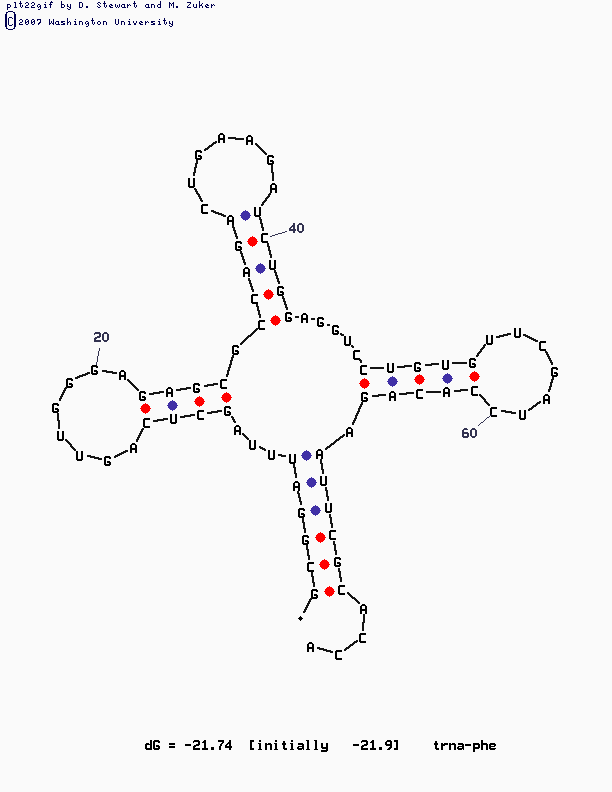
В этом случае программа работает только с немодифицированными основаниями и учитывает канонические взаимодействия, а в данной РНК много модифицированных оснований, поэтому предсказание структуры не очень близко к реальности.
© Даниленко Светлана