Ключевые слова : Геном; Протеом; Электронные таблицы; Pseudoalteromonas; Pseudoalteromonas phenolica.
Pseudoalteromonas phenolica — вид аэробных грамотрицательных палочковидных бактерий, обитающих в толще морских вод. [2] Представители не так давно открытого рода Pseudoalteromonas известны прежде всего своей способностью продуцировать различные высокоактивные вещества, обладающие альгицидными, антибактериальными, противовирусными и многими другими интересными свойствами. [1]
Рассматриваемая мною бактерия Pseudoalteromonas phenolica не является исключением, и вырабатываемые ею фенольные соединения проявляют антибактериальную активность по отношению к метициллин-резистентному золотистому стафилококку (S. aureus), который представляет опасность здоровью людей из-за своей резистентности к большинству наиболее распространенных антибиотиков. [2][4]
Генетический аппарат бактерии представ лен двумя хромосомами. Размер генома составляет 4868993 пар оснований с общим количеством генов — 4310. [3]
Электронные таблицы:
1. Сортировка ячеек в соответствии с условием (возрастание/убывание);
2. Разделение импортированного текста по столбцам;
3. Применение функции ВПР;
4. Фильтр строк;
5. Распространение формул по таблице;
6. Корректная ссылка на другие ячейки и листы (с использованием символов ‘$’ и ‘!’);
7. Использование функции СЧЁТЕСЛИ для под счета ячеек, удовлетворяющих определённым условиям (или СЧЁТЕСЛИМН, если условий больше одного);
8. Построение диаграмм и графиков;
9. Использование функций СРЗНАЧ, СУММ, МАКС, МИН.
10. Удаление повторяющихся значений; 11. Транспонирование.
Linux:
1. wordcount -wordsize 1: подсчёт числа каждого из нуклеотидов;
2. wordcount -wordsize 2: подсчёт числа каждого из 2-меров;
3. cbcalc -s 2mers -K: подсчёт O/E ratio для 2- меров.
| genomic_accession | length |
| NZ_CP013187.1 | 3845520 |
| NZ_CP013188.1 | 1023473 |
| Total | 4868993 |
| Gene type | Chromosome I | Chromosome II | Total count |
| protein_coding | 3363 | 790 | 4153 |
| rRNA | 18 | 10 | 28 |
| tRNA | 67 | 46 | 113 |
| pseudogene | 16 | 1 | 17 |
| RNase_P_RNA | 1 | 0 | 1 |
| ncRNA | 1 | 0 | 1 |
| tmRNA | 1 | 0 | 1 |
| SRP_RNA | 1 | 0 | 1 |
| Total: | 3468 | 847 | 4315 |
На основе полученных данных можно заметить, что в геноме Pseudoalteromonas phenolica из азотистых оснований в наибольшей степени представ лен тимин (1454756 н.), в наименьшей— цитозин (974560 н.). Причём именно такое распределения по распространённости в последовательности ха рактерно для обеих хромосом. Точные числовые данные представлены в Таблице 3.
Также важно отметить, что в процентном соотношении аденин встречается почти так же часто, как и тимин, а цитозин, соответственно, как гуанин. Это свидетельствует о том, что выполняется второе правило Чаргаффа. Точные процентные соотношения азотистых оснований представлены на Рисунке 1.
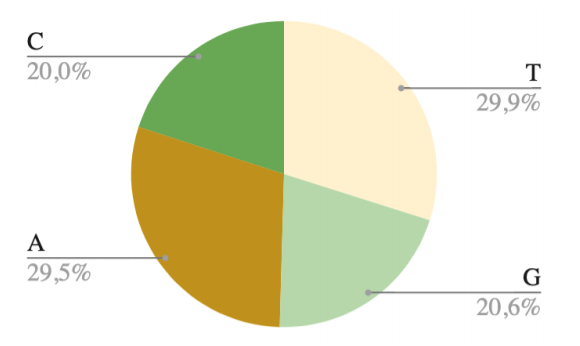
| Nucleotide | Chromosome I | Chromosome II | Total |
| T | 1142776 | 311980 | 1454756 |
| A | 1134313 | 304089 | 1438402 |
| G | 786955 | 214320 | 1001275 |
| C | 781476 | 193084 | 974560 |
На Рисунке 2 представлена гистограмма длин белков P.phenolica. Наиболее представленным диапазоном длин является значение 100-200 аминокислотных остатков, причём средняя длина белка составляет около 341 а.о., что слегка превосходит среднее значение для бактерий в целом (320 а.о.). [6]
Самым длинный белок — синтетаза нерибосомных пептидов (6705 а.о.), самый короткий — фермент транспозаза (30 а.о.).
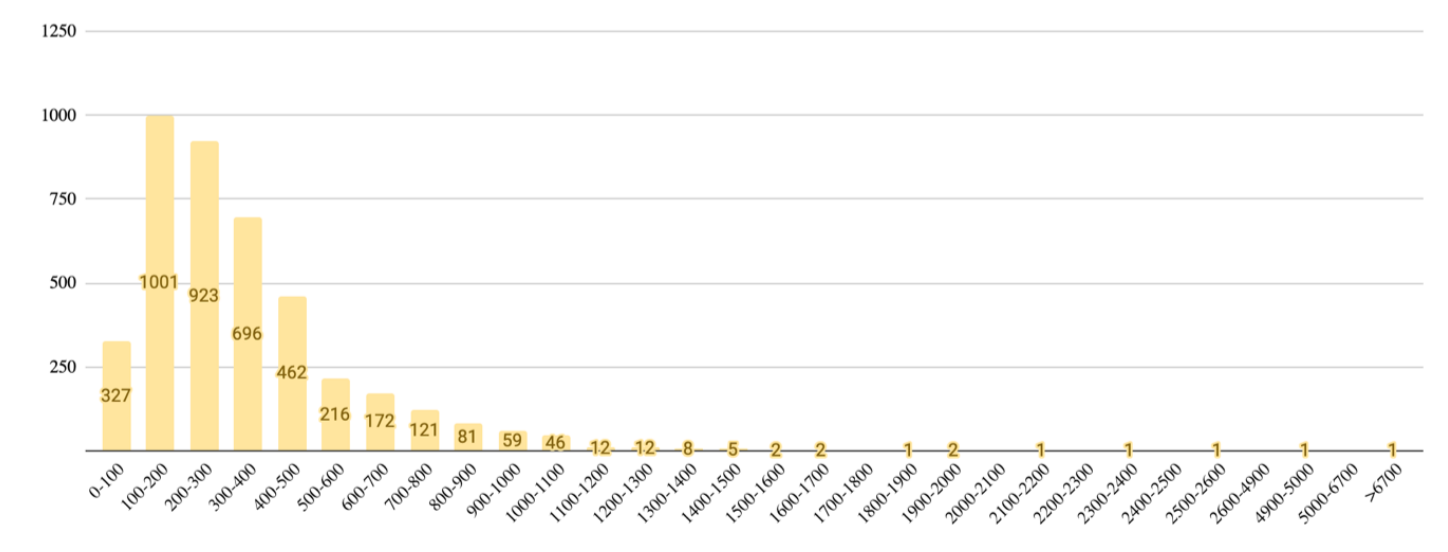
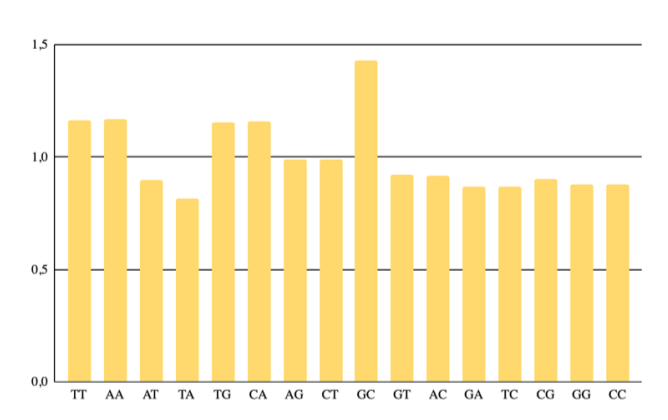
Диаграмма на Рисунке 3 отображает отношение числа фактически представленных 2-меров в нуклеотидной последовательности к их математически ожидаемой величине. Хорошо видно, что сильно превосходит ожидаемое значение число следующих 2-меров: GC, TG, CA, TT, AA (>1,2), они перепредставлены в геноме. Причём интересно то, что 2-мер GC, наиболее всех превосходящий единицу, составлен из наименее представленных в геноме нуклеотидов (см. раздел 4.3)
Наиболее приближено к ожидаемому число AG, CT (чуть меньше 1). Представленность же остальных 2-меров в более значительной степени ниже ожидаемой, но ни один из показателей не опускается ниже отметки 0,8.
В ходе проделанной работы были рассмотрены, проанализированы и обобщены важные основополагающие аспекты генома и протеома Pseudoalteromonas phenolica.
Так, были исследованы нуклеотидный состав, распространенность 2-меров, диапазон распределения длин белков, встречаемость различных типов генов. Геном бактерии был проверен на соответствие второму правилу Чаргаффа.
Дальнейшее исследование Pseudoalteromonas phenolica может быть полезно в связи с её биотехнологическим потенциалом, связанным с продуцированием антибактерицидных веществ против патогенных представителей Staphylococcus aureus.
Директория c файлами, откуда были взяты последовательность нуклеотидов и иная информация о геноме
Электронная таблица в Google Sheetsprotein_length: расчёты для построения гисто- граммы длин белков
gene2: информация о белок-кодирующих генах
2mers_analysis: расчёты для построения гистограммы O/E ratio 2-меров
genome_size: расчёты длин последовательностей
genes_per_types: подсчёт числа генов по категориям
gene_types_per_chromosome: подсчёт числа генов по категориям в отдельных хромосомах
nucleotides_count: данные по числу каждого из нуклеотидов, построение круговой диаграммы
genes: сводная таблица, использующаяся во всех вышеперечисленных.
1. Carola Holmström, Staffan Kjelleberg, Ma- rine Pseudoalteromonas species are associ- ated with higher organisms and produce bio- logically active extracellular agents, FEMS Mi- crobiology Ecology, Volume 30, Issue 4, De- cember 1999, Pages 285– 293, https://doi.org/10.1111/j.1574- 6941.1999.tb00656.x
2. Isnansetyo A., Kamei Y. ( 2003 ). Pseudoalt- eromonas phenolica sp. nov., a novel marine bacterium that produces phenolic anti-methi- cillin-resistant Staphylococcus aureus sub- stances. . Int J Syst Evol Microbiol 53, 583– 588, https://doi.org/10.1099/ijs.0.02431-0
3. BioCyc: общие сведения о Pseudoalter- omonas phenolica и её геноме https://biocyc.org/organism-summary?ob- ject=GCF_001444405
4. Siddiqui A., Koirala J., Methicillin Resistant Staphylococcus Aureus, 2020, электронная статья на сайте NCBI, Books https://www.ncbi.nlm.nih.gov/books/NBK48 2221/
5. diCenzo GC, Finan TM. 2017. The divided bacterial genome: structure, function, and evolution. Microbiol Mol Biol Rev 81:e00019- 17. https://doi.org/10.1128/MMBR.00019- 17 .
6. Tiessen, A., Pérez-Rodríguez, P. & Delaye-Ar- redondo, L.J. Mathematical modeling and comparison of protein size distribution in dif- ferent plant, animal, fungal and microbial spe- cies reveals a negative correlation between protein size and protein number, thus provid- ing insight into the evolution of proteo- mes. BMC Res Notes 5, 85 (2012). https://doi.org/10.1186/1756-0500-5-85