Клюбина Д.С.
Московский государственный университет имени М.В. Ломоносова факультет Биоинженерии и биоинформатики
Аннотация
В рамках данного мини-обзора рассматривается распределение белков, закодированных в геноме Citrobacter freundii, по их длинам, частота встречаемости старт-кодонов и антикодонов аминокислот в генах, кодирующих тРНК.
Введение
Бактерия Citrobacter freundii принадлежит к роду Citrobacter, семейству Enterobacteriaceae, отряду Enterobacterales, классу Gammaproteobacteria, типу Pseudomonadota [1].
Вид грамотрицательных, споронеобразующих, факультативно анаэробных бактерий. Citrobacter freundii обитает в толстом кишечнике человека, но при попадании в другие органы или при нарушении баланса микрофлоры может вызвать ряд инфекций [2]. Род Citrobacter был открыт в 1932 году Веркманом и Гилленом. Культуры C. freundii были выделены и идентифицированы в том же году из почвенных экстрактов [3].
Материалы и методы
Последовательности белок-кодирующих участков и таблицу локальных особенностей генома были взяты в National Center for Biotechnology Information [4].
Для создания гистограммы, изучения частоты встречаемости антикодонов в генах, кодирующих тРНК, и встречаемости старт-кодонов я использовала методы Google sheets (см. D, E, F сопроводительных материалов).
Для изучения встречаемости старт-кодонов были использованы ещё методы bash (см. коды A, B, C сопроводительных материалов).
Результаты
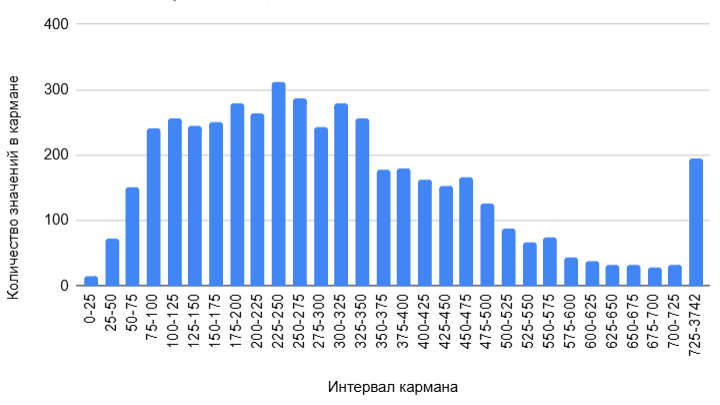
— Длины белков, закодированных в геноме бактерии Citrobacter freundii
По полученному распределению на рис.1 видно, что длины белков широко варьируются от самых маленьких (до 25 аминокислотных остатков) до довольно больших (3742 остатка) цепей. Наиболее распространены белки с длинной 50-500. Самая распространённая длина - 225-250 аминокислотных остатков.

—Частота встречаемости разных старт-кодонов в геноме Citrobacter freundii
Полученное количество старт-кодонов (см. F сопроводительных материалов) показывает, что всего 7 вариантов триплетов из 64 возможных могут быть старт-кодонами. Наиболее распространённый вариант: ATG. Помимо 7 вариантов, встречающихся в нормальных CDS, есть ещё 20 вариантов триплетов, с которых начинаются псевдогены.
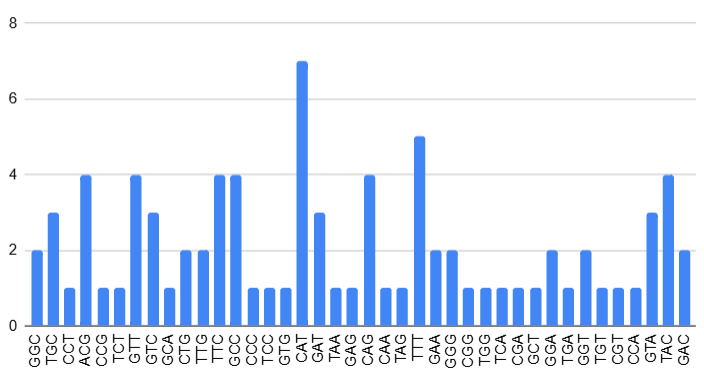
—Количество генов, кодирующих тРНК с соответствующими антикодонами
Из рис.2 видно, что количество закодированных тРНК с разными антикодонами варьируется от 1 до 7. Т.е. для каких-то антикодонов существует всего один ген, в котором он закодирован, а для других 4 или даже 7. Наиболее часто закодирован CAT, который соответствует аминокислоте изолейцин. Причем связи между количеством антикодонов, кодирующих какую-то аминокислоту, и их встречаемостью на данном этапе изучения установить не удаётся.
Сопроводительные материалы
Код для всех CDS
grep -A 1 '>' *_cds_from_genomic.fna | grep -v '-' | grep -v '>' | cut -c '1-3' | sort | uniq -c
Программа получает на вход таблицу CDS в геноме бактерии и выводит количество уникальных старт-кодонов для всех CDS.
Код для CDS-псевдогенов
grep -A 1 'pseudo=true' *_cds_from_genomic.fna | grep -v '-' | grep -v '>' | cut -c '1-3' | sort | uniq -c
Программа получает на вход таблицу CDS в геноме бактерии, отбирает соответствующие псевдогенам и выводит количество уникальных старт-кодонов для CDS-псевдогенов.
Код для всех кодонов
echo {A..D}{A..D}{A..D} | tr 'BD ' 'GT\n'
Программа выводит все возможные варианты триплетов.
Литература