Практикум 10: работа с программой BLAST
1. Поиск гомологов белка Xanthomonas cucurbitae в Swiss-Prot
Для поиска гомологов был выбран белок бактерии Xanthomonas cucurbitae шаперон DnaK (Chaperone protein DnaK), имеющий в базе UniProt ID A0A2S7DYB4_9XANT. Использовались следующие значения параметров BLAST:
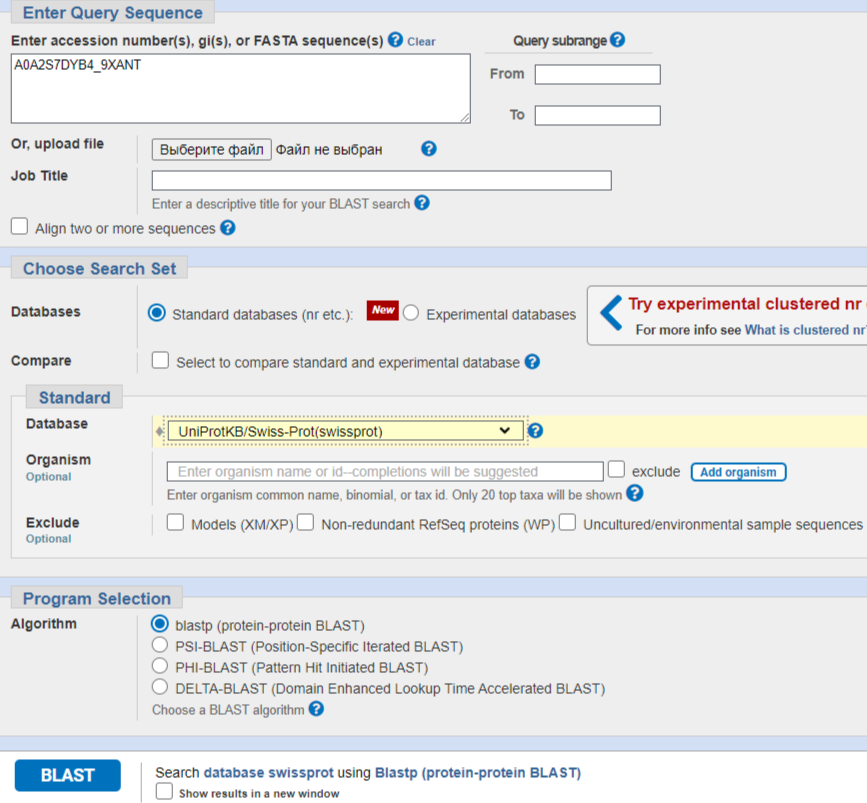
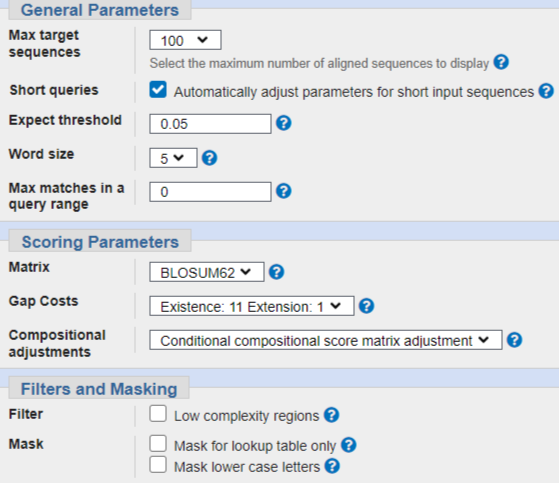
Для данного белка в базе SwissProt было найдено более 100 гомологов - cсылка на текстовый файл с выдачей.
Из находок были выбраны 7 белков (cсылка на текстовый файл с выбранными белками). С помощью программы множественного выравнивания Muscle в визуализаторе JalView было произведено выравнивание 7 находок с исследуемым белком
С помощью программы Muscle в визуализаторе JalView было построено выравнивание, представленное поссылке. Все из последовательностей оказались гомологичны друг другу по всей длине выравнивания, отмечается высокая степень консервативности большинства позиций
2. Поиск гомологов зрелого вирусного белка, вырезанного из полипротеина
В базе данных Swiss-Prot для поиска вирусных полипротеинов был введен поисковой запрос (taxonomy_id:10239) AND (protein_name:polyprotein) AND (reviewed:true). Среди полученных результатов был выбран полипротеин Envelopment polyprotein вируса New York virus (NYV) (AC: Q83887, ID: GP_NYV). Этот полипротеин разделяется на 2 белка, имеющих координаты 18-652 и 653-1140. Мною был выбран белок (18:652). Этот белок был вырезан из полипротеина средствами командной строки с помощью следующей программы: descseq 'sw:GP_NYV[18:652]' -outseq chain1.fasta . Последовательность вырезанного белка размещена по ссылке
Последовательность вырезанного белка был дана на вход программе BLAST с теми же параметрами, что и в пункте 1 выше. Найденные программой гомологичные последовательности (их 18) можно посмотреть по ссылке. Из них были выбраны 4 находки, их AC: P08668, Q806Y7, P16493, P33455. С помощью JalView было построено множественное выравнивание этих 4 последовательностей и последовательности вырезанного белка программой Muscle (проект JalView с выравниванием зрелых белков доступен по ссылке).
По результатам выравнивания можно видеть высокую степень сходства последовательностей по всей длине выравнивания.
3. Исследование зависимости E-value от объёма банка
Когда при поиска гомологов для вырезанного белка в BLAST диапазон организмов был сужен до вирусов, число находок не изменилось (те же 22 находки, выдачу можно посмотреть здесь). Однако изменились значения E-value для тех находок, у которых оно изначально не было равно нулю. Так, для белка A6XIP3 вируса Hazara virus (isolate JC280) при поиске в базе всех организмов Е-value составляло 1e-04, а при поиске только среди вирусных последовательностей 4e-06. Доля вирусных белков равна отношению E-value для поиска среди вирусов к E-value для поиска среди всех таксонов: (4e-06/1e-04)*100% = 4%.