Практикум 8: Нуклеотидный BLAST
1. Поиск гена, кодирующего дельта-субъединицу АТФ-синтазы
В прошлом практикуме я работала с геномной сборкой организма Arabidopsis thaliana, и в данном практикуме работа с этой сборкой была продолжена. В скачанном файле геномной сборки был найден белок, аннотированный как дельта-субъединица АТФ-синтазы. Идентификатор этого белка NP_192703.1, последовательность белка в формате fasta-файла можно посмотреть по ссылке.
Идентификатор нуклеотидной записи гена, кодирующего данный белок (Gene ID): 826551, последовательность данного гена в формате fasta доступна по ссылке.
2. Проведение разных вариантов BLAST для фрагментов ДНК
Далее мне необходимо было потренироваться в поиске последовательностей белков или ДНК, сходных с последовательностью гена дельта-субъединицы АТФ-синтазы Arabidopsis thaliana. Для этого нужно было выбрать группу организмов, отдаленную от систематической группы, к которой относится Arabidopsis thaliana. Мною было выбрано семейство Кошачьи (Felidae) класса Млекопитающие.
В первой части задачи можно было воспользоваться на выбор программами blastn / megablast. Я выбрала программу blastn, поскольку она выдает больше находок, чем megablast. Параметры blastn и выдача показаны на снимках экрана ниже. Я ожидала увидеть небольшое число находок, и мои ожидания оправдались - алгоритм выдал 4 находки, причем у всех из них высокий e-value, поэтому эти находки можно даже не рассматривать всерьез.
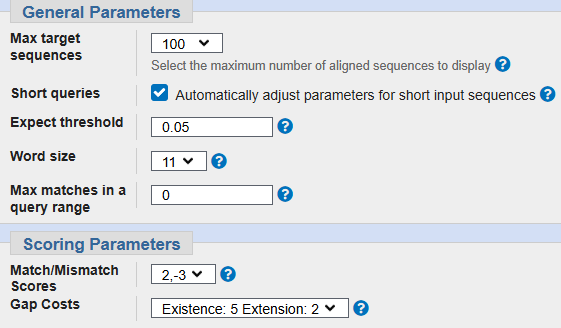
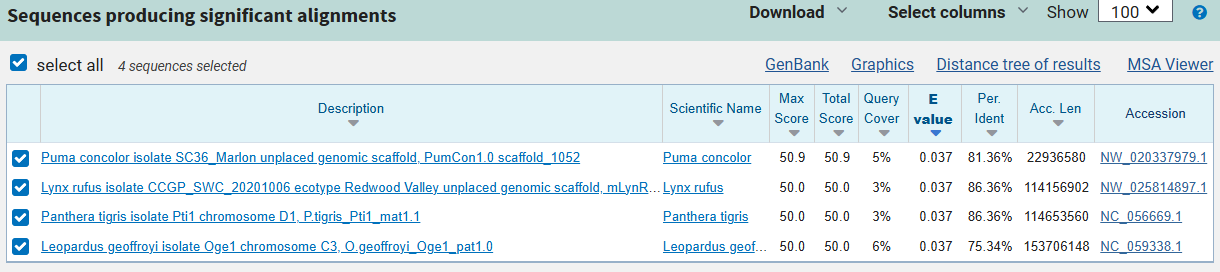
Во второй части задачи можно было воспользоваться на выбор программами tblastn \ tblastx. Я выбрала программу tblastn, предназначенную для поиска сходных белковых последовательностей. Выбор tblastn был продиктован тем, что в отличие от tblastx, которая на вход принимает ген, tblastn берет именно белковую последовательность, что позволяет более точно оценить именно сходство белковых последовательностей. При запуске tblastn с длиной слова 5 и expect threshold 0,05 (параметры по умолчанию) находок найдено не было, поэтому далее значение длины слова было уменьшено до 2, а expect threshold был изменен на 0,1. Параметры tblastn (после указанных изменений) и выдача показаны на снимках экрана ниже.
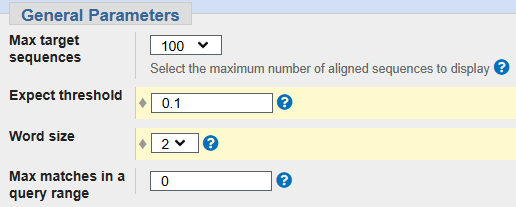
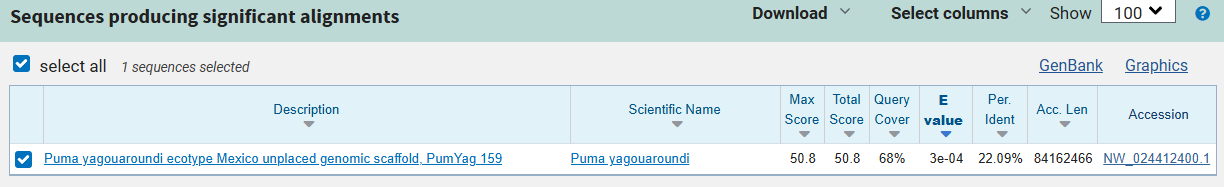
Я ожидала, что tblastn обнаружит больше находок, так как митохондриальная АТФ-синтаза - всё-таки белок консервативный, и его последовательность должна быть во многом схожа у эукариот. Однако, возможно, дело в том, что биохимические особенности митохондрий растений и животных довольно сильно отличаются, поэтому и схожести в данной субъединице АТФ-синтазы между растительным и животными организмами почти не нашлось.
3. Поиск в геноме эукарита основных рибосомальных РНК по далекому гомологу
Из файла были скачаны последовательности рибосомальных РНК (далее - рРНК) бактерии Escherichia coli. В файле приведены две последовательности, соответствующие 16S рРНК и 23S рРНК. Оба этих типа рРНК играют в составе рибосомы структурную роль, определяя положение рибосомальных белков. 16S рРНК входит в состав малой субъединицы рибосомы, а 23S входит в состав большой субъединицы рибосомы. 16S рРНК взаимодействует с 23S рРНК, способствуя связыванию большой и малой рибосомальных субъединиц. Также 16S рРНК содержит последовательность, с помощью которой 16S рРНК связывается с мРНК. В составе эукариотических рибосом гомологом бактериальной 16S рРНК является 18S рРНК, а гомологом бактериальной 23S рРНК является 28S рРНК.
Целью данной задачи практикума было найти в геноме Arabidopsis thaliana последовательности, схожие с последовательностями данных рРНК Escherichia coli с помощью средств BLAST. Вначале были проведена подготовка: последовательности 16S рРНК и 23S рРНК были скопированы в файлы '16S_rRNA_ecoli.fasta' и '23S_rRNA_ecoli.fasta' соответственно, а геном Arabidopsis thaliana (файл 'genomic.fna') был проиндексирован с помощью команды makeblastdb -in genomic.fna -dbtype nucl.
Для поиска сходных последовательностей был выбран blastn, поскольку работы с белковыми последовательностями задача не предлолагала (исключается использование blastp, blastx и tblastx), а сходство эукариотических и прокариотических последовательностей обещало быть небольшим (использование megablast может не дать желаемых результатов). Использовались две команды:
blastn -task blastn -query 16S_rRNA_ecoli.fasta -db genomic.fna -out res_16s -outfmt 7 - для поиска последовательностей, сходных с 16S рРНК
blastn -task blastn -query 23S_rRNA_ecoli.fasta -db genomic.fna -out res_23s -outfmt 7 - для поиска последовательностей, сходных с 23S рРНК
Были получены следующие выдачи для 16S рРНК и 23S рРНК
В выдаче для 16S рРНК оказались 19 последовательностей. Из них были отобраны последовательности с достаточно низким e-value (не выше 0,001) - всего 10 находок. Чтобы понять, какие из них являются гомологами, каждая из находок была проверена в геномном браузере. Из этих 10 находок гомологами являются 7. Процесс их поиска я описала по ссылке.
В выдаче для 23S рРНК оказались 36 последовательностей. Из них были отобраны последовательности с достаточно низким e-value (не выше 0,05) - всего 26 находок. Из них были отобраны последовательности с достаточно высоким e-value (не выше 0,001), их оказалось 20. Из этих значимых последовательностей гомологичными нашему запросу можно считать 15. Описание того, почему я считаю эти последовательности гомологами, можно найти по ссылке.
Источники информации для пункта 3
1. Статья из Википедии про 60S рРНК
2. Схема строения бактериальных и эукариотических рибосом с портала Research Gate
3. Обзорная статья из Википедии про рибосомы
4. Лекции для студентов Факультета Биоинженерии и Биоинформатики по дисциплине "Основы молекулярной биологии"