Практикум 7. Трансмембранные белки
Задание 1. Сравнение предсказаний трансмембранных участков в бета-листовом белке
Цель задания: сравнить результаты предсказаний трансмембранных участков по последовательности (с помощью DeepTMHMM) и по трехмерной структуре (база данных OPM) в β-листовом белке.
В базе данных OPM был выбран белок из типа Transmembrane, класса Beta-barrel transmembrane, суперсемейства Sugar porins (n=18,S=22), семейства Malptoporin-like proteins. Название белка - Sucrose-specific porin (сахарозо-специфичный белок), он находится во внешней мембране Грам-отрицательной бактерии Salmonella enterica. Идентификаторы в PDB и UniProt - 1a0s и SCRY_SALTM соответственно. Примечательно, что в PDB название бактерии отображается как Salmonella enterica subsp. enterica serovar Typhimurium, а в Uniprot - Salmonella typhimurium. Судя по описанию в Uniprot, это белок-порин, ответственный за поглощение клеткой сахарозы.
Координаты трансмембранных учатков приведены в OPM:
1(75-86),2(116-124),3(136-145),4(162-167),5(181-186),6(208-213),7(223-230),8(244-253),9(256-263),10(287-296),11(306-314),12(336-344),13(351-360),14(374-383),15(389-398),16(417-426),17(438-447),18(473-482)
Из UniProt была скачана последовательность белка в формате fasta. Для этой последовательности была запущена программа DeepTMHMM, в результате которой было получено представленное ниже графическое изображение.
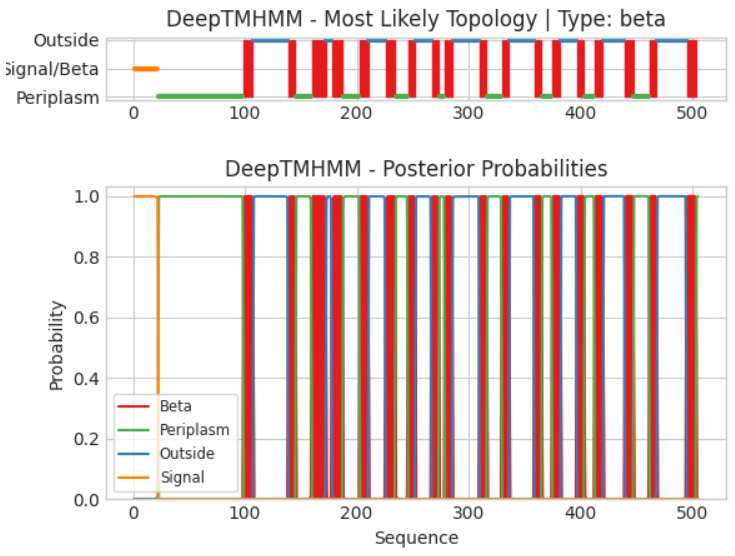
Из данного изображения видно, что наш белок имеет пронизывающие мембрану участки (показаны красными линиями) и небольшую локализованную в мембране сигнальную часть (показана желтым). Также у белка есть участки, локализованные в периплазме (показаны зеленым) и снаружи клетки (показаны синим). Файл с результатами предсказаний DeepTMHMM доступен по ссылке.
Как трансмембранные были предсказаны участки 1(99-107), 2(139-145), 3(160-172), 4(178-187), 5(203-210), 6(226-234), 7(246-252), 8(267-273), 9(279-285), 10(310-316), 11(330-336), 12(359-365), 13(375-382), 14(397-403), 15(413-420), 16(440-447), 17(462-468), 18(495-503). Т.е. всего предсказано 18 трансмембранных участков.
Таблица сравнения трансмембранных участков, приведенных в OPM и найденных DeepTMHMM, приведена ниже:
| Номер участка | OPM | DeepTMHMM |
|---|---|---|
| 1 | 75-86 | 99-107 |
| 2 | 116-124 | 139-145 |
| 3 | 136-145 | 160-172 |
| 4 | 162-167 | 178-187 |
| 5 | 181-186 | 203-210 |
| 6 | 208-213 | 226-234 |
| 7 | 223-230 | 246-252 |
| 8 | 244-253 | 267-273 |
| 9 | 256-263 | 279-285 |
| 10 | 287-296 | 310-316 |
| 11 | 306-314 | 330-336 |
| 12 | 336-344 | 359-365 |
| 13 | 351-360 | 375-382 |
| 14 | 374-383 | 397-403 |
| 15 | 389-398 | 413-420 |
| 16 | 417-426 | 440-447 |
| 17 | 438-447 | 462-468 |
| 18 | 473-482 | 495-503 |
Сравнивая результаты OPM и DeepTMHMM, можно отметить, что несмотря на совпадение количеств найденных участков (18) их координаты не совпадают. DeepTMHMM не "отловил" первый трансмембранный участок из OPM, и далее координаты найденных DeepTMHMM участков опережают координаты соответствующих для OPM, несмотря на то что некоторые из этих участков совпадают (например, 2 и 7 участок OPM почти полностью совпадает с 3 и 8 участками DeepTMHMM соответственно). Из графического изображения DeepTMHMM видно, что примерно первые 20 аминокислот последовательности определены как сигнальный пептид. В трансмембранных белках сигнальный пептид - это последовательность на N-конце белка, направляющая белок в мембрану, которая затем отрезается от белка. Скорее всего, наблюдаемый результат (смещение координат DeepTMHMM относительно координат OPM) связан с тем, что в OPM хранится именно зрелый белок, у которого сигнальный пептид уже отрезан.
Вычтем 20 из координат участков DeepTMHMM, чтобы оценить, насколько эти "поправленные" координаты совпадают с координатами OPM.
| Номер участка | OPM | DeepTMHMM |
|---|---|---|
| 1 | 75-86 | 79-87 |
| 2 | 116-124 | 129-125 |
| 3 | 136-145 | 140-152 |
| 4 | 162-167 | 158-167 |
| 5 | 181-186 | 183-190 |
| 6 | 208-213 | 206-214 |
| 7 | 223-230 | 226-232 |
| 8 | 244-253 | 247-253 |
| 9 | 256-263 | 259-265 |
| 10 | 287-296 | 290-296 |
| 11 | 306-314 | 310-316 |
| 12 | 336-344 | 339-345 |
| 13 | 351-360 | 355-362 |
| 14 | 374-383 | 377-383 |
| 15 | 389-398 | 393-400 |
| 16 | 417-426 | 420-427 |
| 17 | 438-447 | 442-448 |
| 18 | 473-482 | 475-483 |
Видим, что совпадение теперь куда заметнее. Таким образом, несовпадение координат, обнаруженное сначала, объясняется различиями структур (наличием и отсутствием сигнального пептида).
Задание 2. Сравнение предсказаний трансмембранных участков в альфа-спиральном белке
Цель задания: сравнить результаты предсказаний трансмембранных участков по последовательности (с помощью DeepTMHMM) и по трехмерной структуре (база данных OPM) в α-спиральном белке
В качестве структуры, содержащей трансмембранный α-спиральный белок, мне была выдана структура 6c3i. Это переносчик двухвалентных металлов MntH (Divalent metal cation transporter MntH) бактерии Deinococcus radiodurans. Идентификаторы в PDB и UniProt - 6c3i и MNTH_DEIRA соответственно. Согласно данным из Uniprot, этот белок осуществляет H+ -стимулируемое поглощение ионов двухвалентных металлов. Поскольку в белке две цепи (А и В), мною была выбрана цепь А (примечательно, что при запросе по названию белка в OPM в разделе Subunits выдается информация только о цепи А).
OPM определяет локализацию этого белка как находящийся во внутренней мембране грамотрицательной бактерии.
В OPM приведены следующие координаты трансмембранных участков выбранной цепи: 1( 47- 67), 2( 73- 92), 3( 114- 145), 4( 151- 166), 5( 175- 196), 6( 217- 237), 7( 262- 282), 8( 313- 336), 9( 349- 366),10( 370- 394),11( 410- 431)
Запуск DeepTMHMM для последовательности цепи А показал графический результат, представленный ниже:
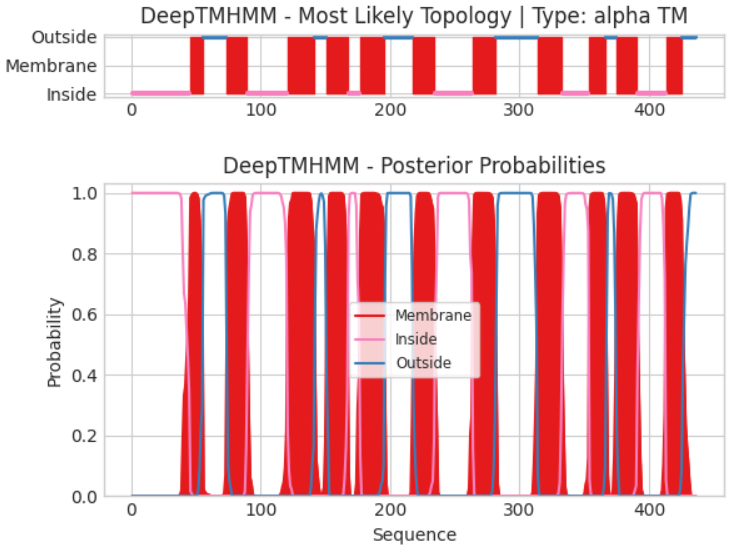
Из данного изображения видно, что наш белок имеет пронизывающие мембрану участки (показаны красными линиями), участки, обращенные наружу (синие) и внутрь клетки (розовые). Файл с результатами предсказаний доступен по ссылке, трансмембранные альфа-спиральные участки указаны в выдаче как TMhelix. Координаты этих участков: 46-55, 74-89, 121-141, 151-167, 177-195, 218-234, 264-281, 314-332, 354-366, 375-390, 413-425.
| Номер участка | OPM | DeepTMHMM |
|---|---|---|
| 1 | 47- 67 | 46 55 |
| 2 | 73- 92 | 74 89 |
| 3 | 114- 145 | 121 141 |
| 4 | 151- 166 | 151 167 |
| 5 | 175- 196 | 177 195 |
| 6 | 217- 237 | 218 234 |
| 7 | 262- 282 | 264 281 |
| 8 | 313- 336 | 314 332 |
| 9 | 349- 366 | 354 366 |
| 10 | 370- 394 | 375 390 |
| 11 | 410- 431 | 413 425 |
И OPM, и DeepTMHMM нашли 11 трансмембранных участков. Координаты этих участков перекрываются лучше, чем для бета-спирального белка, но для 9, 10 и 11 участка DeepTMHMM как бы "недопредсказал" участки - координаты участков DeepTMHMM "уже", чем указанные в OPM.