Практикум 8. Сигналы в геноме.
В качестве сигнала для поиска мною был выбран OriC участок репликации у бактерий. Носитель сигнала - бактериальные кольцевые хромосомы и плазмиды. Сигнал адресован белкам, отвечающим за инициацию репликации у бактерий. Согласно данным этой публикации, у большинства бактерий главным белком - инициатором репликации является белок DnaA. Этот белок работает в виде олигомера, его олигомеризацию модулируют другие белки. DnaA обеспечивает расплетание ДНК в АТ-богатом участке ориджина, а затем привлекает белок DnaB, являющийся хеликазой.
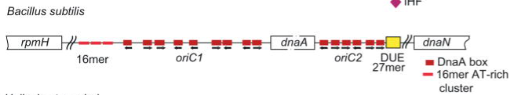
Для примера ниже показана схема ориджина репликации OriC бактериии Bacillus subtilis. В ориджине есть несколько функциональных элементов - DnaA box (сайт посадки белка DnaA), 16mer AT-rich cluster (АТ-богатый кластер) и DUE (DNA unwinding element; АТ-богатый участок, в котором DnaA плавит ДНК). В ориджине Bacillus subtilis между двумя участками DnaA box находится ген белка DnaA.
Моя гипотеза о силе сигнала: мне кажется, сигнал OriC можно считать сильным, поскольку репликация - важнейший процесс для бактерий, без которого не осуществимы клеточные деления, и привлечение белков инициации репликации на бактериальные хромосомы должно осуществляться быстро и точно.
Для поиска OriC можно использовать программу Ori-Finder 2022. Она разработана учеными из Tianjin University в 2022 году и доступна по ссылке. Опубликованное разработчиками описание программы можно найти по ссылке. Ниже представлена схема работы программы
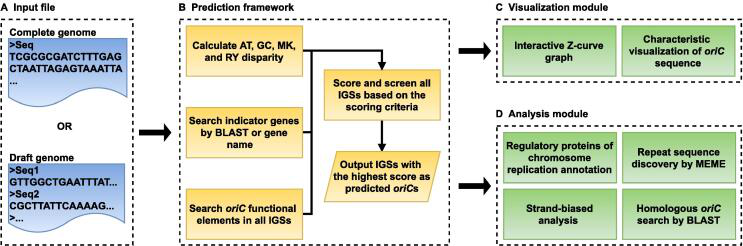
На вход программе дается файл с геномом или участком генома бактерии. Программа рассчитыет GC, AT, MK, RY составы для участков заданного генома, ищет индикаторные гены (те, рядом с которыми с большей вероятностью находится OriC, обычно это гены белков репликации) и функциональные элементы ориджинов. На основании этих параметров программа рассчитывает вес для каждого из участков генома, и выдает участки с наибольшим весом как наиболее вероятные ориджины. Полученные данные визуализируются на выходе с помощью интерактивной z-кривой.
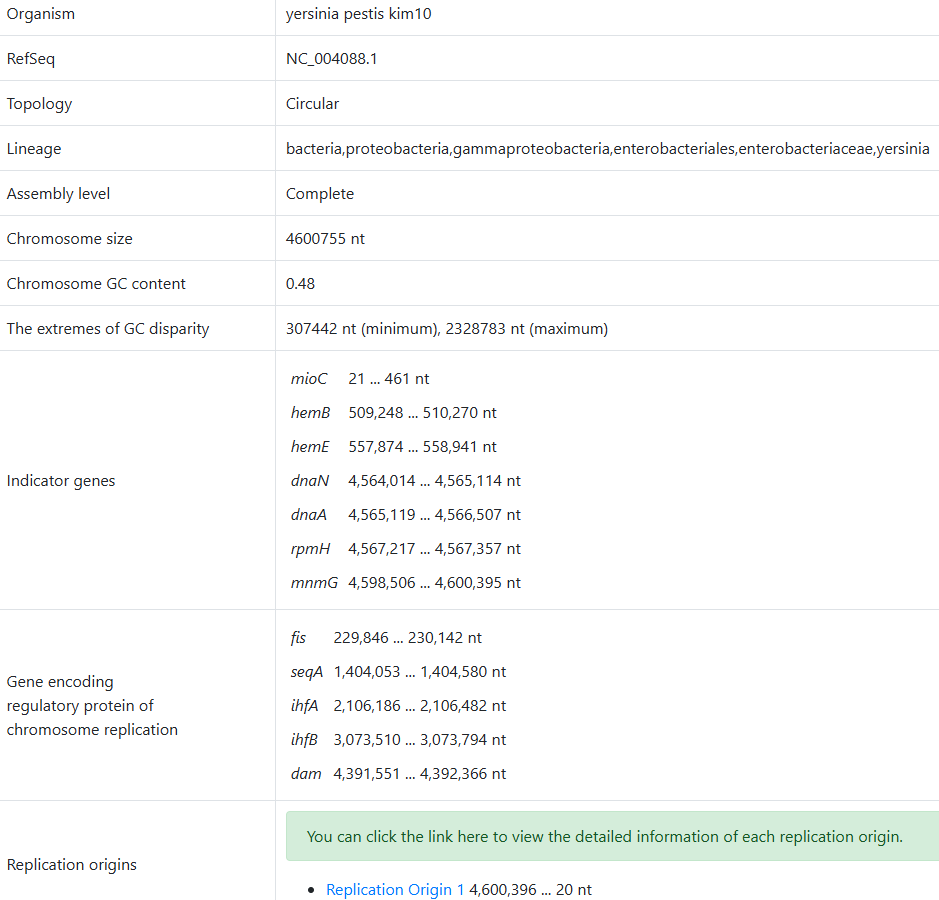
Протестируем работу этой программы, найдя ориджины в полном геноме бактерии Yersinia pestis KIM+ (данный организм использовался в примере на сайте; до него я пыталась поработать с Bacillus subtilis, но программа несколько раз не срабатывала и долго висела, видимо из-за некорректно введенного систематического положения). Систематическое положение этой бактерии: Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Yersinia. На вход подавалась полная последовательность хромосомы бактерии. Был выбран strand-biased analysis, в качестве референса для поиска мотива использовался мотив E.Coli, поскольку эта бактерия является близким родственников нашей.
В результате работы программа выдает информацию о данной на вход ДНК, координатах ориджина репликации (4,600,396 ... 20 nt), индикаторных и регуляторных генах и их координатах. По ссылке можно посмотреть расширенную информацию - последовательность ориджина, карту различных мотивов в нем и схему распределения генов на лидирующей и отстающей цепи.
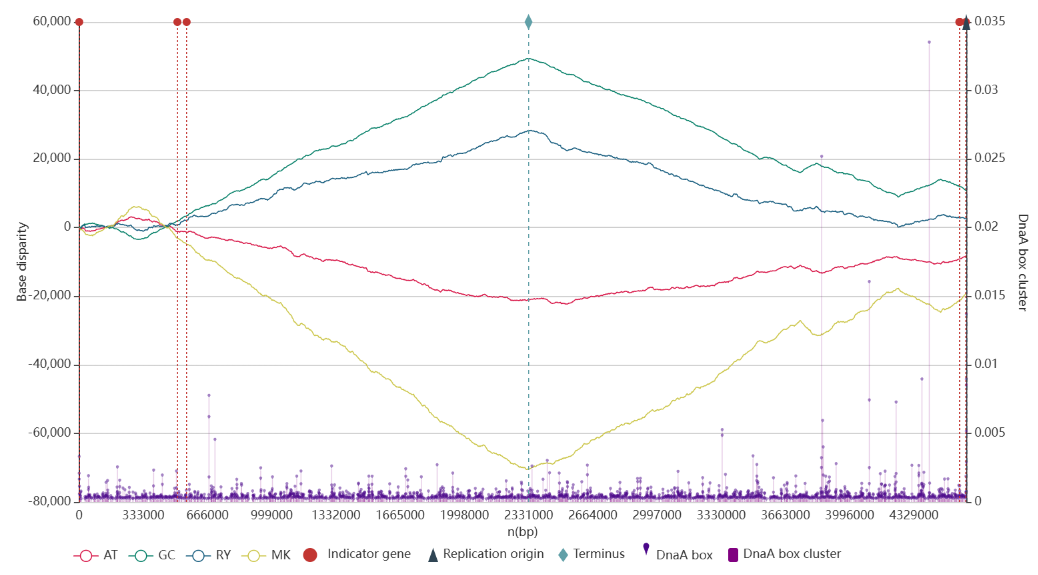
Также программа выдает z-кривую, на которой в наглядном виде представлено распределение пар нуклеотидов в предсказанных ей участках. Так, на полученной кривой мы видим один ориджин репликации и 4 индикаторных гена.
Итак, в ходе данного практикума я подробнее познакомилась с сигналом OriC и программой Ori-Finder 2022, которую можно использовать для нахождения этого сигнала.