

Предварительно были выравнены все иследуемые протеомы. Результат реконструкции в Newick формате:
((((((((CLPX_MYCTU,CLPX_MYCLE),CLPX_RHOJR),CLPX_ARTS2),(CLPX_LEIXX,CLPX_CLAMS)),(RUVB_ARTS2,A0JR82_ARTS2)),((Q6ACQ0_LEXX,B0RHW4_CLAMS),(Q0S8E3_RHOJR(FTSH_MYCTU,FTSH_MYCLE)))),(CLPX_BIFLO,A0K1M3_ARTS2)), ((RUVB_BIFLO, Q8G3S2_BIFLO), (A0K236_ARTS2,(QOS6Y7_RHOJR,(Q8G671_BIFLO,Q0S8C7_RHOJR)))));

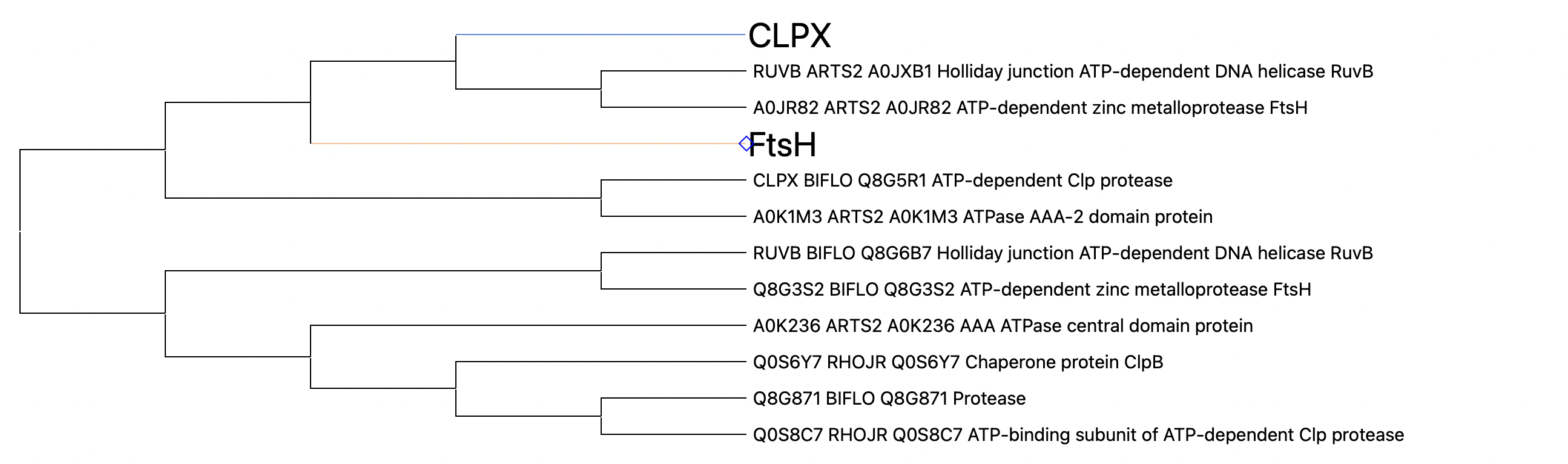
В полученном дереве я выделила две большие группы ортологов.
FtsH - цинковые металлопротеазы. В эту группу входят только LEIXX, CLAMS,RHOJR, MYCTU, MYCLE. С деревом видов сходится. На дереве с такой функцией есть еще и ATRS2, но это другой белок, реконструирующийся далеко от нашей группы и не соотносящейся с деревом видов.
Остальные белки не образуют характерных групп.