Трансмембранные белки
1. Знакомство с базой данных OPM
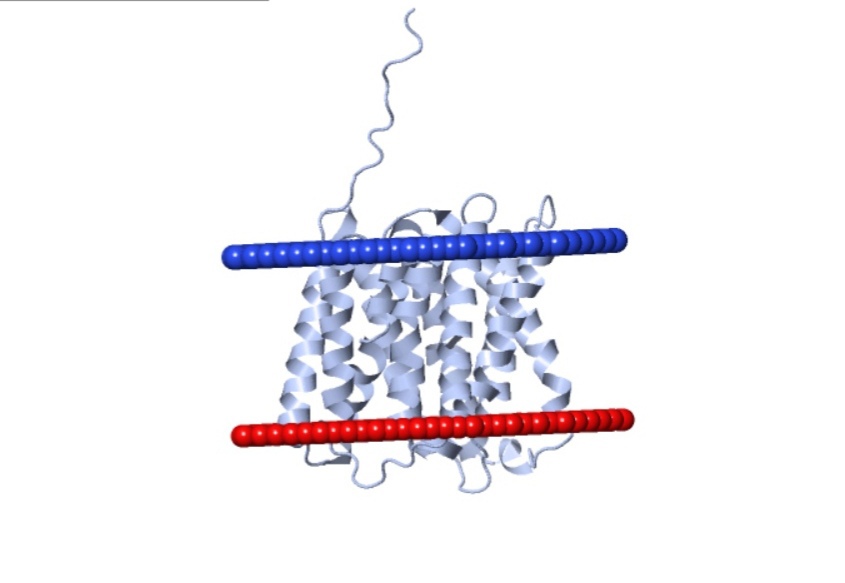
Я выбрала Attachment invasion locus protein (белок инвазивного прикрепления) в базе данных ОРМ. Этот белок находится в периплазме и пронизывает внешнюю мембрану бактерии Yersinia pestis, возбудителя бубонной чумы. Этот белок важен для прикрепления бактерии к клетке-хозяину и для уклонения от имунных реакций хозяина, поэтому это неплохой кандидат для разработки вакцины от чумы [1].
Общая информация об исследуемом белке приведена в таблице ниже:
Координаты трансмембранных участков: 1( 29- 37), 2( 54- 62), 3( 69- 75), 4( 98- 106), 5( 113- 119), 6( 141- 150), 7( 157- 162), 8( 173- 180).
2. DeepTMHMM
В начале у меня было двe входные последовательности:
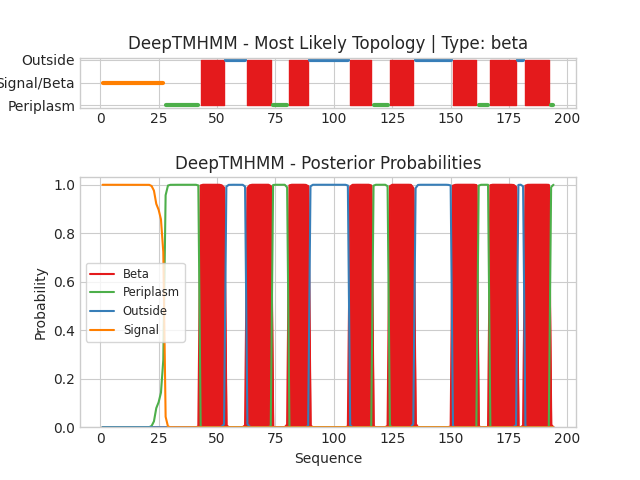
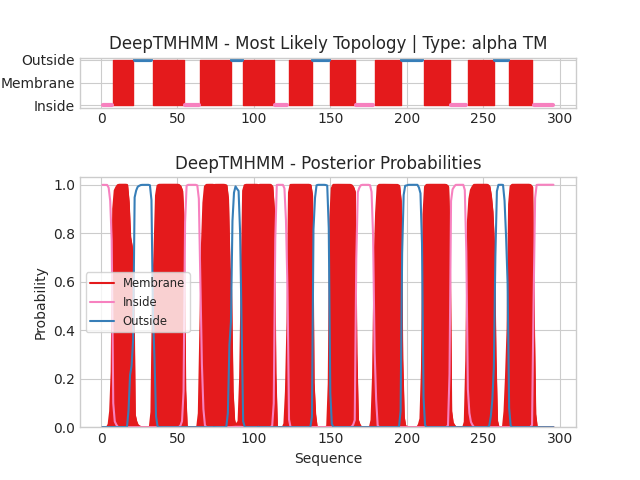
выбранный мною в предыдущем пункте β-листовой белок 3qra и выданный мне α-спиральный белок YWFM_BACSU.Используя сервис DeepTMHMM я получила графические и текстовые данные:
textНа нижней схеме представленны вероятности пренадлежности аминокислотного остатка к одной из групп: сигнал локализации на внешней мембране (оранжевый), β-листовой трансмембранный участок (красный), локализация во внешней среде (синий), локализация в периплазме (зеленый), а на верхней схеме наиболее вероятная топология этого белка относительно мембраны.
textНа нижней схеме представленны вероятности пренадлежности аминокислотного остатка к одной из групп: трансмембранная α-спираль (красный), локализация во внешней среде (синий), локализация в цитоплазме (розовый), a на верхней изображена наиболее вероятная топология белка.
3. PPM
Я использовала PPM 3.0, так как я тоже за будущее!
Были выбраны следующие параметры:
Type of membrane Gram-negative bacteria outer membrane
Allow curvature no
Topology (N-ter) in (по результату DeepTMHMM)
Transmembrane secondary structure segments: 1(5 - 24), 2(33 - 55), 3(65 - 86), 4(91 - 110), 5(120 - 138), 6(147 - 167), 7(176 - 196), 8(209 - 231), 9(236 - 257),10(264 - 282).
4. Сравнение алгоритмов предсказания трансмембранных спиралей
Сравним координаты трансмембранных участков альфа-спирали у данного мне белка (по данным ОРМ) и по предсказанию DeepTMHMM:
ОРМ: 1(5 - 24), 2(33 - 55), 3(65 - 86), 4(91 - 110), 5(120 - 138), 6(147 - 167), 7(176 - 196), 8(209 - 231), 9(236 - 257),10(264 - 282)
DeepTMHMM: (8 - 21), (34 - 54), (65 - 85), (93 - 113), (123 - 138), (150 - 166), (179 - 196), (211 - 228), (240 - 257), (267 - 282)
Количество предсказанных транс участков совпадает с данными в OPM (10 штук) и в целом координаты довольно похожи. Это говорит о том, что DeepTMHMM довольно хорошо предсказывает структуру.
Далее сравним бета-слои у выбранного мной белка (та же борьба между OPM и DeepTMHMM):
ОРМ: 1( 29- 37), 2( 54- 62), 3( 69- 75), 4( 98- 106), 5( 113- 119), 6( 141- 150), 7( 157- 162), 8( 173- 180)
DeepTMHMM: (43 - 53), (63 - 73), (81 - 89), (107 - 116), (124 - 134), (151 - 161), (167 - 178), (182 - 192)
Сначала в глаза бросается, что количество трансмембранных участков снова совпадает (8 штук). Затем кажется, что координаты как-то непохожи.
С третьего взляда начинает казаться, что они как-то странно и подозрительно похожи и непохожи одновременно.
Загвоздка кроется в наличии сигнальной структуры в бета-листовом белке, которую учитывает DeepTMHMM, но не учитывает OPM так как сигнальный пептид имеет свойство отщеплятся. Так что смещение координат обосновано, DeepTMHMM все еще внушает доверие.5. База данных TCDB
AC P39649
Нашелся результат!
2.A.7.3.4
2. Потенциалзависимые электрохимические транспортные каналы
А. Транспортные системы для катализа унипортера, симпортера и антипортера
7. Надсемейство переносчиков лекарств/метаболитов (DMT)
3. Семейство 10 экспортеров наркотиков/метаболитов (DME)