Практикум 12
Сравнение выравнивания одних и тех же последовательностей разными программами.
Для этого задания я выбрала белки с ID: P53_RAT, P53_DANRE, P53_HUMAN, P53_BOSIN. Данный белок называется p53 и является транскрипционным фактором, который препятствует образованию злокачественных опухолей.
1. Выравнивания, построенные в Jalview с помощью программ Muscle и ClustalO (Colour: Percentage Identity):
В результате визуального сравнения двух выравниваний можно увидеть следующие совпадающие участки: 2-30, 80-183, 184-292, 313-351, 371-391.
2. Сравнение выравниваний в VerAlign.
Далее я решила применить сервис VerAlign, чтобы сравнить выдачу результатов программ Muscle и ClustalO.
Выравнивание по совмещению структур.
Я выбрала белки из семейства протеинкиназ (Pkinase, PF-номер:PF00069). Это семейство состоит из ферментов, которые с помощью каталитической субъединицы, переносят остатки фосфорной кислоты от АТФ (чаще всего) к одному или нескольким аминокислотным остаткам белкового субстрата, что, как правило, изменяет функцию субстрата [1].
Запрос на поиск белков с известной 3D-структурой в UniProtKB:
"(xref:pfam-PF00069) AND (reviewed:true) AND (existence:1)"
ID взятых мной белков: WEE2_HUMAN (PDB_ID: 5VDK) - получен из организма человека (Homo sapiens), PRAG1_RAT (PDB_ID: 6EWX) - из организма крысы (Rattus norvegicus), BAK1_ARATH (PDB_ID: 3TL8)- из резуховидки Таля (Arabidopsis thaliana).
Выравнивание структур осуществлялось в Pymol с помощью команды align:
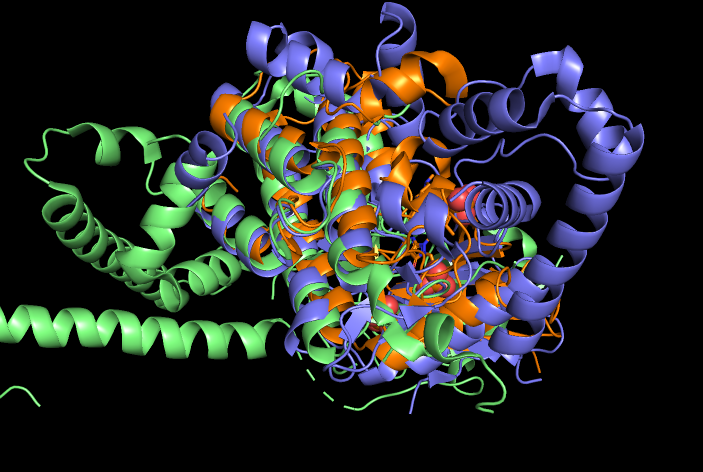
В структурном выравнивании можно увидеть как совпадающие (в осносвном это α-спирали), так и несовпадающие участки, при этом несопадающих больше.
Выравнивание тех же белков в Jalview показало очень маленькое количество совпадений в последовательностях:
Процент совпадения равен 30%, значит, последовательности гомологичны.
Несовпадающие участки: 1-9, 14-18, 33-36, 54-62, 89-95,102-106, 109-114, 118-120, 140-145, 159-166, 173-192, 200-206, 217-220, 228-263, 295-300.
Совпадающие участки: 10 (у человека и крысы), 13 (человек и резуховидка), 19 (человек и крыса), 22 (человек и резуховидка), 29-32, 39-40 (человек и резуховидка), 43 (человек и крыса), 44-46, 49 (крыса и резуховидка), 63 (крыса и резуховидка), 68 (человек и крыса), 70 (человек и крыса), 78-79, 100-101 (человек и резуховидка), 127-130, 134-135, 137-139, 146-150, 167-169, 193-199, 207, 209, 211, 214, 264 (человек и резуховидка), 280-282, 289 (человек и крыса), 290 (крыса и резуховидка), 293, 301.
Описание программы T-Coffee.
T-Coffee (Tree-based Consistency Objective Function for Alignment Evaluation - Целевая функция согласованности на основе дерева для оценки выравнивания ) - одна из программ для множественного выравнивания аминокислотных и нуклеотидных последовательностей. T-Coffee имеет две основные особенности:
1. Обеспечение простого средства создания множественных выравниваний с использованием различных библиотек, которые сгенерированы с использованием как локальных, так и глобальных выравниваний [2].
2. Вторая особенность носит название "Метод оптимизации" (Optimization method). Метод заключается в том, что построение множественного выравнивания складывается из результатов попарного выравнивания каждой последовательности с какой-либо, взятой из библиотеки [2].
Также 3D-Coffee использует информацию о структурах белков из PDB для построения выравниваний.
Список литературы
- Ссылка: Описание семейства протеинкиназ
- Notredame C, Higgins DG, Heringa J. T-Coffee: A novel method for fast and accurate multiple sequence alignment. 2-3. Doi:10.1006/jmbi.2000.4042. PMID 10964570