Сборка генома de-novo (NGS)
1. Триммирование чтений
В этом пратикуме были использованы чтения с кодом доступа SRR4240359.
Скачивание архива с помощью команды wget:
Для того, чтобы дальше удалить адаптеры, подготовим файл, где будут лежать все адаптеры (которые находятся в папке /mnt/scratch/NGS/adapters). Для этого воспользуемся следующей командой:
Удалим адаптеры из чтений:
Теперь с правых концов чтений избавимся от нуклеотидов с качеством ниже 20 (параметр - TRAILING:20) и оставим чтения, длина которых не меньше 32 нуклеотидов (параметр - MINLEN:32):
2. Программа velveth (подготовка k-меров)
Для сборки de novo подготовим k-меры длины k=31, т.к. чтения после триммирования не короче 30 нуклеотидов. Используем программу velveth. Все результаты находятся в /mnt/scratch/NGS/daria.yastreb/pr15/velvet.
Параметр -short — короткие непарные чтения.
3. Программа velvetg (сборка на основе k-меров)
Запустим сборку из получившихся k-меров с помощью команды:
В папке velvet лежат следующие файлы:
contigs.fa Graph LastGraph Log PreGraph Roadmaps Sequences stats.txt
Файл Log содержит информацию о N50 = 70 607. В файле stats.txt можно посмотреть самые длинные контиги и их покрытие. Таблица stats.txt была отсортирована с помощью команды: sort -nk 2 stats.txt. Следующие контиги оказались самыми длинными: ID 11 (длина - 125 674, покрытие - 44,55) ID 1 (длина - 108 447, покрытие - 42,00), ID 14 (длина - 71 403, покрытие - 39,41).
Один из контигов с аномально большим покрытием: ID 111 (длина - 1, покрытие - 411220).
4. Анализ с помощью megablast
Сравним программой megablast каждый из трёх самых длинных контигов с хромосомой Buchnera aphidicola (GenBank/EMBL AC — CP009253).
Задание было выполнено на сайте.
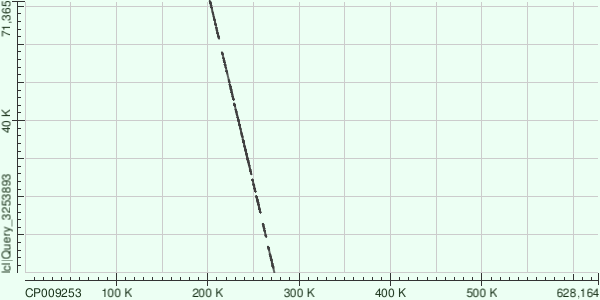
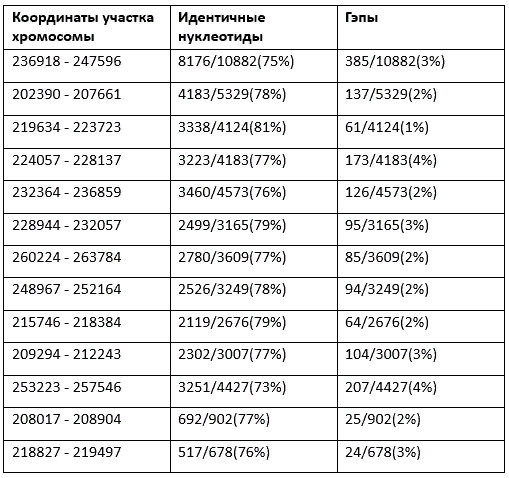
Ниже представлены карты локального сходства, характеристики каждого контига отображены в таблицах.
ID 1
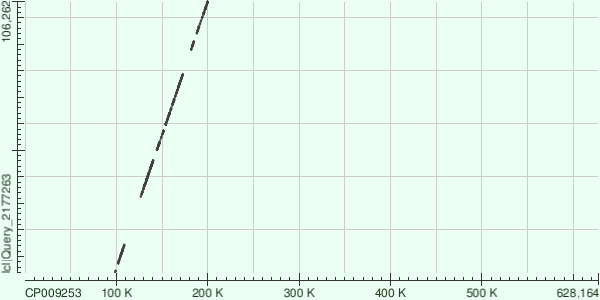
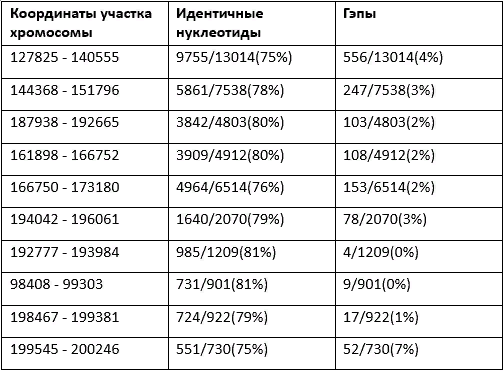
ID 11
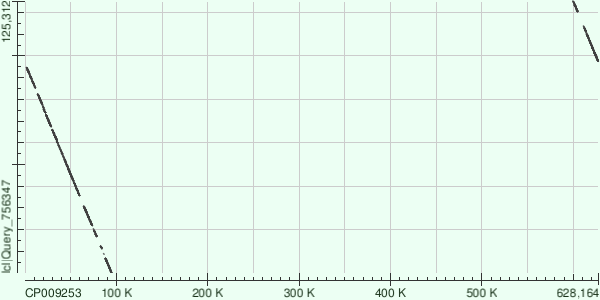
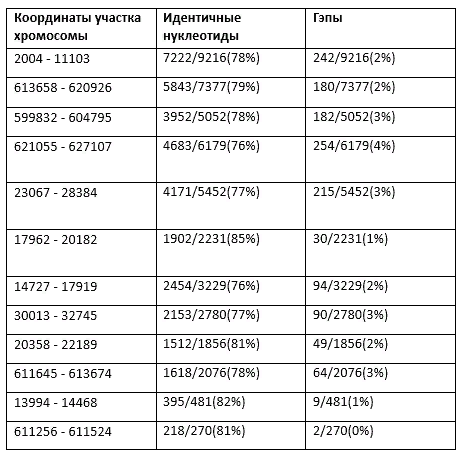
ID 14