Мотивы в белках
I. Поиск консервативных мотивов в выравнивании
Из Pfam был выбран домен Na/K-АТФазы (PF00287), а именно бета-субъединицы Na/K-АТФазы.
Далее с сайта было скачано выравнивание seed, содержащее 55 последовательностей. При redundancy treshold 100% и 90% ничего не выделяется, поэтому продолжаем работать с 55 последовательностями.
В выравнивании обнаружено не очень большое количество мотивов. Я визуально выделила консервативный участок:
Запись паттерна в Jalview-формате: W..I..[FY]Y..[FY]Y..[LM]
Паттерн в PROSITE-формате: W-x-x-I-x-x-[FY]-Y-x-x-[FY]-Y-x-x-[LM]
Данный мотив был найден во всех 55 последовательностях. Теперь выполним поиск по этому паттерну в базе данных SwissProt в PROSITE. Было обнаружено всего две находки, принадлежащие бета-субъединицам Na/K-АТФаз. Можно предположить, что выбранный мотив не является характерным для данного семейства.
II. Поиск мотива, специфичного для одной клады филогенетического дерева
Для этого задания построим филогенетическое дерево методом NJ по выравниванию, которое у нас есть. Была выбрана клада с мотивом, представленном на Рис. 1:
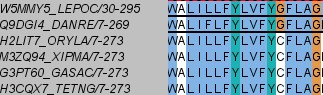
Исходя из выравнивания этих белков, можно сказать, что мотив данной клады совпадает с мотивом последовательностей из предыдущего задания, т. е. мотив этой клады не специфичен для неё.
III. PSI-BLAST
Выберем белок с AC: P74518. Этот белок представляет собой фактор гибернации рибосом (осуществляет переход рибосом в неактивное состояние путём димеризации активных 70S рибосом в 100S рибосомы, которые трансляционно неактивны). Фактор выделен из бактерии Synechocystis sp, штамм PCC 6803. Воспользуемся белковым BLAST в NCBI (внесём AC в специальное окно, выберем PSI-BLAST и поиск по Swiss-Prot). После каждого запуска BLAST заполнялась новая строка Таблицы 1 (на Рис. 2).
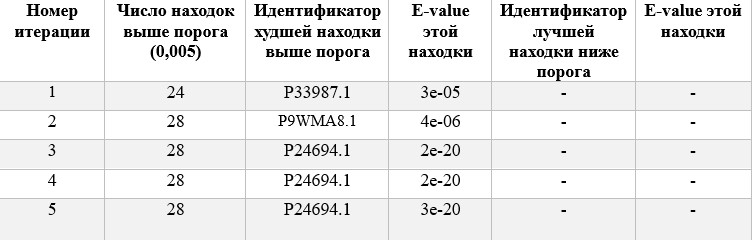
Стабилизация результатов наступает на третьей итерации. Отметим, что для данной группы белков не было белков, со значением E-value выше порогового (0.005). Можем сказать, что семейство белков обособленно.