Домены и профили
I. Отбор последовательностей
Изначально во время работы в классе был выбран домен Laminin B (Domain IV), PF00052. Ламинины - гликопротеины, являющиеся основными компонентами базальных мембран. Ламинины участвуют в процессах клеточной дифференцировке, миграции и адгезии клеток. Однако при выполнении этого практикума пришлось отказаться от данного домена, потому что не было найдено подсемейств с двудоменной архитектурой, у которых число представителей подсемейства находилось в пределах 40 - 200 (число представителей было меньше 40).
По вышеуказанным причинам был выбран другой домен - GAGA-factor, PF09237. Белки, являющиеся GAGA-факторами, связываются с консенсусным сайтом связывания ДНК 5'-GAGAG-3', и также содержат ядро цинковых пальцев типа Cys2-His2 (эти аминокислоты координируют ион цинка в цинковых пальцах).
Для выбранного домена есть 29 архитектур, возьмём из них архитектуру, содержащую два домена: PF00651 - PF09237 (включает 194 последовательности). 194 последовательности были скачаны и выравнены в Jalview алгоритмом Mafft with Defaults: Выравнивание последовательностей белков подсемейства (позитивная выборка).
Далее с помощью Remove redundancy (с порогом 90%) были удалены очень похожие последовательности, после этого действия осталось 138 последовательностей: Выравнивание (тестовая выборка).
Для отрицательного (негативного) контроля были взяты последовательности с двудоменной архитектурой: PF09237 - PF13909. Добавим эти последовательности ко всем последовательностям выбранного подсемейства: Итоговая выборка.
II. Создание HMM-профиля подсемейства PF00651 - PF09237
Для создания HMM-профиля были использованы следующие команды из пакета HMMER:
hmm2build -g hmm_out.txt test.fa
hmm2calibrate hmm_out.txt
hmm2search --cpu 1 hmm_out.txt full.fa > hmm2search_out.txt
hmm2build выдаёт профиль по выравниванию - профиль HMM двудоменной архитектуры, Далее после калибровки статистик поиска, проводили сам поиск по итоговой выборке - находки (анализ по итоговой выборке).
III. Анализ HMM-профиля
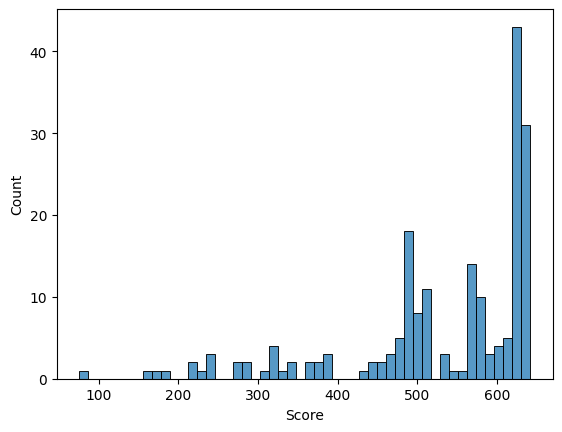
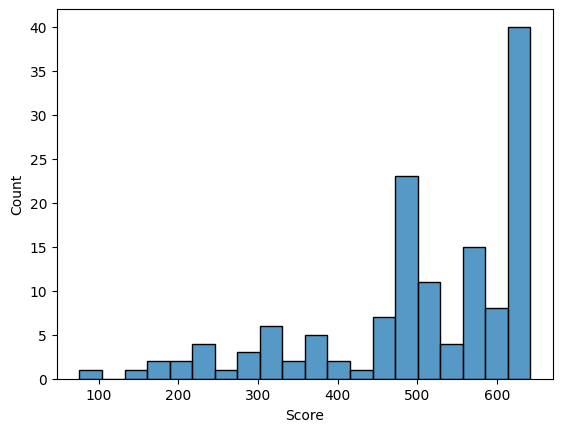
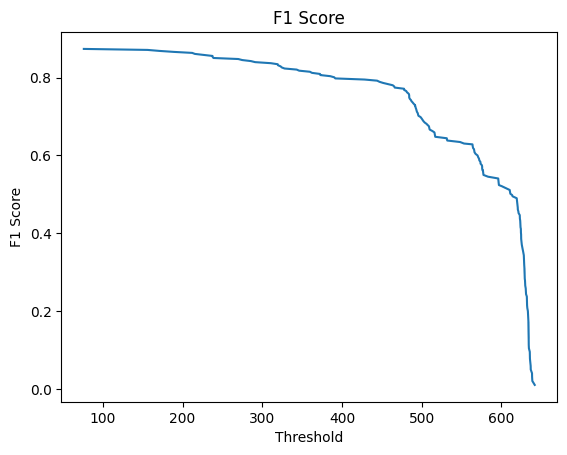
С помощью скрипта, предоставленного Каримовой Кариной, были представлены следующие результаты. Из гистограмм видно, что у нас отсутствуют отрицательные веса, что довольно странно. Порог лучше брать от 480.