Практикум 4
Составление списка гомологичных белков, включающих паралоги
Таблица из предыдущего практикума с отобранными бактериями:
| Название | Мнемоника |
|---|---|
| Pasteurella multocida | PASMU |
| Shewanella denitrificans | SHEDO |
| Burkholderia mallei | BURMA |
| Polynucleobacter asymbioticus | POLAQ |
| Thiobacillus denitrificans | THIDA |
| Neisseria meningitidis | NEIMA |
| Bartonella henselae | BARHE |
Для поиска достоверных гомологов белка CLPX_ECOLI в бактериях, которые были отобраны мной в предыдущих практикумах, была использована программа blastp.
Сначала были скачаны протеомы из директории /P/y22/term4/Proteomes и произведено копирование последовательностей в один файл proteomes.fasta с использованием следующией команды:
cat PASMU.fasta SHEDO.fasta BURMA.fasta POLAQ.fasta THIDA.fasta NEIMA.fasta BARHE.fasta > ~/term4/pr4/proteomes.fastaТеперь создадим базу данных из последовательностей белков:
makeblastdb -dbtype prot -in proteomes.fasta -out myprot_dbНайдём гомологи белка CLPX_ECOLI в созданной базе данных с E-value = 0.001:
blastp -db myprot_db -query CLPX_ECOLI.fasta -out homologs -evalue 0.001В результате был получен следующий файл: blastp_results.
Реконструкция и визуализация
Последовательности находок находятся в этом файле.
Далее построим филогенетическое дерево по последовательностям полученных находок гомологов с помощью программы FastME (сайт: NGPhylogeny.fr). Для выравнивания последовательностей был использован MAFFT.
Параметры для FastME:
Gamma distributed rates across sites - No
Starting tree - BIONJ
No refinement
100 bootstrap реплик
Остальные параметры - по умолчанию
Newick-формула дерева без укоренения в среднюю точку. Ниже на Рис. 1 представлено дерево после укоренения в среднюю точку.

По построенному дереву найдем пары ортологов и паралогов:
>> Ортологи - гомологичные белки из разных организмов, разделение их общего предка (на линии, ведущие к белкам) произошло в результате видообразования.
>> Паралоги - гомологичные белки, произошедшие от одного общего предка, но выполняющие разные функции в клетке.
Построим дерево, покрасив клады в соответствии с ортологичными группами:

Далее построим дерево, у которого все ортологические группы, содержащие более трёх последовательностей, - "схлопнуты".

1) FTSH - группа АТФ-зависимых цинк металлопротеаз, контролирует качество интегральных мембранных белков.
2) HSLU - группа АТФазных субъединиц HslU АТФ-зависимых протеаз. Связывание АТФ и его последующий гидролиз необходимы для подготовки субстрата к HslV гидролизу.
3) CLPX - группа АТФ-связыващих субъединиц ClpX АТФ-зависимых протеаз Clp.
На рис. 4 отобразим белки каких бактерий отстуствуют в данных ортологических группах:
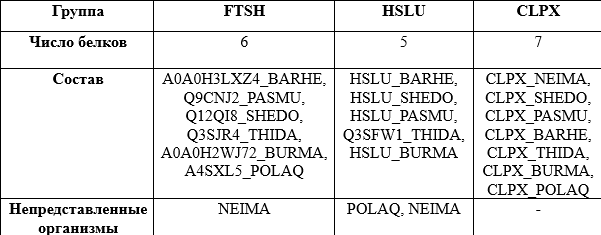
1) > FTSH - для представленных в ней белков филогения по ним полностью соответствует филогении бактерий.
2) > HSLU - для представленных белков наблюдается несоответствие внутри клады {HSLU_THIDA, HSLU_BURMA} с соответствующей для бактерий, в остальном деревья совпадают.
3) > CLPX - филогения белков не соотвествует филогении бактерий. Но были правильно выделены следующие нетривиальные ветви:
1. {BARHE, NEIMA, THIDA, POLAQ, BURMA} против {SHEDO, PASMU}
2. {BARHE, SHEDO, PASMU, NEIMA} против {THIDA, POLAQ, BURMA}