Трансмембранные белки
I. Сравнение предсказаний трансмембранных участков в бета-листовом белке
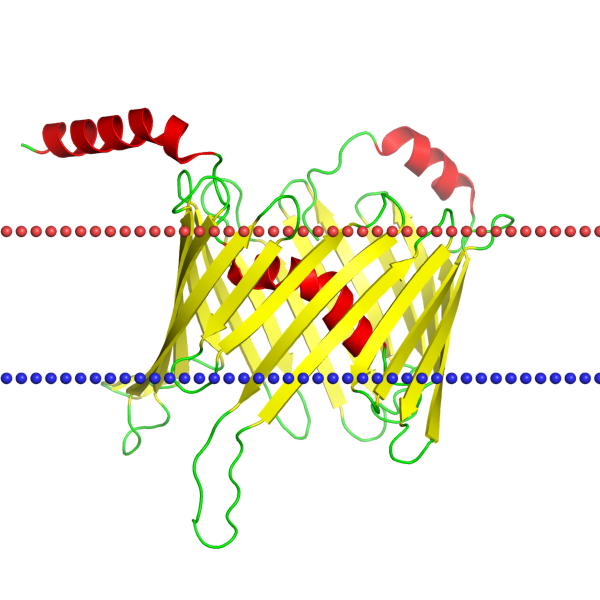
С помощью поиска по уровням классификации в базе данных OPM мной был найден белок со следующими характеристиками:
Изображение структуры белка:
Координаты трансмембранных участков белка, приведенные в OPM:
Запуск DeepTMHMM для последовательности белка:
Из UniProt скачаем последовательность белка. Загрузим последовательность в DeepTMHMM.
Выдача DeepTMHMM:
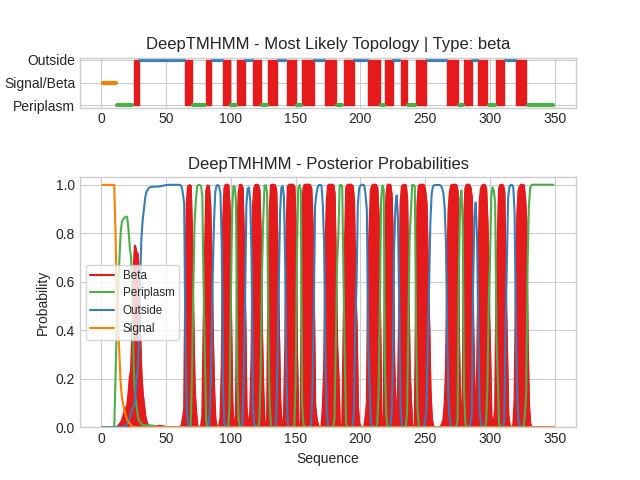
Однако, если воспользоваться этой версией DeepTMHMM, то результаты предсказаний будут иными:
Сравнение трансмембранных участков: OPM; DeepTMHMM (по второй версии DeepTMHMM)
Координаты трансмембранных участков незначительно различаются (лишь в одном случае полностью совпадают - см. 8). К тому же с помощью DeepTMHMM было обнаружено 20 β-тяжей, OPM заметил 19.
II. Сравнение предсказаний трансмембранных участков в альфа-спиральном белке
Для сравнения результатов предсказаний трансмембранных участков по последовательности и по AlphaFold-модели структуры в α-спиральном белке мне был выдан белок COXX_RHOPB со следующими характеристиками:
Запуск DeepTMHMM для последовательности белка:
Из UniProt скачаем последовательность белка и загрузим последовательность в DeepTMHMM.
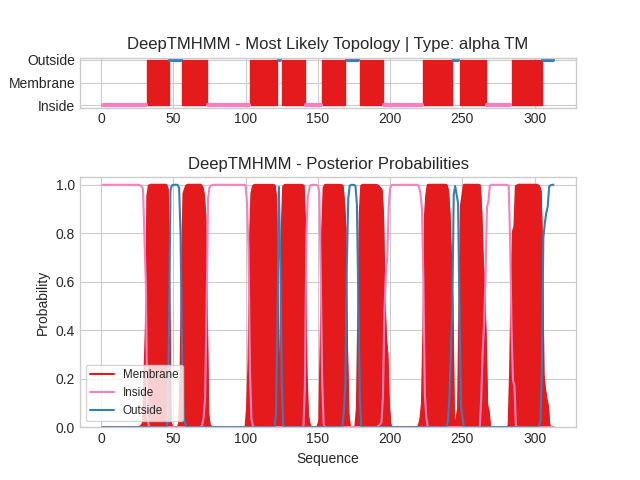
В этот раз предсказания обеих версий DeepTMHMM оказались одинаковыми.
Выдача DeepTMHMM:
AlphaFold:
Скачаем предсказание AlphaFold трехмерной структуры белка Protoheme IX farnesyltransferase (раздел "Structure" записи в UniProt): файл
Запуск PPM:
Полученный файл загрузим в PPM 3.0 со следующими параметрами:
Сравнение трансмембранных участков: PPM 3.0; DeepTMHMM
И PPM 3.0, и DeepTMHMM в этот раз нашли одинаковое количество трансмембранных участков. Как и в первом случае координаты трансмембранных участков отличаются незначительно (наибольшее отличие по координатам было найдено в 4 участке), также есть один случай полного совпадения координат (см. 8).