Поиск de novo сигналов в ДНК
I. Выбор бактерии и подготовка данных
Для поиска сигнала посадки σ-субъединицы РНК-полимеразы была выбрана бактерия Escherichia coli, штамм K-12 (NCBI RefSeq assembly: GCF_000005845.2).
Мной были скачаны два файла: последовательность хромосомы в формате FASTA и аннотация в формате GFF. Далее файлы были загружены в Operon Mapper для получения списка оперонов.
Затем для дальнейшего анализа был использован скрипт, позаимствованный у Георгия Муравьева. На вход подавались список оперонов и геном бактерии. Получены следующие данные:
II. Поиск с помощью MEME
Для поиска сигналов в последовательностях воспользуемся локальным MEME на kodomo с помощью следующей команды:
meme housekeeping.fasta -dna -nmotifs 3 -minw 6 -maxw 50Параметры команды:
Выдача MEME в текстовом формате.
В результате работы MEME найдены три мотива, характеристики которых представлены ниже:
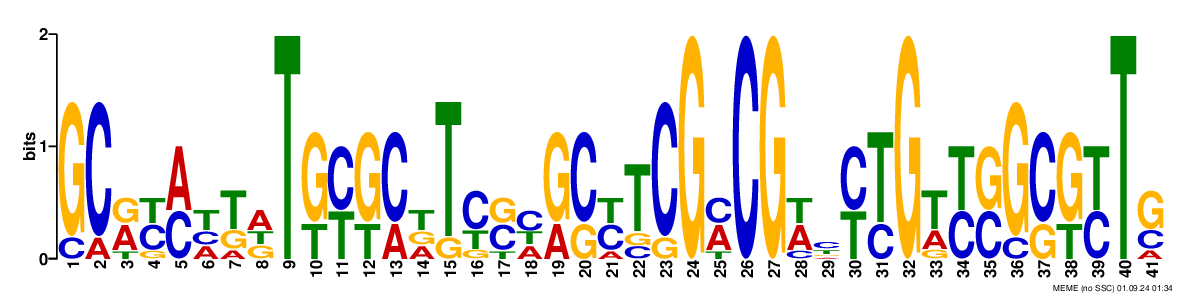


Для дальнейшей работы выберем первый мотив, потому что у него значение E-value наименьшее (8.9e-009).
Консенсусная последовательность первого мотива: GCRYMHKDTGYGCDTYSHGCYTCGMCGWYYTGWYSGCGYTS.
III. Поиск с помощью FIMO
Мотив GCRYMHKDTGYGCDTYSHGCYTCGMCGWYYTGWYSGCGYTS был проверен на материале тестовой выборки (положительный контроль) и на выборке отрицательного (негативного) контроля с помощью FIMO на kodomo:
fimo --norc -motif GCRYMHKDTGYGCDTYSHGCYTCGMCGWYYTGWYSGCGYTS -thresh 0.01 ~/term4/pr9/meme_out/meme.txt promotors.fasta
fimo --norc -motif GCRYMHKDTGYGCDTYSHGCYTCGMCGWYYTGWYSGCGYTS -thresh 0.01 ~/term4/pr9/meme_out/meme.txt negative.fasta
Выдача в формате HTML для положительного и отрицательного контролей.
С порогом на P-value (-thresh) равным 0.01 найдено 3368 последовательностей в тестовой группе, в группе отрицательного контроля - 363 последовательности. Это не совсем удачный результат, т.к. всего промоторных последовательностей в тестовой выборке насчитывается 4445, в отрицательном контроле - 312.