PDBSum
Выдача программы PROCHECK.Карта Рамачандрана:
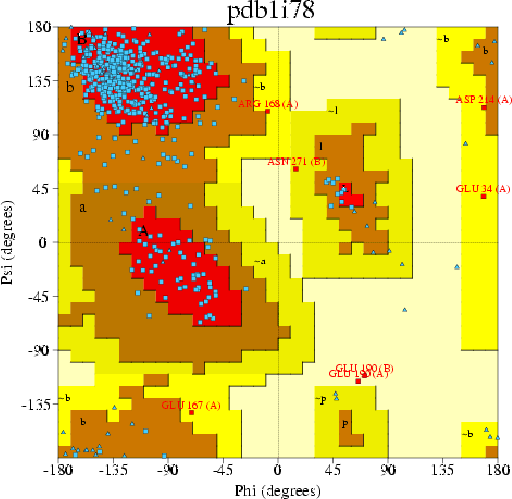
Доля остатков (отличных от Gly и Pro), попавших в предпочитаемые области на карте: 85.4%.
Модель считается качественной, если данная величина > 90%. Наше значение чуть меньше порога, поэтому нашу модель можно считать более или менее качественной.
EDS -> Significant regions
В цепи A 13, а в цепи B 14 остатков имеют большой Z-score по RSR.Выберем остаток Glu 167.
RSR = 0.325
коэффициент корреляции для электронной плотности = 0.709
Z-score по RSR = 2.115906
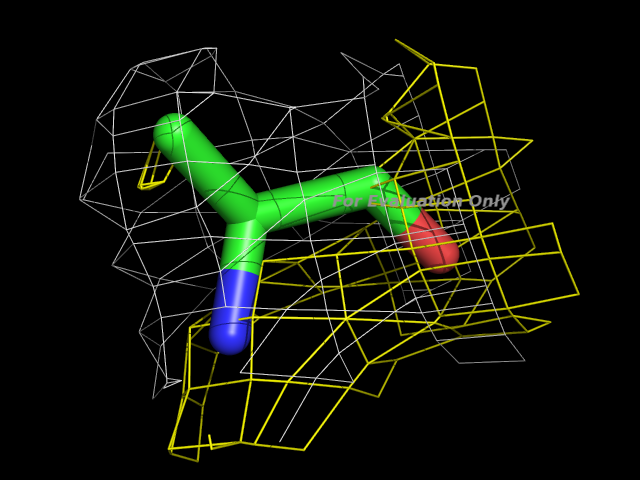 Изображение создано в PyMOL
Изображение создано в PyMOL
Жёлтым изображены линии электронной плотности уровня 1, белым - уровня 0.5.
Качество структуры по данному остатку очень низкое. Соответсвующая ему электронная плотность имеет низкие значения, поэтому было трудно его вписать, настолько, что авторы даже решили "урезать" карбоксильную группу. Неудивительно, что значения таких характеристик, как RSR, коэффициент корреляции для электронной плотности и Z-score по RSR, выходят за допустимые пределы.
EDS -> PDB_REDO
Данный раздел даёт информацию об улучшенной структуре.
| From PDB header | Calculated from data | After conservative optimisation | After full optimisation | |
|---|---|---|---|---|
| R | 0.2380 | 0.2377 | 0.1997 | 0.1975 |
| R-free | 0.2800 | 0.2453 | 0.2255 | 0.2380 |
| σR-free | 0.0047 | 0.0043 | 0.0045 | |
| R-free Z-score | 15.77 | 9.98 | 6.09 |
Первый столбец содержит значения из PDB-записи, второй - значения, рассчитанные по данным, последний - те же значения для оптимизированной структуры. Очевидные улучшения по всем показателям.
| Original PDB entry | Conservatively optimised | Fully optimised | |
|---|---|---|---|
| 1st generation packing quality1 | 0.535 | 0.625 | 0.823 |
| 2nd generation packing quality1 | -0.938 | -0.575 | -0.422 |
| Ramachandran plot appearance1 | -2.376 | -0.614 | -0.515 |
| Chi-1/Chi-2 rotamer normality1 | -2.373 | -1.860 | -1.571 |
| Backbone conformation1 | -0.111 | -0.214 | -0.102 |
| Bond length RMS Z-score2 | 0.328 | 0.553 | 0.536 |
| Bond angle RMS Z-score2 | 0.611 | 0.715 | 0.688 |
| Total number of bumps3 | 110 | 51 | 37 |
| Unsatisfied H-bond donors/acceptors3 | 26 | 19 | 24 |
1 Чем выше тем лучше
2 Должно быть меньше 1.000
3 Чем меньше, тем лучше
Аналогичное разделение по столбцам: слева значения по записи в PDB, справа - по оптимизированной структуре. Здесь видно, что улучшились показатели по карте Рамачандрана, по ротамерам, конформации остова цепи, количеству незадействованных возможных водородных связей, количеству слишком коротких расстояний между атомами, а также непонятные показатели 1st & 2sd generation packing quality.
(*) Скачанная оптимизированная структура(EDS -> PDB_REDO)
На взгляд оптимизированная структура отличается от оригинальной тем, что часть атомов немного немного изменили своё положение.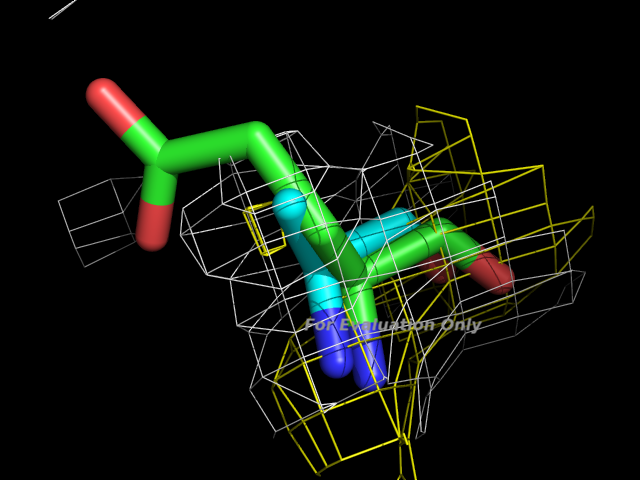 Изображение создано в PyMOL
Изображение создано в PyMOL
Оптимизированная структура с зелёными углеродами, оригинальная - с голубыми.
Для остатка из упражнения 2 была сделана картинка с изображением обеих структур и электронной плотностью. В такую электронную плотность невозможно как следует вписать глутамат, тем не менее оптимизированная структура вписывается чуть лучше, кроме того в ней глутамат не урезан так, как это сделано в оригинальной.
(*) Выдача WHAT_CHECK (для структуры из PDB)
Выдача данной программы очень объёмная и всесторонняя, но наиболее полезной информацией являются всевозможные ошибки (Error).- Error: Nomenclature error(s)
Список ошибок в номенклатуре некоторых глутаматов и аспартатов. - Error: Abnormally short interatomic distances
Список слишком коротких межатомных расстояний. - Error: Water molecules without hydrogen bonds
Молекулы воды, не образующие водородные связи. - Error: HIS, ASN, GLN side chain flips
Неправильно повёрнутые указанные остатки. При повороте на 180 градусов они бы образовывали энергетически более выгодные водородные связи.