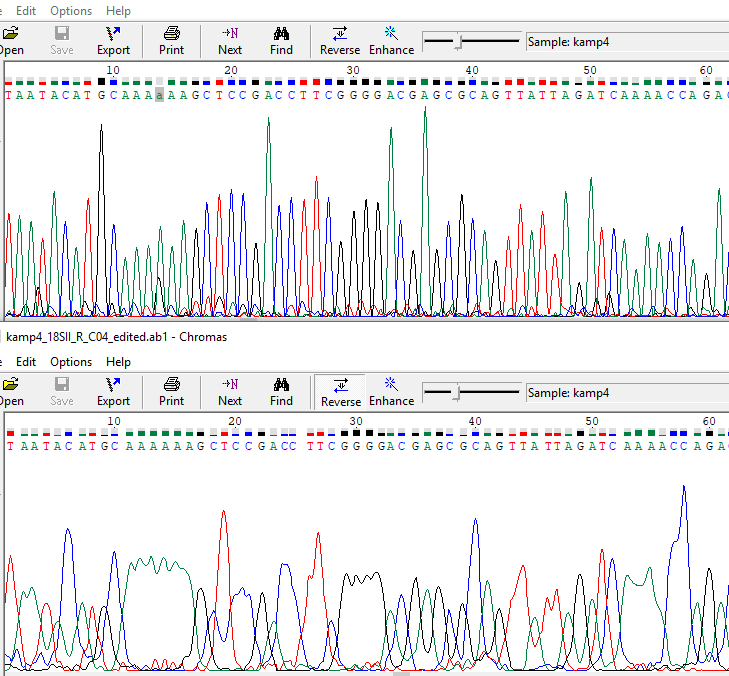
Прочтение последовательностей по Сэнгеру
Итоговая последовательность в формате fasta
Проект JalView c выравниванием прямого и обратного прочтений
Исходный файл с прямым прочтением
Исходный файл с обратным прочтением
Пояснения: изначально были удалены нечитаемые концы: для прямого прочтения нечитаемый 5'-конец составил 50 нуклеотидов, 3'-конец - 196, для обратного прочтения - нечитаемый 5'-конец - 50 нуклеотидов, 3'-конец - 175. Для обратного прочтения был сделан Reverse-Complement. После этого последовательности были сохранены в fasta-формате и выровнены. После выравнивания пришлось удалить ещё 107 нуклеотидов с 5'-конца прямого прочтения и 174 нуклеотида с 3'-конца Reverse-Complement обратному прочтения (так случилось, потому что изначально нечитаемые 3'-концы были длиннее, чем 5'). В остальном, выравнивание получилось почти идеальным, за исключением 5 мест, где в прямом прочтении стоял неопознанный нуклеотид (N). Однако, к счастью, во всех этих позициях в обратном прочтении нуклеотиды были узнаны точно (Qality > 30), поэтому не составляло труда исправить недочёты в прямом прочтении. Иных исправлений не потребовалось, так как обе хроматограммы имели достаточно хорошее качество. Можно отметить, что для прямого прочтения к концу, начиная примерно с 633 нуклеотида (в нередактированной версии) качество несколько падает (пики становятся более широкими, менее выраженными), неопознанные нуклеотиды возникают в позициях 171, 413, 602, 630, 686 (14, 256, 445, 473, 529 после редактирования) из-за слишком высокого уровня шума и, что интересно, чаще всего шум вносит чёрный пик G. В окрестности позиции 602 (445 после редактирования) прямого прочтения наблюдается сильный шум и наложение пиков. Однако это же место в обратном прочтении прочитано качественно. В случае обратного прочтения пики также начинают размываться к концу (тоже примерно с 630-640 в нередактированной версии), но при переворачивании они оказываются в начале. В целом, обратное прочтение менее зашумлено, чем прямое.

Рис.1а. Проблемный 14 нуклеотид
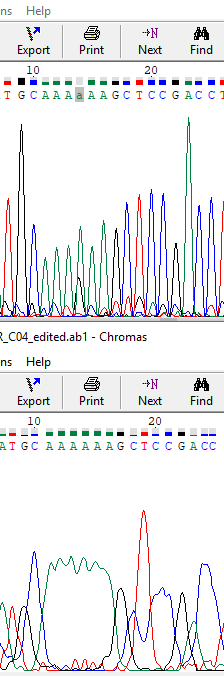
Рис.1б. Проблемный 14 нуклеотид вблизи. Тот случай, когда хроматограмма прямого прочтения более качественная, однако, несмотря на наложение пиков, последовательность в обратномпрочтении угадывается и для 14 нуклеотида можно с высокой степенью уверенности поставить букву А
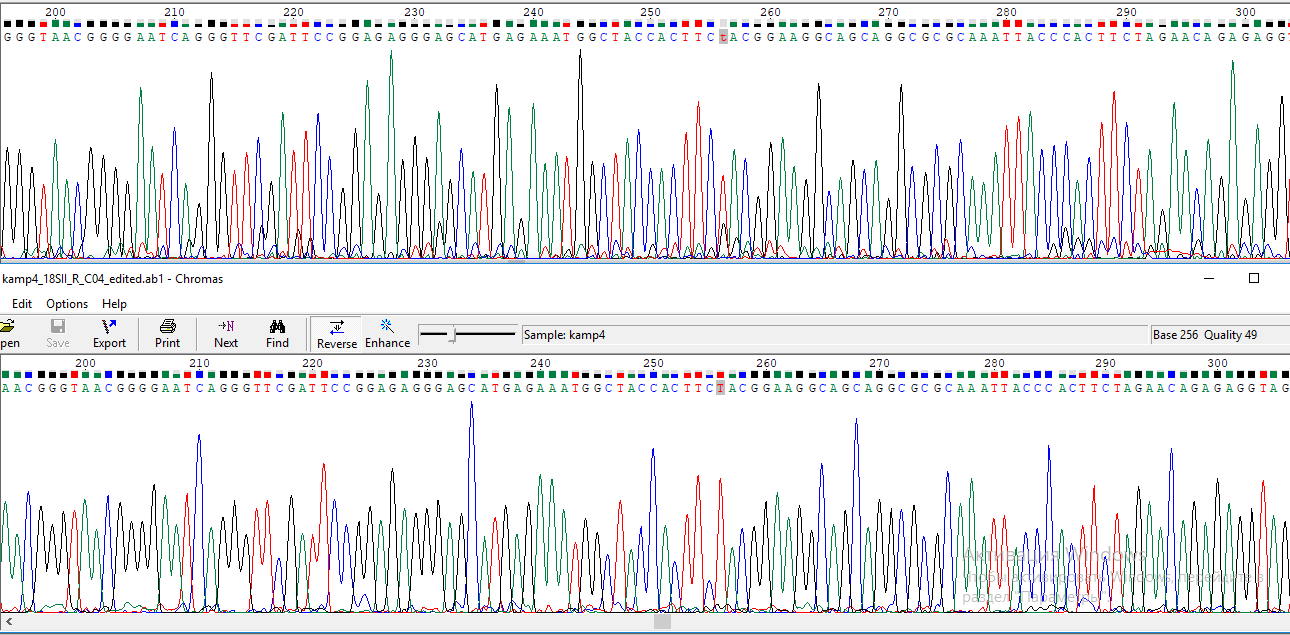 Рис.2. Проблемный 256 нуклеотид
Рис.2. Проблемный 256 нуклеотид
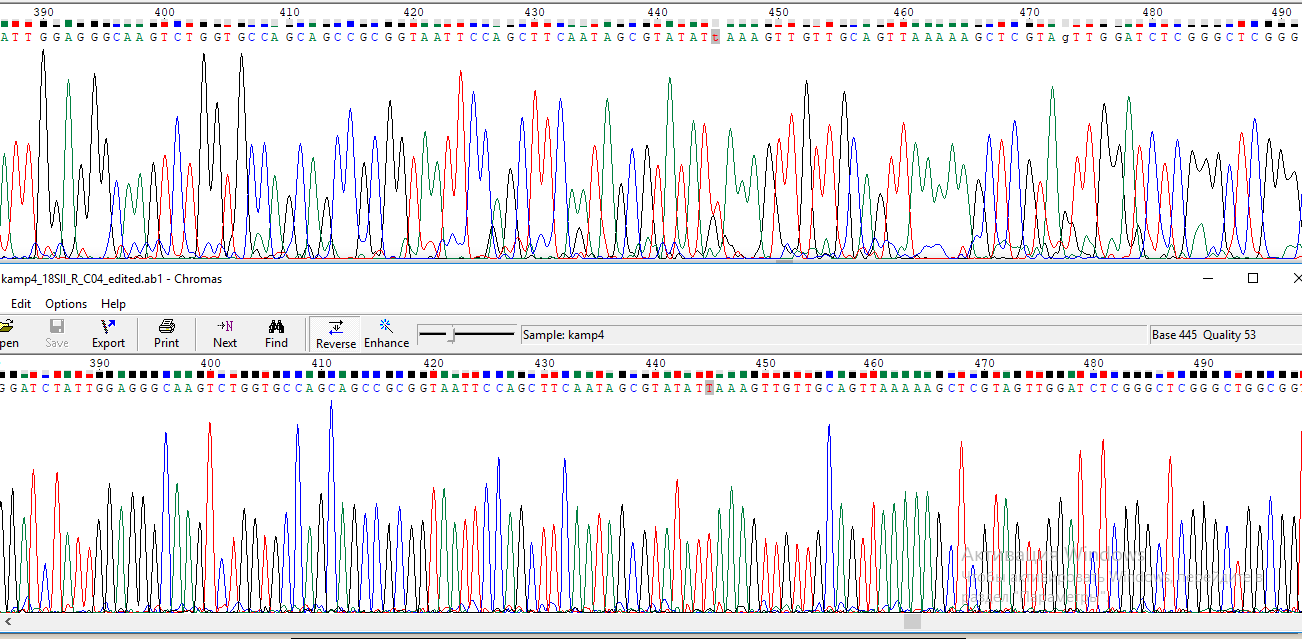 Рис.3а. Проблемный 445 нуклеотид (проблемный участок 442-450). Снова побочный пик G, но хроматограмма обратного прочтения
в этом месте почти идеальная
Рис.3а. Проблемный 445 нуклеотид (проблемный участок 442-450). Снова побочный пик G, но хроматограмма обратного прочтения
в этом месте почти идеальная
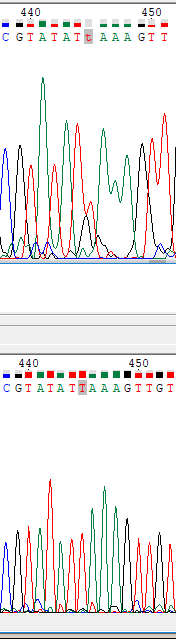
Рис.3б. Проблемный участок вблизи
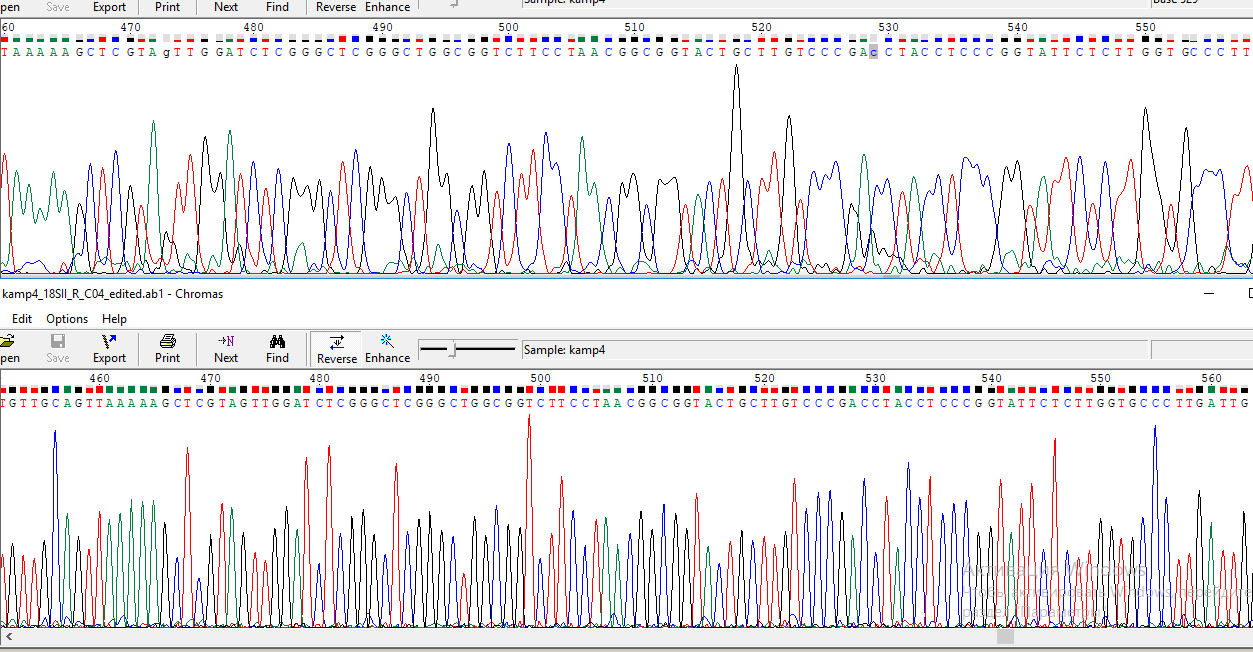
Рис.4. Проблемные 473 и 529 нуклеотиды. Как видно, под конец хроматограмма прямого прочтения сильно портится, но согласуется со второй хроматограммой
Задание 2. Пример плохой хроматограммы
В качестве примера нечитаемой хроматограммы взят файл WSWS2931_H3_R_E10_2013-06-11-22-39-58.ab1
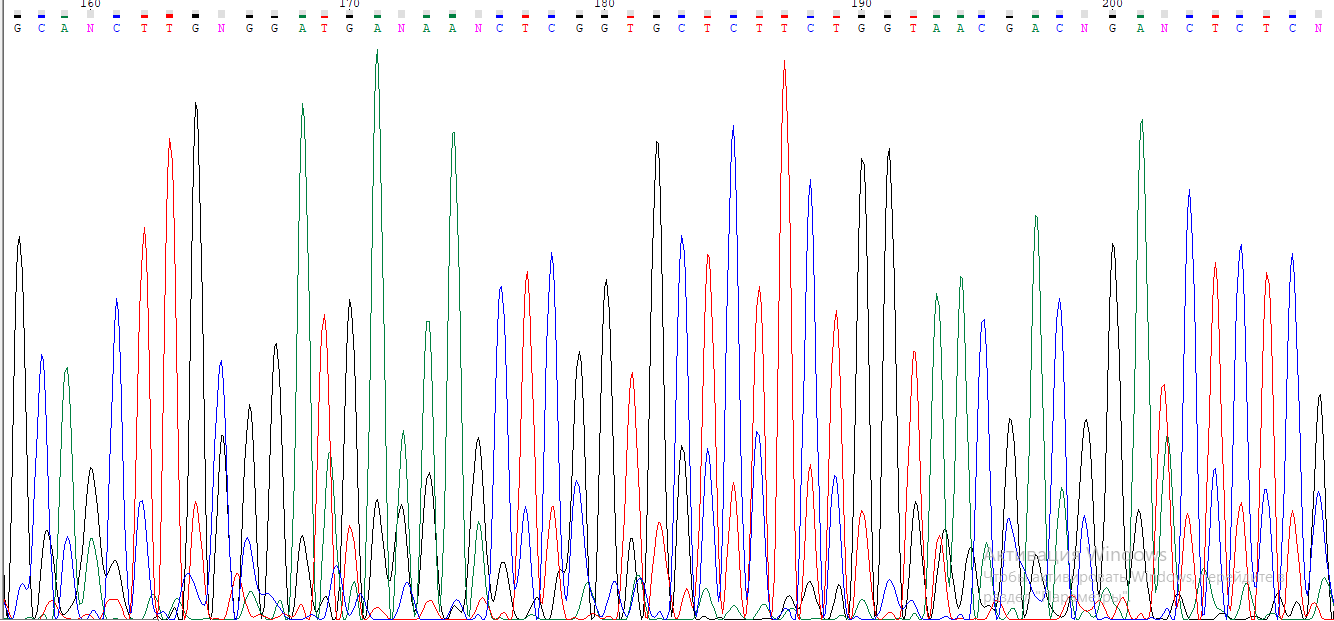
Основная проблема здесь - высокий уровень шума (часто - примерно 1/3 - 1/2 от высоты основного пика). Однако если присмотреться к "шумовым" пикам становится понятно, что они повторяют "нормальные", отставая от них на один нуклеотид. Вероятно, в образце было две последовательности, одна из которых несла вставку/делецию по отношению к другой (место вставки/делеции по данной хроматограмме определить нельзя, так как она испорчена с самого начала).
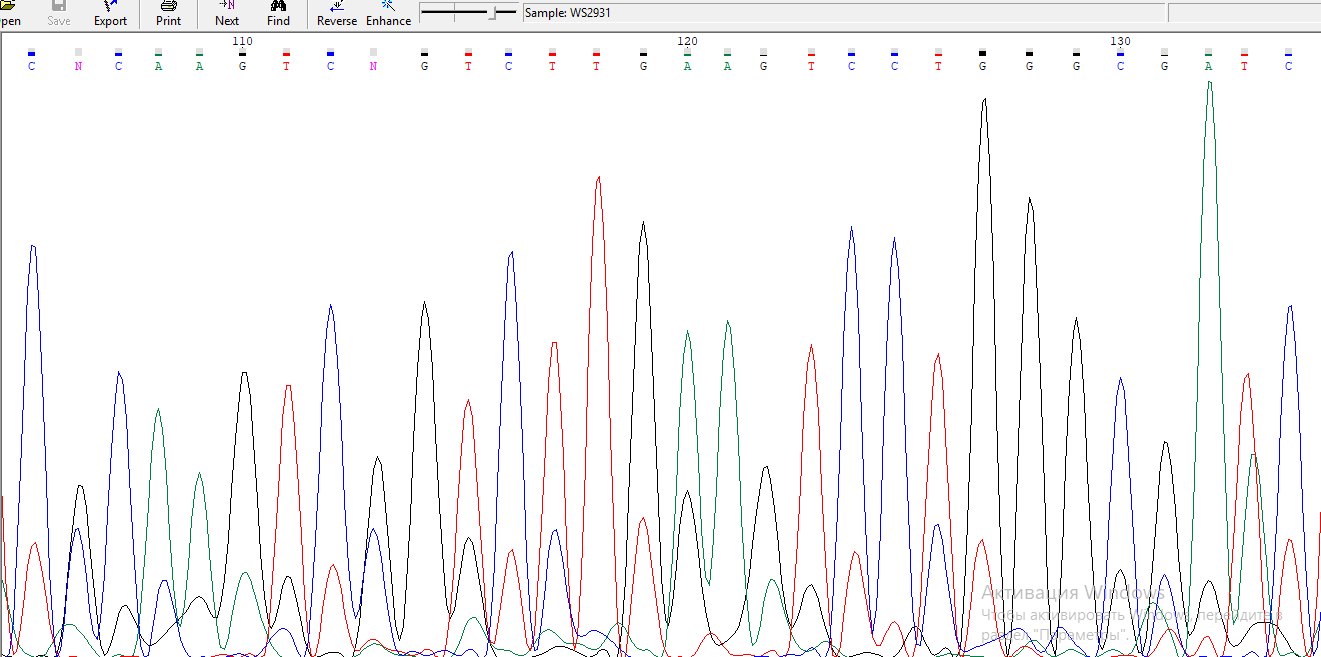 Более наглядная картинка
Более наглядная картинка
© Быкова Даша, 2018