Дан PDB файл с приблизительными координатами какого-то белка. Используя сервисы поиска по структуре, необходимо сделать какие-то предположения о его функции, необходимых кофакторах и участках их связывания и т.д.
Первый хит в выдаче PDBeFold имеет PDB id 1yvd и это ГТФаза Rab-22. При этом идентичность выравнивания по последовательностям составляет 100%, то есть это тот же самый белок, но, по-видимому, немного в другой конформации. Следовательно, рассматриваемый белок относится к семейству Rab, умеет связывать GDP и GTP и обладает ГТФазной активностью. В качестве кофактора выступает ион магния. При этом, как всякие G-белки, белки семейства Rab выполняют сигнальную функцию, активируются при связывании GTP и переходят в неактивную форму при связывании GDP. При этом в активной форме Rab белки могут участвовать в связывании самых разнообразных молекул, на основе чего происходит дальнейшее разделение внутри семейства. Глобально белки Rab обычно локализуются в эндосомах и участвуют в эндоцитозе и внутриклеточном транспорте белков. Таким образом, помимо связывания GTP и GDP, белок, по-видимому, должен иметь сайт связывания с неким эффектором, то есть белком, который будет подвержен регуляции (такой сайт подробно описан в работе Structural basis of family-wide Rab GTPase recognition by rabenosyn-5).
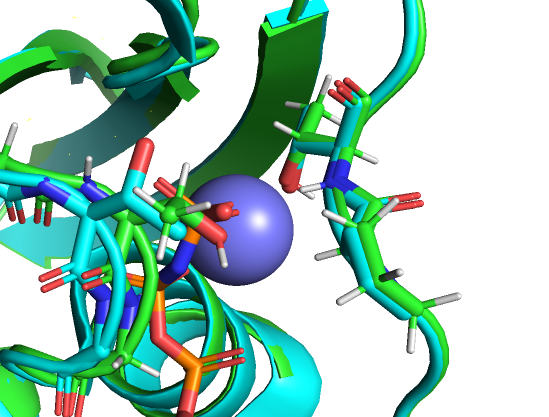
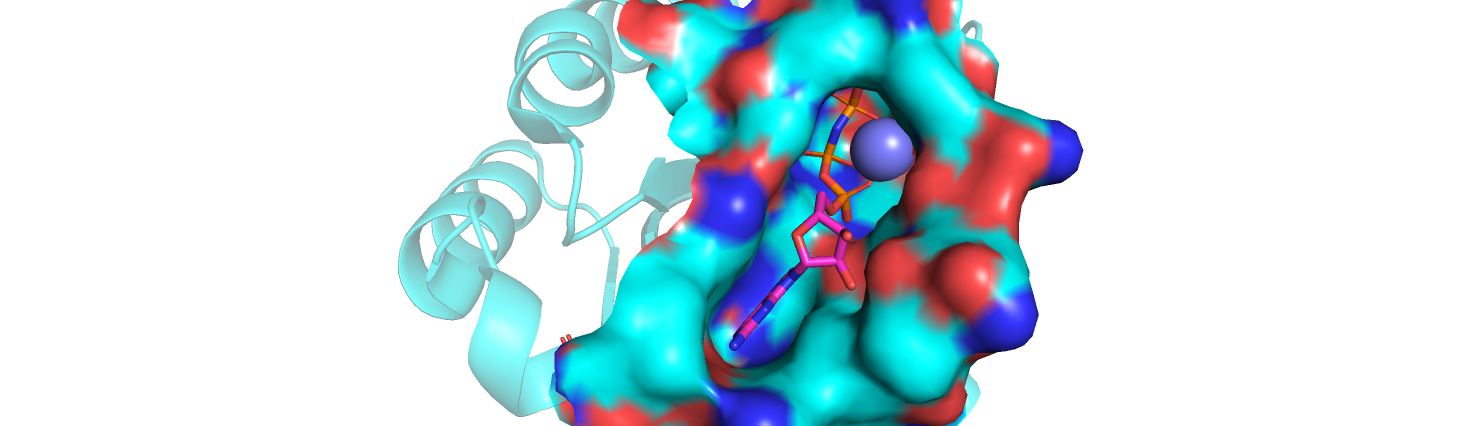
Накладывая структуру 1yvd на исходную, замечаем, что совпадение практически полное за теми исключениями, что петля, образованная остатками 149-151, в 1yvd в представлении cartoon отображается в виде некоторой вторичной структуры (отсюда не полное совпадение вторичных структур в выравнивании PDBeFold), а также несколько изменено положение петли, образованной остатками 13-15. Здесь надо отметить, что структура с pdb id 1yvd содержит не только сам фермент, но и аналог субстрата GNP (GppNHp), а также кофактор Mg, и все различия с исходной структурой приходятся на сайт связывания GNP. Предполагаемый сайт связывания магния и карман, в котором связывается аналог субстрата (и, значит, сам субстрат), показаны на рис.1.


Белок достаточно маленький (166 остатков) и визуально выделить в нём отдельные домены мне не удалось. CATH оправдывает моё неведение и выделяет для структуры 1yvd (и для исходной структуры, конечно, тоже) всего 1 домен - 1yvdA00. Данный домен относится к суперсемейству (Homologous Superfamily) P-loop containing nucleotide triphosphate hydrolases (3.40.50.300), имеет топологию (Topology) Rossmann fold (3.40.50), архитектуру (Architecture) - 3-Layer(aba) Sandwich (3.40), класс - Alpha Beta (3).
P-loop (phosphate-binding loop) - это некоторый общий мотив для белков, связывающих АТФ или ГТФ, для него характерен паттерн G-x(4)-GK-[TS], он действительно есть в нашем белке и, похоже, именно он определяет связывание субстрата и кофактора (рис.2). Отметим, что исходный белок, по-видимому, дан в несвязывающей конформации, так как треонин в нём повёрнут слишком близко к фосфату.
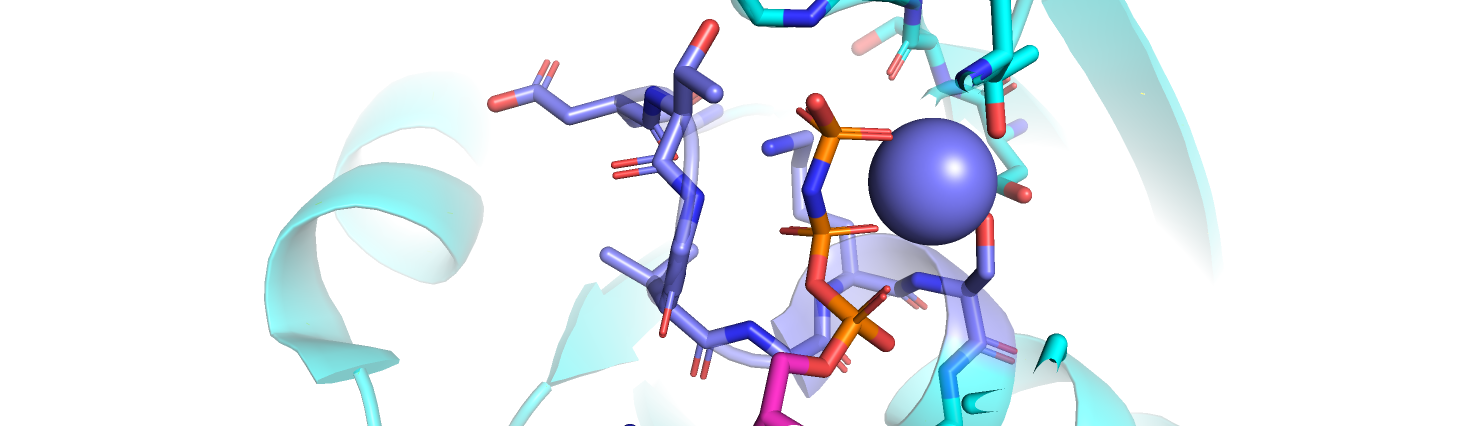
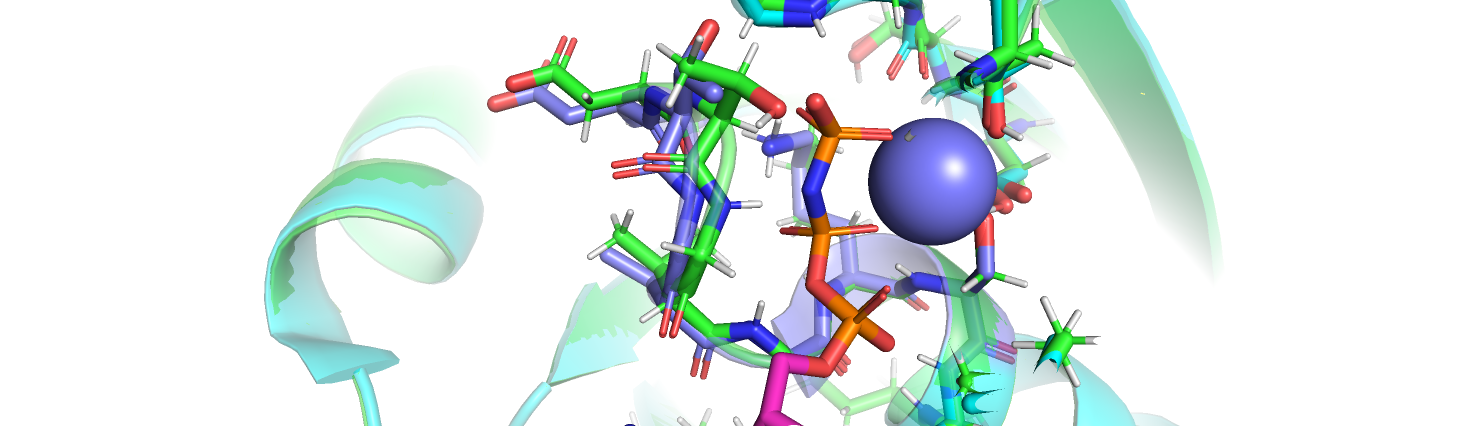
Используем тот же PDB файл. Проведём поиск в PDBeFold, модифицировав параметры по умолчанию (выключила Match connectivity, Best matches only, сортировала результаты по Seq %). Ожидаемо, в топе выдачи, даже при менее 35% идентичности по последовательности, оказались разнообразные представители суперсемейства Ras. Однако, разглядывая хиты, попавшие в самый конец списка, я обнаружила более интересные находки. Например, микобактериальный транскрипционный фактор MtrA (PDB 3nhz:C), регулятор ответа на стресс RcsB из E.coli (PDB 5i4c:A), а также RCP1 из цианобактерии Synechocystis sp. PCC 6803 (PDB 1i3c:B). Все три белка в CATH имеют код 3.40.50.2300, то есть класс, архитектура и топология те же, что и в исходном белке, группа гомологии - Response regulator. Все они активируются путём фосфорилирования, все используют ионы металлов в качестве кофакторов (однако сами металлы могут быть разными - например, MtrA связывает не только ион магния, но и кальция, а RCP1 в качестве кофактора использует марганец), MtrA и RcsB также связывают ДНК (то есть некоторую специфическую нуклеотидную последовательность). Итак, сопоставляя данные молекулы, мы можем рассчитывать увидеть мотив для связывания ионов металлов, мотив для связывания нуклеотидов, а также для связывания фосфата.
© Быкова Даша, 2020