1. Общая информация о белке
| UniProt ID | PFKA_LACDE |
| UniProt AC | P80019 |
| RefSeq ID | WP_003619813.1 |
| PDB ID | 1ZXX |
| Длина (Length) | 319 AA |
| Молекулярная масса (MW) | 34009 Da |
| Рекомендуемое название (RecName) | Full = ATP-dependent 6-phosphofructokinase Short = ATP-PFK Short = Phosphofructokinase |
Комментарии к таблице
Данный белок катализирует следующую реакцию: ATP + D-fructose 6-phosphate = ADP + D-fructose 1,6-bisphosphate (один из этапов гликолиза)
Коферментом фосфофруктокиназы является Mg (II)
Организм, которому принадлежит данный белок - Lactobacillus delbrueckii subsp. bulgaricus (таксономическое положение: Bacteria; Firmicutes; Bacilli; Lactobacillales; Lactobacillaceae; Lactobacillus).
По структуре представляет собой гомотетрамер. На pdb представлена структура одной субъединицы.
2. Описание кластеров UniRef
| Cluster id | Cluster name (название кластера) | Size (количество белков в кластере) |
| UniRef100_P80019 | Cluster: ATP-dependent 6-phosphofructokinase | 6 |
| UniRef90_P80019 | 24 | |
| UniRef50_P80019 | 429 |
Комментарий: Если искать по полному названию, то выдаёт 21764 результатов (парадоксально, что из них 14909 относятся к Ref100, 6136 - к Ref90 и 719 - к Ref50), по названию "phosphofructokinase" - 53541 кластер, "ATP PFK" - 30.
3. Результаты сеансов поиска в UniProt
| Цель запроса | Текст запроса | Количество найденных белков | Из раздела Reviewed | Комментарии | |
| Поиск по полному названию | name:"atp dependent 6 phosphofructokinase" | 15250 | 338 | ||
| Поиск по краткому названию | name:"atp pfk" | 14953 | 338 | ||
| Поиск по другому краткому названию | name:phosphofructokinase | 33980 | 392 | ||
| Поиск по названию среди белков организма | name:"atp dependent 6 phosphofructokinase" organism:"lactobacillus delbrueckii subsp bulgaricus" | 8 | 1 | В Reviewed - изначально рассматриваемый белок, остальные предсказаны по гомологии. По краткому названию (atp pfk) белков находится столько же. По "phosphofructokinase" находится в 2 раза больше. | |
| Поиск по названию среди организмов того же семейства | name:"atp pfk" taxonomy:"Lactobacillaceae [33958]" | 290 | 10 | ||
| Поиск по названию среди организмов того же отдела | name:"atp pfk" taxonomy:"Firmicutes [1239]" | 4581 | 129 | Можно и просто name:"atp pfk" taxonomy:firmicutes | |
| Поиск гомеобоксных белков | name:homeobox | 44012 | 1396 | ||
| Поиск гомеобоксных белков у Ciliophora | name:homeobox taxonomy:ciliophora | 2 | 0 | Статус "Predicted". Оба ДНК-связывающие, один транскрипционный регулятор. | |
| Поиск гомеобоксных белков у Vertebrata | name:homeobox taxonomy:vertebrata | 26135 | 994 | Интересно добавить колонки "function" и "developmental stage" | |
| Поиск по слову "трипсин" | name:trypsin | 18951 | 311 | ||
| Поиск трипсина без его ингибиторов | name:trypsin NOT name:inhibitor | 15037 | 101 | Альтернативные запросы такие: name:trypsin NOT inhibitor, name:trypsin NOT name:"trypsin inhibitor", name:trypsin NOT "trypsin inhibitor", выдают соответственно 14973, 15197 и 15159 записей. Первый из них отсеивает честные трипсины (равносилен поиску name:trypsin NOT (name:trypsin inhibitor) по количеству результатов), хотя для надёжности, вероятно, лучше использовать его, а остальные выдают лишние ингибиторы. |
4. Отличия записи RefSeq от записи UniProt
Дата последнего изменения в UniProt - 25-OCT-2017, RefSeq Protein - на полгода раньше, - 09-APR-2017. В UniProt, на мой взгляд, удобнее представлена информация о функционально значимых участках белка, а также есть информация о публикациях и о структуре, более подрбно описана функция, представлены кинетические параметры. Впрочем, в RefSeq имеются ссылки на другие ресурсы (например, в поле "Region"), где можно получить более подробную информацию (например, о семьях, которым принадлежит данный белок или о сайтах связывания). Итак, запись UniProt хороша тем, что из неё можно извлечь больше информации и полезна, если в центре нашего внимания сам белок. В RefSeq можно сразу увидеть место расположения гена данного белка в геноме организма (Graphics), а также перейти к самому организму - это удобно, если нас интересует не столько структура и функция белка, а, например, функциональная связь белка с белками, закодированными по соседству.
5. История изменения записи UniProt
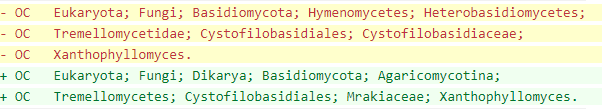
Рассмотрим запись Q7Z859 (LCPS_PHARH). Она была создана 01.10.2003 и с тех пор пережила 63 акта редактирования (последняя версия несёт номер 64, последние изменения были внесены 28.02.2018). При переходе от версии 26 к 27 было изменено название записи с Q7Z859_PHARH на LCPS_PHARH, запись переехала из TrEMBL в Swiss-Prot, произошло объединение с двумя другими: Q7Z856 и Q9UUQ0. Также в некоторый момент было изменено таксономическое положение организма, были добавлены публикации и комментарии с описанием функции и другая информация (например, наличие трансмембранных доменов, кодирующий ген, наличие пролин-богатого региона), менялся статус последовательности (на данный момент - "Experimental evidence at transcript level"). Примеры изменений приведены ниже.


6. Нестандартные аминокислотные остатки
В записи UniProt нестандартные аминокислотные остатки (селеноцистеин или пирролизин) представлены в Feature Table как NON_STD. Примеры: P24183 ("FT NON_STD 196 196 Selenocysteine."), Q8TTA5 ("FT NON_STD 356 356 Pyrrolysine. {ECO:0000250}."). Селеноцистеин имеет трёхбуквенный код Sec (однобуквенный - U), пирролизин - Pyl (O). Если селеноцистеин входит в состав активного центра фермента, то его номер указывается как в графе "Non-standard residue", так и в "Active site": P07203.
© Быкова Даша, 2018